BAL 31 NucleaseまたはExonuclease IIIによる直鎖DNAの限定分解
pBR322ベクターを制限酵素Ava Iで1ケ所切断した直鎖DNAをBAL 31 Nuclease単独、またはExonuclease IIIとS1 Nucleaseの組合せによって、限定分解した例を以下に紹介する。
操作手順(BAL 31 Nuclease)
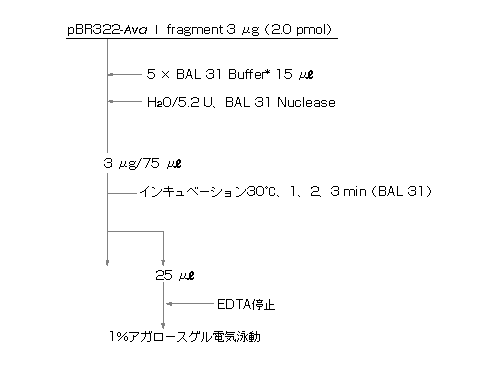
* 5 × BAL 31 Buffer
100 mM Tris-HCl(pH8.0)、60 mM CaCl2、60 mM MgCl2、5 mM EDTA、3 M NaCl
操作手順(Exonuclease III-S1 Nuclease)

* 10 × Exo III Buffer
660 mM Tris-HCl(pH8.0)、770 mM NaCl、50 mM MgCl2、100 mM DTT
** 5 × S1 Buffer
250 mM 酢酸ナトリウム緩衝液(pH5.0)、1 M NaCl、5 mM 酢酸亜鉛
結果
BAL 31 Nucleaseは220~1000塩基前後と大きく切除する目的に適しており、Exonuclease III は数10~数100の少ない塩基の切除に適している。BAL 31 Nucleaseの場合、塩基依存性が強いといわれているため、反応の進みにくいDNAの場合は、塩基の影響の少ないExonuclease IIIを用いて時間をかけて分解するのがよい。生成したDNA fragmentが平滑末端である確率はいずれの場合でも10%前後なので、Klenow Fragment(またはT4 DNA Polymerase)により末端修復を行う方が、ライゲーション効率を高めることができる。
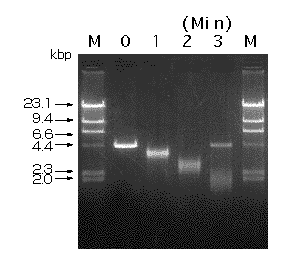
図1 BAL 31 NucleaseによるpBR322 DNA Ava I フラグメントの分解
DNAは前記条件で、BAL 31により約2,400 base/3 min(約400 base/min/end)の割合で分解された。
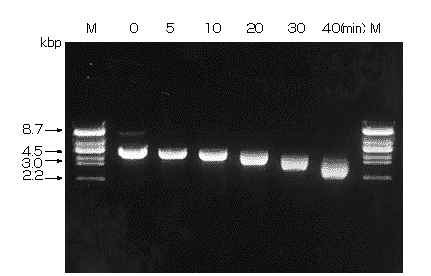
図2 Exo III によるpBR322 DNA Ava I フラグメントの分解
DNAは前記条件で、約1,400 base/40 min(約17.5 base/min/end)の割合で分解された。
