プラスミドpAUR101を用いたADE1遺伝子のgene disruption
原理
遺伝子破壊の原理をADE1遺伝子をモデルとして、図1.に示す。ADE1遺伝子の一部を含むpDADE1で形質転換を行うと、プラスミド上のADE1断片と染色体上のADE1遺伝子の相同領域が相同組換えを起こし、染色体の遺伝子内へpDADE1ベクターが挿入された状態となる。その結果、染色体上のADE1遺伝子は機能しなくなる(左側の遺伝子はC末端を欠き、右側の遺伝子はN末端を欠く)。すなわち、ADE1遺伝子を特異的に破壊したことになり、この現象を遺伝子破壊(gene disruption)という。二倍体の酵母を宿主とした場合、pDADE1を用いた形質転換により、一方のADE1遺伝子のみが破壊される場合と2本が同時に破壊される場合とが考えられる。両染色体上のADE1遺伝子が破壊された場合、アデニン要求性(Ade-)となり、アデニンを添加していないYPD プレート上ではピンク色のコロニーを形成し、容易にその検出が可能となる。しかし、2本の染色体上の対立遺伝子が同時に破壊される確率は非常に低いと考えられる。
操作手順
清酒酵母協会701号からゲノムDNAを取得し、これを鋳型DNAとしたPCRにより、ADE1 gene disruptionに必要なADE1遺伝子の一部領域(約500 bp)を得た。このDNA断片をpAUR101のSma I サイトに挿入し、gene disruption用プラスミド(pDADE1)を作製した。pDADE1 約5 μgをADE1断片上のNde I 部位で1ヵ所切断して直鎖とし、プロトコールにしたがって、清酒酵母の形質転換を行った。形質転換処理後、アデニンを添加したYPD液体培地で一晩培養した菌体を、0.5 μg/ml AbAを含むYPD選択プレートに適宜、塗布し、30℃で3日間培養した。
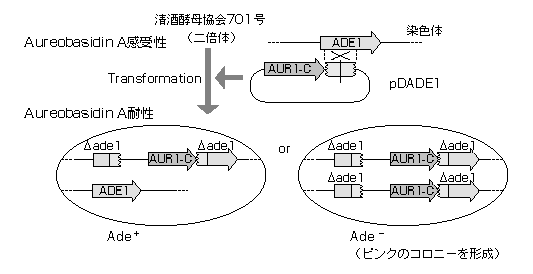
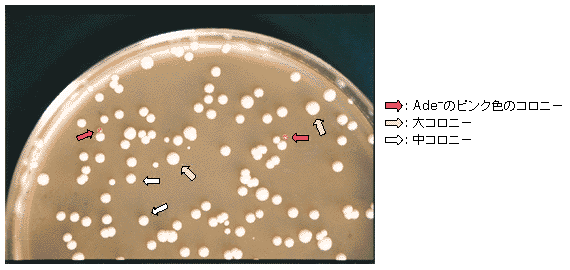

図1. Gene disruptionの原理

図2. ADE1-disrupted clonesの選択プレート上での生育
