Viable Bacteria Selection Kit for PCR (Gram Positive)
黄色ブドウ球菌の生菌選択的DNA検出<条件検討およびモデル実験>
EMAは死菌由来DNAを効率よく修飾するが、菌種や菌数、処理条件によっては生菌にも若干の影響を与えることが知られている。純培養した生菌を段階希釈して、本製品によるEMA処理あり/なしの結果を比較することにより、生菌への影響を確認する。
培養した
Staphylococcus aureusを生理食塩水に懸濁して生菌サンプルとした。また、その懸濁液の一部を95℃、5分間熱処理したものを、死菌サンプルとした。
本製品の手順に従って以下の条件にてEMA処理を行い、得られたDNAを鋳型として、CycleavePCR
Staphylococcus aureus (DnaJ gene) Detection Kit(製品コード CY228)とThermal Cycler Dice Real Time System
II(終売)を用いたリアルタイムPCRを行った。
| Solution B-gp | 希釈せず |
| EMA処理時間 | 5分 |
| EMA処理回数 | 2回 |
1.EMA処理の生菌への影響
| 方法 : | 生菌サンプルを段階希釈して、2×107~2×103個の生菌サンプルをEMA処理に供し、EMA処理あり/なしサンプルの増幅曲線を比較することにより、生菌への影響を確認した。 |
EMA処理なし
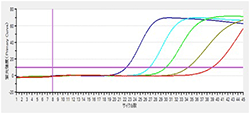
|
EMA処理あり
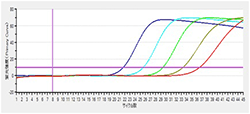
|
結果: EMA処理あり/なしでほぼ同等の結果が得られており、検出感度には影響がないことが分かります。
2.死菌抑制効果
対象のグラム陽性菌について、EMA処理により、どの程度までの死菌量に対して死菌由来DNAからのPCR増幅を抑制できるかをあらかじめ確認しておくことが重要である。純培養した生菌から熱処理等により死菌を調製し、段階希釈して、本製品によるEMA処理あり/なしの結果を比較することにより、死菌抑制効果を確認する。
エンドポイントPCRの場合は、目的サイズのバンドの有無で完全に抑制可能な最大死菌量を確認し、その範囲内で実検体の測定を行う。(その範囲を超える死菌が存在する場合には、EMA処理により完全に抑制できない可能性がある。)死菌抑制効果が不十分な場合は、EMA処理回数を増やす、あるいはPCR増幅サイズを大きくするといった条件検討を行う。
リアルタイムPCRの場合には、完全に抑制可能な最大死菌量のEMA処理なしでのCt値(Xとする)を参考にすることで、検体サンプル中の死菌由来DNAが完全に処理されたかどうかを推測することも可能である。EMA処理なしサンプルのCt値(Y)がY>Xであれば、死菌由来DNAはほぼ修飾されたと推測できる。Y≦Xの場合には、検体中の死菌量が多く、EMA処理により完全には修飾できなかった可能性を考慮する必要がある。
| 方法 : | 死菌サンプルを段階希釈して、2×107~2×103個の死菌サンプルをEMA処理に供し、EMA処理あり/なしサンプルの増幅曲線を比較することにより、死菌抑制効果を確認した。 |
EMA処理なし
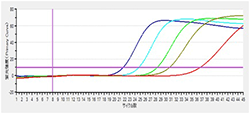
|
EMA処理あり
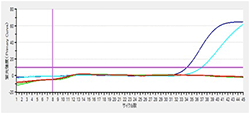
|
| 結果 : | EMA処理あり/なしのCt値の差は14程度であることから4 log程度の死菌抑制効果があり、2×104~2×105個までの死菌を抑制できると推測されます。
|
Viable Bacteria Selection Kit for PCR (Gram Positive)




