環状RNAのNGS解析におけるRNase R活用例
【背景】
ヒトをはじめとした真核生物において、環状RNAはmRNA前駆体からバックスプライシングを通じて形成され、バックスプライスジャンクション(BSJ)と呼ばれる直鎖状RNAには存在しないスプライスジャンクションが存在します。そのため、シーケンス結果に基づいてBSJを含むフラグメントを集計することで、同じ遺伝子領域からコードされる直鎖状RNAと環状RNAを識別したり、環状RNAを定量することができます。
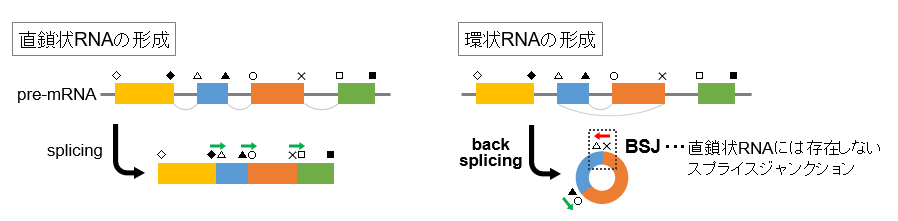
しかし、環状RNAは存在量が少なく直鎖状PolyA-RNAの1%程度であるという報告1)もあり、環状RNAの詳細解析においては、サンプル中の環状RNAを選択的に解析する手法が必要とされています。
本実験では、環状RNAを標的としたNGS解析を行うにあたり、RNase Rによりサンプル中の直鎖状RNAを特異的に分解し、環状RNAの濃縮を行う手法の有効性を評価しました。

しかし、環状RNAは存在量が少なく直鎖状PolyA-RNAの1%程度であるという報告1)もあり、環状RNAの詳細解析においては、サンプル中の環状RNAを選択的に解析する手法が必要とされています。
本実験では、環状RNAを標的としたNGS解析を行うにあたり、RNase Rによりサンプル中の直鎖状RNAを特異的に分解し、環状RNAの濃縮を行う手法の有効性を評価しました。
【実験内容・結果】
HEK293T細胞由来のTotal RNA 100 μgに対し、spike-inとして既知配列の人工環状RNAを10 ng添加しました。その後、RNase R処理なしおよびRNase R処理あり(40 U、37℃、30分間)についてそれぞれRNAを精製し、各1 ngを用いてRNA-Seqを行いました。
Spike-inとして添加した人工環状RNA領域のTPM値を算出しました(表1)。
その結果、RNase R処理ありのサンプルにおける人工環状RNA領域のTPM値は、処理なしの場合と比較して約4.8倍高くなりました。
これにより、RNase Rを用いて環状RNAの濃縮を行ったサンプルを解析に使用することで、存在量の少ない環状RNAをより選択的に検出できることが示されました。
表1. RNase R処理の有無による人工環状RNA領域発現量(TPM*値)の比較
次に、ヒトゲノムにおいて環状RNAの発現が報告されている6つの遺伝子について、BSJを含むフラグメント数をRNase R処理の有無で比較しました(表2)。
その結果、RNase R処理ありの場合、処理なしのサンプルよりBSJを含むフラグメントが多く検出されました。また、RNase R処理なしではBSJを含むフラグメントが検出されなかった遺伝子に関しても、RNase R処理ありでは検出が可能となりました。
これにより、RNase Rを用いた環状RNAの濃縮は、定量性の向上および検出感度の引き上げに有用であることが示されました。

1)Salzman, Julia et al.(2013)PLoS genetics vol. 9,9: e1003777.
Spike-inとして添加した人工環状RNA領域のTPM値を算出しました(表1)。
その結果、RNase R処理ありのサンプルにおける人工環状RNA領域のTPM値は、処理なしの場合と比較して約4.8倍高くなりました。
これにより、RNase Rを用いて環状RNAの濃縮を行ったサンプルを解析に使用することで、存在量の少ない環状RNAをより選択的に検出できることが示されました。
表1. RNase R処理の有無による人工環状RNA領域発現量(TPM*値)の比較
| RNase R処理 | TPM*1値 |
| なし | 5,964 |
| あり | 28,890 |
*1 Transcripts per million
ここでは、100万フラグメントあたりの人工環状RNA由来のフラグメントの数を示す。
ここでは、100万フラグメントあたりの人工環状RNA由来のフラグメントの数を示す。
次に、ヒトゲノムにおいて環状RNAの発現が報告されている6つの遺伝子について、BSJを含むフラグメント数をRNase R処理の有無で比較しました(表2)。
その結果、RNase R処理ありの場合、処理なしのサンプルよりBSJを含むフラグメントが多く検出されました。また、RNase R処理なしではBSJを含むフラグメントが検出されなかった遺伝子に関しても、RNase R処理ありでは検出が可能となりました。
これにより、RNase Rを用いた環状RNAの濃縮は、定量性の向上および検出感度の引き上げに有用であることが示されました。
表2. RNase R処理の有無によるBSJを含むフラグメント数の比較
| ヒト由来環状RNA | BSJ/40Mフラグメント | BSJ/160Mフラグメント | |||
| RNase R処理なし | RNase R処理あり | RNase R処理なし | RNase R処理あり | ||
| HIPK3 | 15 | 131 | 45 | 480 | |
| LINC00632 | 248 | 679 | 965 | 2,695 | |
| MTO1 | 0 | 10 | 0 | 48 | |
| PLOD2 | 0 | 28 | 19 | 124 | |
| SMARCA5 | 34 | 100 | 125 | 407 | |
| XPO1 | 24 | 63 | 105 | 294 | |
シーケンスに使用した試薬等
*2 フラグメントの両末端から150塩基ずつシーケンス解析を行う。| 解析 | 工程 | 使用した試薬、ソフトウェア等 |
| RNA-Seq | ライブラリー作製 | SMART-Seq Stranded Kit (製品コード 634442) |
| シーケンス解析 | NextSeq2000 (Illumina)、150PE*2 | |
| 情報解析 | マッピング解析 | BWA-MEM |
| 参照配列 | ヒトゲノム(GRCh38) | |
| 発現量算出、BSJを跨ぐリードの検出 | CIRI2 |
1)Salzman, Julia et al.(2013)PLoS genetics vol. 9,9: e1003777.
