Guide-it™ SNP Screening Kit Q&A
Guide-it SNP Screening Kit原理紹介
- Q1 キットのワークフローは?
-
A1.Guide-it SNP Screening Kitの操作ステップを示します。96サンプルを4時間以内に分析可能です。
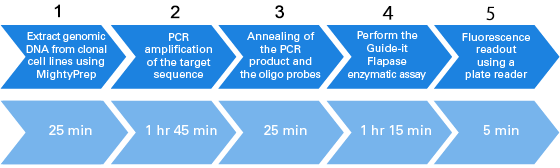
1、クローニングした細胞サンプルからMightyPrepを用いてゲノムDNAを抽出
2、ターゲット配列をPCR増幅
3、PCR産物とオリゴプローブをアニーリング
4、Guide-it Flapaseによる酵素反応
5、プレートリーダーで蛍光値を測定 - Q2 準備が必要なオリゴDNAの種類は?
-
A2.Guide-it SNP Screening KitでSNPを検出するには下記のオリゴDNAが必要です。
・ゲノムDNAからターゲット配列を増幅するためのPCRプライマー(オレンジ)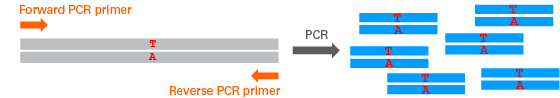
・PCR産物上の置換塩基を検出するためのDisplacement oligo(緑)とflap-probe oligo(紫)
・Displacement oligoとflap-probe oligoが機能することを確認するための SNP control oligoとwild-type(WT) control oligo(青)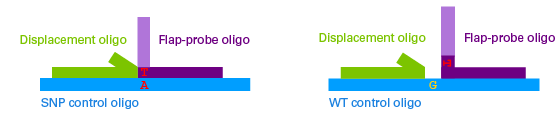
Guide-it SNP Screening Kitで使用するオリゴDNAには蛍光やクエンチャーなどの修飾は必要ありません。また、特別な精製は不要で、標準的な脱塩で十分です。
ただし、Flap-probe oligoの3’末端にはヘキサンジオール(hexanediol, /3C6/) によるブロック修飾が必要です。 - Q3 Guide-it Flapaseはどの様にSNPを認識するのか?
-
A3.Guide-it Flapaseはリコンビナントの構造特異的ヌクレアーゼで、隣接した二重フラップ(double-flap)構造を認識して切断します。フラップ間にギャップが存在する場合には切断しません(下図)。Guide-it SNP Screening Kitでは、この性質を利用してSNPの認識を行います。二重フラップ構造とギャップ構造の大きな違いは、Flap-probe oligoとPCR産物との間でSNP検出のターゲットとなる塩基(Interrogated base)のハイブリダイゼーションが起こるかどうか、という点にあります。

Displacement oligo、Flap-probe oligo、PCR産物のハイブリダイゼーションによる三重構造 図は、C>T置換を検出するために設計されたアッセイ系の構造を示している。
Displacement oligo(緑)とFlap-probe oligo(紫、置換された塩基Tを含む)がPCR産物(青)にアニーリングすると、PCR産物の配列が塩基置換(edited)されたかWild Typeかによって二種類の異なる構造をとる。
二重フラップ構造(左図):塩基置換が起こった場合、Flap-probe oligoはターゲット部位(Interrogated base)と対合して完全な相補鎖となる
ギャップ構造(右図):塩基置換が起こらずWild typeの配列になっている場合は、Flap-probe oligoはターゲット部位と対合せず、ギャップが生じる。 - Q4 Guide-it SNP Screening Kitにはどの様な試薬が含まれているか?
-
A4.Guide-it SNP Screening Kitには哺乳類細胞サンプルからのゲノムDNA抽出、PCR増幅、およびGuide-it Flapaseアッセイに必要な全ての試薬が含まれています。 本キットを使用するには、目的配列を特異的に検出するためのオリゴDNA、サーマルサイクラー、および蛍光プレートリーダーが必要です。
- Q5 どの様にアッセイシグナルを検出するのか?
-
A5.アッセイシグナルの検出には、標準的な蛍光プレートリーダー(excitation 485 nm、capture 535 nm)が使用できます。お持ちのプレートリーダーのフィルターセットの波長が異なる場合は、キットに含まれるGuide-it SNPコントロールセットを用いて、最適なフィルターの組み合わせを調べることができます。
- Q6 Guide-it SNP Screening KitはゲノムDNA上のどの位置でも標的部位の塩基置換を検出することができるか?
-
A6.はい。Guide-it SNP Screening Kitは、ヒトゲノム上の18の異なる遺伝子座(14の異なる遺伝子)に対してSNPの解析に成功しています。
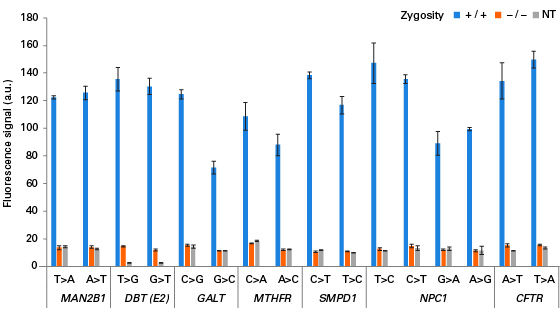
各遺伝子座における塩基置換の検出 コーリエル医学研究所から得られたゲノムDNAを解析した。各ゲノムDNAは各遺伝子座で野生型かホモ接合型である。野生型(赤色)やネガティブコントロール(灰色)で得られた蛍光シグナルと比較して、各塩基置換がホモ接合型のサンプル(青色)では、全ての置換において、明らかに強いシグナルを示した。
- Q7 ターゲット部位がどの塩基に置換されたか不明な場合もGuide-it SNP Screening Kitを使用できるか?
-
A7.Guide-it SNP Screening Kitはゲノム上の特定部位の一塩基置換を検出するように設計されており、Flap-probe oligoの配列がどの塩基への置換を検出するかを決定します。どの塩基に置換されたかを同定したい場合は、G、C、A、Tを検出するFlap-probe oligoをそれぞれ準備して別々にアッセイを行います。そうすれば、いずれかのFlap-probe oligoでシグナルが得られます。
- Q8 Guide-it SNP Screening Kitはジェノタイピングに使用可能か?
A8.可能です。その場合、考えられる塩基に対してそれぞれFlap-probe oligoを設計し、別々にアッセイを行う必要があります。
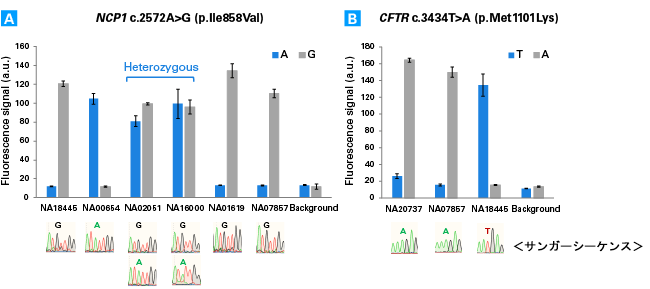
遺伝子型判定での使用 コーリエル医学研究所から得られた、NCP1 またはCFTR 遺伝子にSNPを有する細胞をGuide-it SNP Screening KitとサンガーシーケンスによりSNP解析した。パネルAでは、2種類のflap-probe oligoを作製し、A>G置換でのAとGを別々にアッセイし、ホモかヘテロかを決定した。パネルBでは、T>A置換において、野生型かホモかを解析した。全ての結果は、サンガーシーケンスの結果と一致した。
- Q9 Guide-it SNP Screening Kitはゲノム編集で得られた細胞集団の相同組換え(homologous recombination:HR)の評価に使用可能か?
-
A9.Guide-it SNP Screening Kitはクローニングした細胞株のSNP検出を目的としていますが、検出限界を5%*としてゲノム編集後の細胞集団の相同組換の評価にも使用可能です。
* NucleoSpin Tissue(製品コード 740952.10/.50/.250)などで精製したゲノムDNAを用いることをお勧めします。
詳細はこちらをご参照ください - Q10 Guide-it SNP Screening Kitのアッセイには、カラム精製したゲノムDNAの使用が必要か?
-
A10.いいえ。キットに含まれるMightyPrep reagent for DNAで調製した細胞ライセートをそのままPCRの鋳型として使用できます。特別な精製ステップは不要です。
- Q11 Guide-it SNP Screening Kitで使用実績がある細胞は? 浮遊細胞は使用可能か?
-
A11.Guide-it SNP Screening Kitは、浮遊細胞(Jurkat)、接着細胞(線維芽細胞、hiPS細胞)など、様々なタイプの細胞で成功例があります。
- Q12 Guide-it SNP Screening Kitのアッセイに適した細胞密度は?
-
A12.クローン化された細胞の96ウェルプレートでの増殖効率は細胞によって異なっており、本キットは2×104~2×105 cells/wellの広範囲の細胞密度で使用可能です。ただし、相同組換えの検出を行う場合はNucleoSpin Tissue(製品コード 740952.10/.50/.250)などで精製したゲノムDNAを用いることをお勧めします。
- Q13 Guide-it SNP Screening Kit を用いてhomozygote(ホモ接合体)とheterozygote(ヘテロ接合体)を区別することは可能か?
A13.Guide-it SNP Screening Kitで得られる結果は、置換頻度を定量的に求めるというより定性的な傾向にあります。したがって、単発のアッセイでhomozygoteとheterozygoteの区別を行うことはできません。接合状態の評価を行いたい場合はQ8ジェノタイピングの場合の方法を参考にしてください。
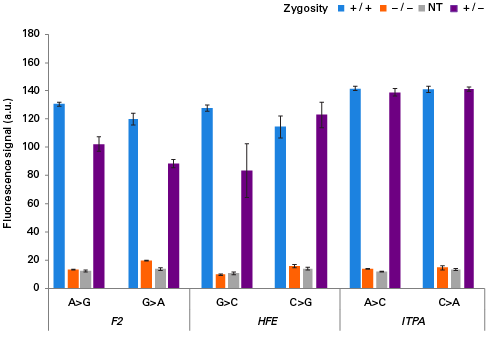
SNPスクリーニング結果の比較 ホモ、ヘテロ、野生型の塩基置換を示す細胞を使用してSNPスクリーニングを行った。各ケースにおいて、ホモとヘテロサンプルで、十分に高い蛍光シグナルが得られた。
- Q14 ゲノム上のターゲット配列のPCRはシングルバンド増幅が必要か?
-
A14.Guide-it SNP Screening Kitのアッセイは、PCR産物中にエキストラバンドが含まれていても影響を受けません。下記の電気泳動写真で示す増幅産物を用いてSNPの検出に成功しています。
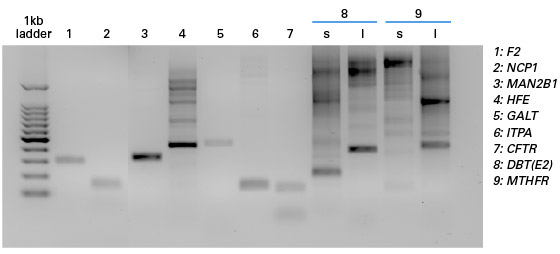
Guide-it SNP Screening Kitに使用されたPCR増幅サンプル例 それぞれのレーンに示されたPCR増幅サンプルは、Guide-it SNP Screening Kitを用いて左に示す遺伝子座の塩基置換の検出に成功したものである。
電気泳動結果に示されている様に、増幅サンプルのサイズは200~700 bpの範囲に分布している。
DBT(E2)、MTHFR については、サイズが異なる2種類のPCR産物でSNPの検出に成功している。 - Q15 ターゲットのSNPの近傍に変異がある場合、どの程度の影響があるか?
-
A15.Guide-it SNP Screening Kitは、ターゲットとなる置換配列の近傍に変異がある場合には敏感に影響を受けます。本キットでSNPを検出するためには、Displacement oligoとFlap-probe oligoが置換配列の部分に正しくアニーリングする必要があります。SNP近傍に未知の変異が存在すると、オリゴとPCR産物のアニーリングが阻害され二重フラップ構造が形成できなくなります。
- Q16 Guide-it SNP Screening Kitで検出するターゲット配列について配列の制限や特別に考慮すべき点はあるか?
A16.プローブを設計する際に考慮すべき唯一の制限は、Displacement oligoの3’末端の配列が5’-GGAGn-3’、または5'-GGAGNn-3’にならないことです(Nは任意の塩基、nはPCR産物上の置換部位と対合しない塩基)。Displacement oligoの3’末端がこれらの配列の場合には、Guide-it Flapase切断の際にバックグラウンドシグナルが生じます。その場合は、PCR産物の逆鎖側にプローブを設計してください。逆鎖側にDisplacement oligoを設計することにより、制限配列の出現を避けることができます(パネルB)。
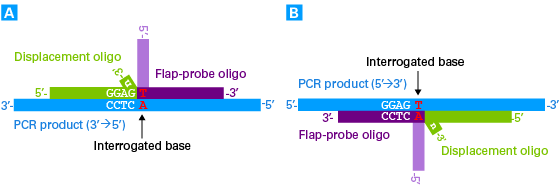
Displacement oligoの設計における制限配列の例 上記の例では、PCR産物のantisense鎖に対してDisplacement oligoを設計すると、Displacement oligoの3’末端が制限配列となる(パネルA)。その場合の対策として、逆鎖(PCR産物のSense鎖)に対してDisplacement oligoを設計すると、制限配列を回避することができる(パネルB)。
