大腸菌の生菌選択的DNA検出<条件検討およびモデル実験>
1. EMA処理の生菌への影響
EMAは死菌由来DNAを効率よく修飾するが、菌種や菌数、処理条件によっては生菌にも若干の影響を与えることが知られている。純培養した生菌を段階希釈して、本キットによるEMA処理あり/なしの結果を比較することにより、生菌への影響を確認する。
方法: 一晩培養した大腸菌の懸濁液を生菌サンプルとして使用。段階希釈を行い、1×107~1×102個の大腸菌から、氷上静置時間=5分、処理回数=3回でEMA処理ありサンプル、ならびにEMA処理なしサンプルをそれぞれ調製した。各サンプルに対して、TB Green Premix Ex Taq (Tli RNaseH Plus)(製品コードRR420A)とThermal Cycler Dice Real Time System II を用いたリアルタイムPCRを行った(qPCR 20 μl系に各サンプルを2 μl使用、ターゲットは大腸菌LacZ、増幅サイズ 70 bp)
| <EMA処理なし> |
増幅曲線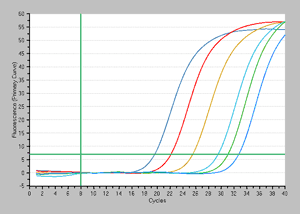 |
融解曲線 |
| <EMA処理あり> |
増幅曲線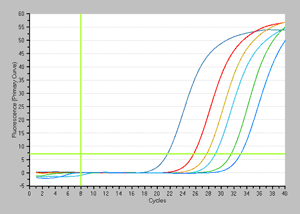 |
融解曲線 |
結果:EMA処理なし(左図)とEMA処理あり(右図)の結果の比較から、107~105 個では1.5~2サイクル程度のCt値の遅れが認められたが、それ以下の菌数では同等の結果が得られており、検出感度には影響がないことが分かる。
2. PCR増幅を抑制可能な死菌量
対象のグラム陰性菌について、EMA処理により、どの程度までの死菌量に対して死菌由来DNAからのPCR増幅を抑制できるかをあらかじめ確認しておくことが重要である。純培養した生菌から熱処理等により死菌を調製し、段階希釈して、本キットによるEMA処理あり/なしの結果を比較することにより、死菌抑制効果を確認する。
エンドポイントPCRの場合は、目的サイズのバンドの有無で完全に抑制可能な最大死菌量を確認し、その範囲内で実検体の測定を行う。(その範囲を超える死菌が存在する場合には、EMA処理により完全に抑制できない可能性がある。)死菌抑制効果が不十分な場合は、EMA処理回数を増やす、あるいはPCR増幅サイズを大きくするといった条件検討を行う。
リアルタイムPCRの場合には、完全に抑制可能な最大死菌量のEMA処理なしでのCt値(Xとする)を参考にすることで、検体サンプル中の死菌由来DNAが完全に処理されたかどうかを推測することも可能である。EMA処理なしサンプルのCt値(Y)がY > X であれば、死菌由来DNAはほぼ修飾されたと推測できる。Y ≦ X の場合には、検体中の死菌量が多く、EMA処理により完全には修飾できなかった可能性を考慮する必要がある。
【検討例2】
エンドポイントPCRの場合は、目的サイズのバンドの有無で完全に抑制可能な最大死菌量を確認し、その範囲内で実検体の測定を行う。(その範囲を超える死菌が存在する場合には、EMA処理により完全に抑制できない可能性がある。)死菌抑制効果が不十分な場合は、EMA処理回数を増やす、あるいはPCR増幅サイズを大きくするといった条件検討を行う。
リアルタイムPCRの場合には、完全に抑制可能な最大死菌量のEMA処理なしでのCt値(Xとする)を参考にすることで、検体サンプル中の死菌由来DNAが完全に処理されたかどうかを推測することも可能である。EMA処理なしサンプルのCt値(Y)がY > X であれば、死菌由来DNAはほぼ修飾されたと推測できる。Y ≦ X の場合には、検体中の死菌量が多く、EMA処理により完全には修飾できなかった可能性を考慮する必要がある。
【検討例2】
方法: 大腸菌生菌サンプルの一部を95℃で5分間熱処理したものを死菌サンプルとして使用。段階希釈を行い、1×107~1×102個の大腸菌死菌から、氷上静置時間=5分、処理回数=3回でEMA処理ありサンプル、ならびにEMA処理なしサンプルをそれぞれ調製した。各サンプルに対して、TB Green Premix Ex Taq (Tli RNaseH Plus)(製品コード RR420A)とThermal Cycler Dice Real Time System II を用いたリアルタイムPCRを行った(qPCR 20 μl系に各サンプルを2 μl使用、ターゲットは大腸菌LacZ、増幅サイズ 70 bp)。
| <EMA処理なし> |
増幅曲線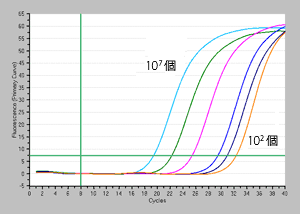 |
融解曲線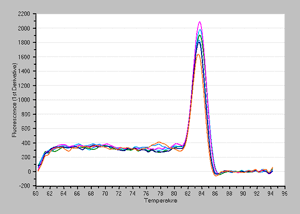 |
| <EMA処理なし> |
増幅曲線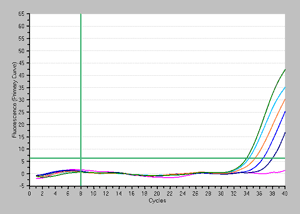 |
融解曲線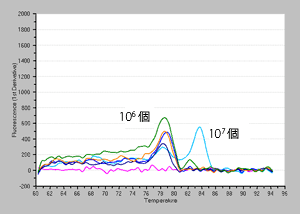 |
結果: 本キットを用いたEMA処理により、少なくとも105 個までの大腸菌の死菌を完全に抑制できることが分かる。この時、EMA処理なしの105個サンプルのCt値は25.4。大きく成分が異ならない検体を用いて同様にEMA処理~検出を行う場合、EMA処理なしの各サンプルがこのCt値(25.4)より大きな数値のCt値を示す場合、死菌由来DNAが完全に修飾されており、死菌由来DNAに起因する増幅は起こらないと考えることができ、増幅はすべて生菌由来DNAに起因するものと推測することができる。
3. 大腸菌を用いたモデル実験
| 方法: | 実際に近い状況で確認するため、103~107個の生菌に一定数(105個)の死菌を混合したものを用意し、EMA処理あり/なしの結果を比較した。 |
| <EMA処理なし> |
増幅曲線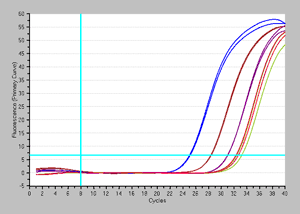 |
融解曲線 |
| <EMA処理あり> |
増幅曲線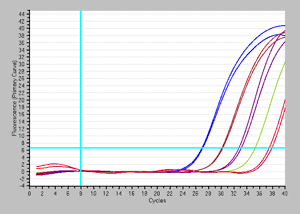 |
融解曲線 |
結果:EMA処理後には、死菌由来の増幅が抑制され、生菌が初発数依存的に検出できた。
