CRISPR/Cas9によるTetプロモーターのノックイン
ヒトHEK293細胞のCCR5遺伝子※上流領域に、ゲノム編集による相同組換え(HR:homologous recombination)によりテトラサイクリン発現誘導システム(Tet-On 3G Inducible Expression System)の応答ベクターのプロモーター配列(PTRE 3G)をノックインし、CCR5遺伝子のTet発現誘導を行った(図1)。
ノックイン用のドナープラスミドをIn-Fusion Cloning Kit(終売)により構築した。さらにCCR5遺伝子上流領域を標的としたsgRNAを設計し、sgRNA発現プラスミドを作製した(図1下部)。
次に、ヒトHEK293細胞にドナープラスミド、sgRNA発現プラスミド、Cas9発現プラスミドをトランスフェクションした。
※HEK293細胞のCCR5発現量は、qRT-PCR解析で検出限界以下である。
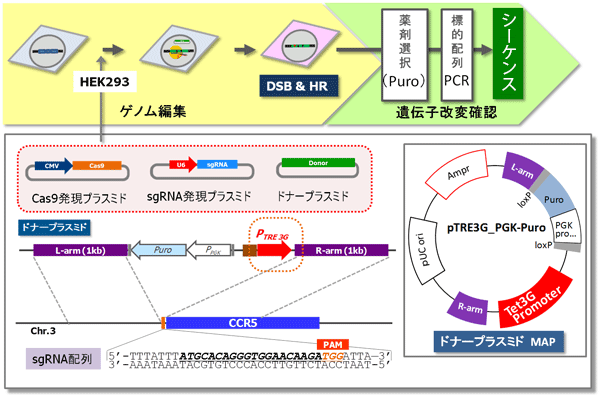
図1.実験フローとドナープラスミドの構造、sgRNAの標的配列
トランスフェクション後、Puromycinによる薬剤選択により、ノックインされた細胞をクローニングした。得られた7クローンについて、ノックインにより予想されるDNA構造をPCR増幅で確認した(図2)。7クローン中5クローンで予想されたサイズのDNA断片の増幅が確認された。さらに1クローン(Clone #1)のPCR産物をシーケンスし、塩基配列が正しくノックインがされていることを確認した。
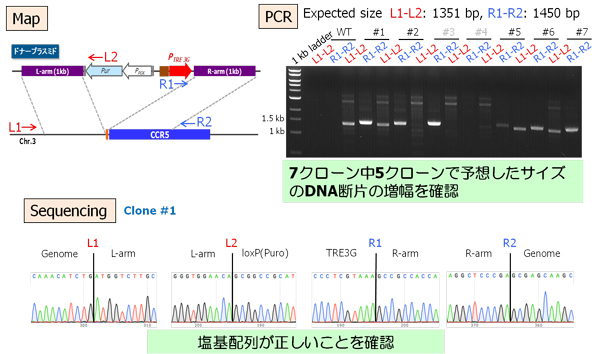
図2.ノックイン細胞クローンの標的部位のPCRとシーケンスによる確認
Tet発現誘導のために、Clone #1細胞に、Tet-Expressタンパク質(販売終了)を添加し、CCR5遺伝子を発現誘導した。Tet-Express添加後のCCR5遺伝子の発現量をqRT-PCRによって定量した(図3)。
Tet 3Gプロモーターをノックインした細胞では、Tet-Expressタンパク質による発現誘導により、CCR5遺伝子が発現誘導された。
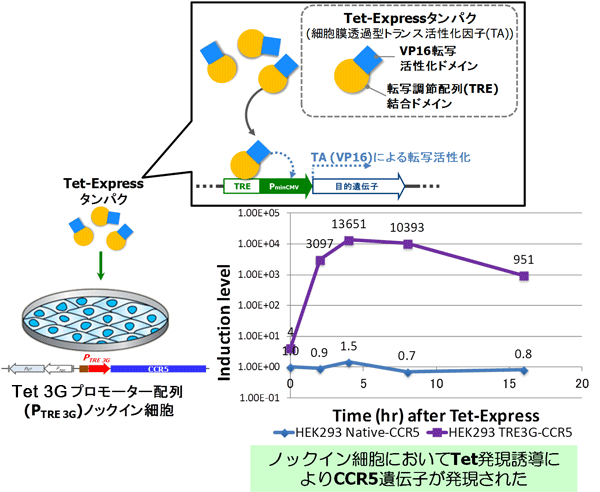
図3.ノックイン細胞におけるCCR5遺伝子のTet発現誘導
ゲノム編集関連製品についてはこちら
ゲノム編集の受託サービスについてはこちら
ノックイン用のドナープラスミドをIn-Fusion Cloning Kit(終売)により構築した。さらにCCR5遺伝子上流領域を標的としたsgRNAを設計し、sgRNA発現プラスミドを作製した(図1下部)。
次に、ヒトHEK293細胞にドナープラスミド、sgRNA発現プラスミド、Cas9発現プラスミドをトランスフェクションした。
※HEK293細胞のCCR5発現量は、qRT-PCR解析で検出限界以下である。

図1.実験フローとドナープラスミドの構造、sgRNAの標的配列
トランスフェクション後、Puromycinによる薬剤選択により、ノックインされた細胞をクローニングした。得られた7クローンについて、ノックインにより予想されるDNA構造をPCR増幅で確認した(図2)。7クローン中5クローンで予想されたサイズのDNA断片の増幅が確認された。さらに1クローン(Clone #1)のPCR産物をシーケンスし、塩基配列が正しくノックインがされていることを確認した。

図2.ノックイン細胞クローンの標的部位のPCRとシーケンスによる確認
Tet発現誘導のために、Clone #1細胞に、Tet-Expressタンパク質(販売終了)を添加し、CCR5遺伝子を発現誘導した。Tet-Express添加後のCCR5遺伝子の発現量をqRT-PCRによって定量した(図3)。
Tet 3Gプロモーターをノックインした細胞では、Tet-Expressタンパク質による発現誘導により、CCR5遺伝子が発現誘導された。

図3.ノックイン細胞におけるCCR5遺伝子のTet発現誘導
ゲノム編集関連製品についてはこちら
ゲノム編集の受託サービスについてはこちら
