ユーザー様実施例:人工ライブラリの作製
データご提供:東京大学大学院 農学生命科学研究科 小川様
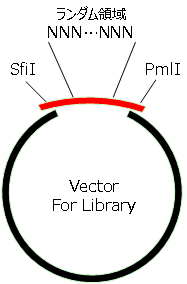
模式図
結果の表
In-Fusionを使った感想はいかがでしたか?
3種類のキットを使ってライブラリ作製操作を行ったが、このような比較実験はしたことがなかったので興味深かった。末端のオーバーラップ配列や、断片長によっては他のキットの方が効率が良くなる可能性も考えられるが、これまでの経験上、第一選択はIn-Fusionで問題ないと思う。
In-Fusionを使ったことがないユーザー様に一言お願いします。
クローニングの際に制限酵素サイトに悩むことがなくなります。また、ライゲーションよりも効率が良いので、特にライブラリ構築の際にはおすすめです。
実験概要
ランダム塩基配列を持つ遺伝子断片(75 bp)をベクター(制限酵素Sfi I、Pml Iで切断。約5,900 bp)にサブクローニングして、ペプチドライブラリを作製する予備検討を行った。切断したベクター 50 ng、インサート0~20 ngを10 μLの系で反応させ、1/10量をエレクトロポレーションにより大腸菌に導入した。菌液の全量の1/10量をプレートに播種して、生じたコロニー数をIn-Fusion HD、他社製品A、他社製品B間で比較した。模式図

結果
生じたコロニー数は表のようになった。他社製品Aはコロニー数は多いものの、インサートなしの場合でも無視できない数のコロニーが出現しており、ライブラリとしては不適であると判断された。In-Fusion HDおよび他社製品Bが、vector:insert=5:1の条件において、同程度のコロニー数の出現で極大値となった。今回のベクターや配列の条件では、In-Fusion HDと他社製品Bに大きな差は見られなかったが、価格面や使用時の手軽さ(他社製品Bは凍結保存しているので使用前に融解が必要)を考慮し、In-Fusion HD 5:1の条件を採用してライブラリ調製を行うこととした。結果の表
| vector 50 ngに対するinsert量(ng) | ||||||||
| 0 | 0.5 | 1 | 2 | 5 | 10 | 20 | ||
| コロニー数 | In-Fusion HD | 1 | 0 | 2 | 2 | 44 | 89 | 34 |
| 他社製品A | 11 | 98 | 87 | 98 | 51 | 383 | 299 | |
| 他社製品B | 0 | 2 | 12 | 10 | 43 | 78 | 47 | |
In-Fusionを使った感想はいかがでしたか?
3種類のキットを使ってライブラリ作製操作を行ったが、このような比較実験はしたことがなかったので興味深かった。末端のオーバーラップ配列や、断片長によっては他のキットの方が効率が良くなる可能性も考えられるが、これまでの経験上、第一選択はIn-Fusionで問題ないと思う。
In-Fusionを使ったことがないユーザー様に一言お願いします。
クローニングの際に制限酵素サイトに悩むことがなくなります。また、ライゲーションよりも効率が良いので、特にライブラリ構築の際にはおすすめです。
