UMI付き、微量total RNA(250 pg~1 μg)から方向性情報を持ったNGS用ライブラリー調製が可能
- ヒト、マウス、ラットの微量(250 pg~1 μg)のtotal RNA、または10~1,000個の細胞からRNA-Seq解析が可能
- UMI(分子バーコード)により、PCR増幅バイアスの影響とシーケンスエラーが軽減され、より正確なNGS解析が可能
- FFPEやLCM由来の分解を受けたRNAやcell-free RNAを含むすべての品質のRNAから信頼性の高い結果を取得
- バッファーの改良により、6.5時間で方向性情報を保持したRNA-Seqライブラリーを調製可能
- rRNA除去試薬の改良により、効果的にrRNAのリードを除去
- Unique Dual Index Kit(製品コード 634752~634756)と組み合わせることにより、最大384サンプルのマルチプレックス解析に対応可能
※
製品名を以下の通り変更いたしました。製品の組成や性能に変更はございません。(2026/1/9)
| 旧製品名 | 新製品名 |
| SMART-Seq Total RNA Pico Input with UMIs (ZapR Mammalian) | SMART-Seq Total RNA Library Prep with ZapR Depletion (with UMIs) |
注:SMART-Seq Total RNA Library Prep with ZapR Depletion (with UMIs)は、SMARTer Stranded Total RNA - Seq Kit v3 - Pico Input Mammalian(製品コード 634485~634488)のリニューアル製品で、rRNA除去試薬の性能が向上しています(図8参照)。既存製品とは異なりインデックスが別売りとなっていますので、必ず別途ご購入ください。(Unique Dual Index Kit参照)
新製品と既存製品の対応表はこちら(TB USA社のサイト)
新製品と既存製品の対応表はこちら(TB USA社のサイト)
※色文字での表示は、キャンペーン価格およびそのキャンペーン期間です。
※本キットにはIndexが含まれていません。Unique Dual Index Kit(製品コード 634752/634753/634754/634755/634756)を別途ご購入ください。
※本キットにはIndexが含まれていません。Unique Dual Index Kit(製品コード 634752/634753/634754/634755/634756)を別途ご購入ください。
カートにいれる
WEB会員ログインが必要です
お知らせ 2025/12/9
サンプルのインプット量が最大1 μgまで対応可能になりました。・変更前:250 pg~10 ng → ・変更後:250 pg~1 μg
インプット量拡張に伴うPCRの推奨条件は以下の通りです。その他のプロトコールはUser Manualをご参照ください。
| Input | PCR 1 cycles (cDNA amplification; Section V.B) | PCR 2 cycles* (Library amplification; Section V.E) |
|---|---|---|
| >10 ng~100 ng | 5 | 14 |
| >100 ng~200 ng | 5 | 12 |
| >200 ng~1 µg | 3 | 12 |
製品説明
SMART-Seq Total RNA Library Prep with ZapR Depletion (with UMIs)は、250 pg~1 μgのヒト、マウス、ラットのtotal RNAまたは10~1,000個のインタクトな細胞から、イルミナ社次世代シーケンサー対応のライブラリーを調製するキットです。高品質のRNAはもちろん、FFPEやLCMサンプルから調製された分解の進んだ低品質RNA*も使用することができます(* 低品質RNAの場合は500 pg~10 ng)。本キットは、SMART(Switching Mechanism at 5' End of RNA Template)テクノロジーを用いており、逆転写およびPCR増幅ステップでインデックスとアダプターを組み込むシームレスなワークフローにより、Strand情報を保持したライブラリーを調製できます。更に、逆転写ステップで8塩基の個別の分子バーコード(Unique Molecular Identifier:UMI)も付加されるため、PCR増幅バイアスによる影響とシーケンスエラーが軽減され、より正確で信頼性の高いNGS解析が可能となります。
本キットのワークフローにはrRNA特異的プローブを使用した独自技術によってrRNA(および一部ミトコンドリアRNA)由来のcDNAを除去する工程が含まれているため、別途リボゾームRNA除去試薬を用意する必要はなく、rRNAへのマッピングが低い再現性の高いデータが得られます。
ライブラリー調製、精製を含む全ての操作は6.5時間で終了し、別売りのUnique Dual Index Kit(製品コード 634752~634756)と組み合わせることにより、最大384サンプルのマルチプレックス解析に対応可能です。
本キットのワークフローにはrRNA特異的プローブを使用した独自技術によってrRNA(および一部ミトコンドリアRNA)由来のcDNAを除去する工程が含まれているため、別途リボゾームRNA除去試薬を用意する必要はなく、rRNAへのマッピングが低い再現性の高いデータが得られます。
ライブラリー調製、精製を含む全ての操作は6.5時間で終了し、別売りのUnique Dual Index Kit(製品コード 634752~634756)と組み合わせることにより、最大384サンプルのマルチプレックス解析に対応可能です。
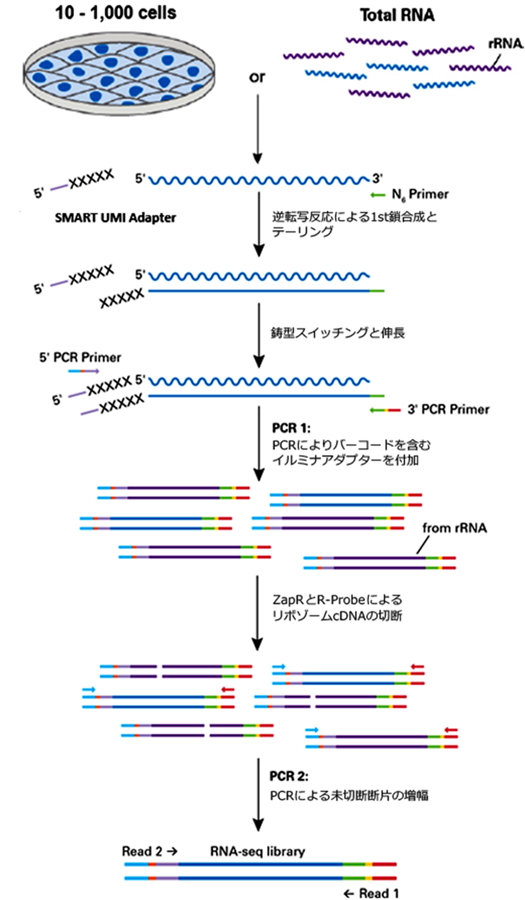
図1. 実験フローチャート

図2. SMART-Seq Total RNA Library Prep with ZapR Depletion (with UMIs)を用いて調製された最終ライブラリーの構造
Unique Dual Index Kit(製品コード 634752~634756)を用いて付加された配列には、イルミナ社シーケンサーのフローセルでクラスタリングできる配列(P7、P5、Index 1 [i7]、Index 2 [i5])と、シーケンスプライマーRead 2およびRead 1を認識する領域が含まれている。Read 1では元のRNAのアンチセンス側の配列が得られ、Read 2では元のRNAのセンス側の配列が得られる。Read 2の最初の8塩基はUMIで、その下流にUMIリンカー3塩基(NNN)とSMART UMI Adapter由来の3塩基(XXX)が続く。paired-end sequencingを行った場合、SMART UMI Adapter由来の3塩基(XXX)をマッピングの前にトリミングする必要がある。
Unique Dual Index Kit(製品コード 634752~634756)を用いて付加された配列には、イルミナ社シーケンサーのフローセルでクラスタリングできる配列(P7、P5、Index 1 [i7]、Index 2 [i5])と、シーケンスプライマーRead 2およびRead 1を認識する領域が含まれている。Read 1では元のRNAのアンチセンス側の配列が得られ、Read 2では元のRNAのセンス側の配列が得られる。Read 2の最初の8塩基はUMIで、その下流にUMIリンカー3塩基(NNN)とSMART UMI Adapter由来の3塩基(XXX)が続く。paired-end sequencingを行った場合、SMART UMI Adapter由来の3塩基(XXX)をマッピングの前にトリミングする必要がある。
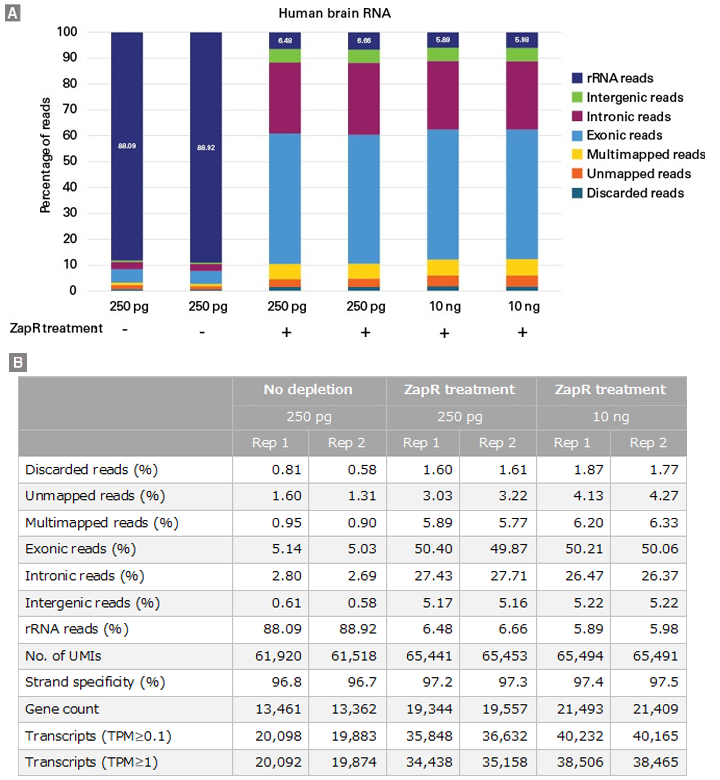
図3. 異なるRNAインプット量から調製したライブラリーのrRNA由来cDNA除去効果と遺伝子数、トランスクリプト数の評価
SMART-Seq Total RNA Library Prep (with UMIs)を用いて、250 pgおよび10 ngのヒト脳total RNAからライブラリーを調製後、キットに含まれるZapR Mammalian rRNA Depletion KitによるrRNA由来cDNA除去処理(ZapR treatment)を実施したものと実施しないもので比較した。ZapR treatmentしたものはPCR増幅による濃縮後、ZapR treatmentしないものはそのままシーケンスに使用した。それぞれのライブラリーについて3×106ペアエンドリードをCogentAPで解析し、その結果を棒グラフ(パネルA)と表(パネルB)に示した。
250 pg 、10 ngのいずれのインプット量でも、ZapR treatmentしたライブラリーのrRNA readsの割合は10%以下となっており、幅広いレンジでrRNA由来cDNAが効率よく除去されることが示された。また、ZapR treatmentしたライブラリーでは、遺伝子数、トランスクリプト数が増加した。
SMART-Seq Total RNA Library Prep (with UMIs)を用いて、250 pgおよび10 ngのヒト脳total RNAからライブラリーを調製後、キットに含まれるZapR Mammalian rRNA Depletion KitによるrRNA由来cDNA除去処理(ZapR treatment)を実施したものと実施しないもので比較した。ZapR treatmentしたものはPCR増幅による濃縮後、ZapR treatmentしないものはそのままシーケンスに使用した。それぞれのライブラリーについて3×106ペアエンドリードをCogentAPで解析し、その結果を棒グラフ(パネルA)と表(パネルB)に示した。
250 pg 、10 ngのいずれのインプット量でも、ZapR treatmentしたライブラリーのrRNA readsの割合は10%以下となっており、幅広いレンジでrRNA由来cDNAが効率よく除去されることが示された。また、ZapR treatmentしたライブラリーでは、遺伝子数、トランスクリプト数が増加した。
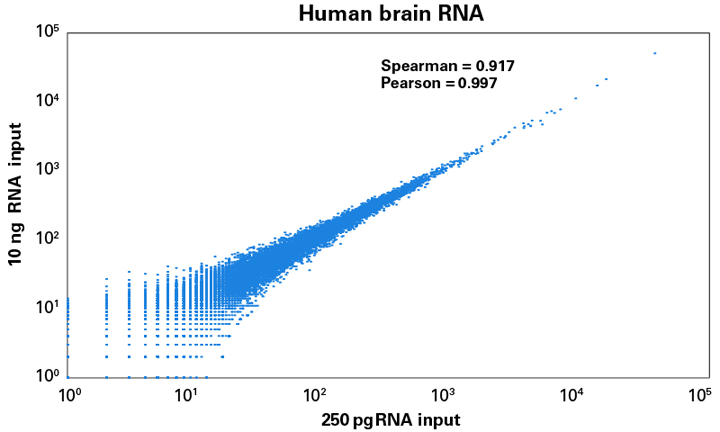
図4. インプット量の異なるサンプル間での相関性
SMART-Seq Total RNA Library Prep with ZapR Depletion (with UMIs) を用いてヒト脳total RNA 250 pgおよび10 ngからRNA-Seqライブラリーを調製し、それぞれのライブラリーについて、3×106ペアエンドリードをCogentAPで解析して相関性を調べた。その結果、インプット量250 pgと10 ngの間で高い相関性が得られた。
SMART-Seq Total RNA Library Prep with ZapR Depletion (with UMIs) を用いてヒト脳total RNA 250 pgおよび10 ngからRNA-Seqライブラリーを調製し、それぞれのライブラリーについて、3×106ペアエンドリードをCogentAPで解析して相関性を調べた。その結果、インプット量250 pgと10 ngの間で高い相関性が得られた。
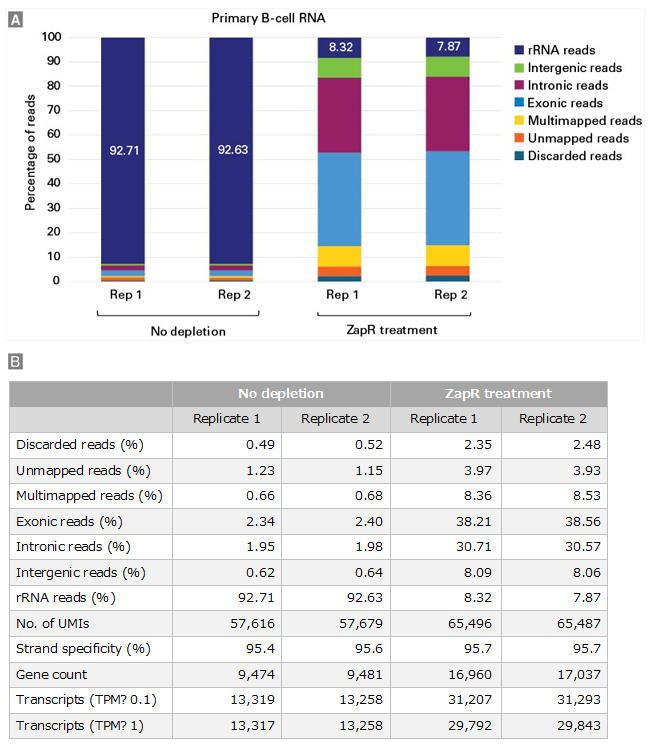
図5. ヒト初代B細胞から調製したtotal RNA-Seqライブラリーの遺伝子数とトランスクリプト数
SMART-Seq Total RNA Library Prep (with UMIs)を用いて、ヒト初代B細胞のtotal RNA 1 ngからライブラリーを調製後、キットに含まれるZapR Mammalian rRNA Depletion KitによるrRNA由来cDNA除去処理(ZapR treatment)を実施したものと実施しないもので比較した。ZapR treatmentしたものはPCR増幅による濃縮後、ZapR treatmentしないものはそのままシーケンスに使用した。それぞれのライブラリーについて、3×106ペアエンドリードをCogentAPで解析し、その結果を棒グラフ(パネルA)と表(パネルB)に示した。
その結果、ZapR treatmentしたライブラリーのrRNA readsの割合は10%以下となっており、rRNA由来cDNAが効率よく除去されることが示された。また、ZapR treatmentしたライブラリーでは、遺伝子数、トランスクリプト数が増加した。
SMART-Seq Total RNA Library Prep (with UMIs)を用いて、ヒト初代B細胞のtotal RNA 1 ngからライブラリーを調製後、キットに含まれるZapR Mammalian rRNA Depletion KitによるrRNA由来cDNA除去処理(ZapR treatment)を実施したものと実施しないもので比較した。ZapR treatmentしたものはPCR増幅による濃縮後、ZapR treatmentしないものはそのままシーケンスに使用した。それぞれのライブラリーについて、3×106ペアエンドリードをCogentAPで解析し、その結果を棒グラフ(パネルA)と表(パネルB)に示した。
その結果、ZapR treatmentしたライブラリーのrRNA readsの割合は10%以下となっており、rRNA由来cDNAが効率よく除去されることが示された。また、ZapR treatmentしたライブラリーでは、遺伝子数、トランスクリプト数が増加した。
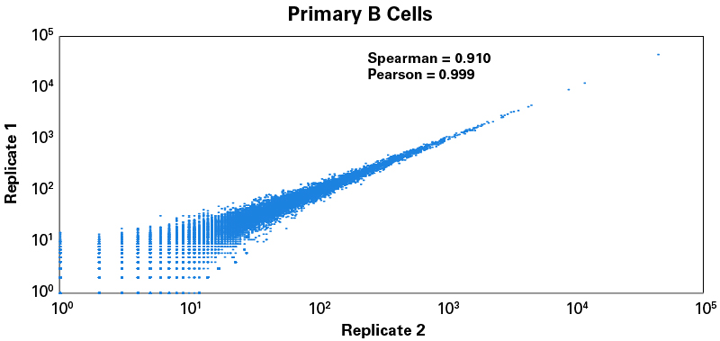
図6. ヒト初代B細胞から調製したライブラリーのレプリケート間での比較
SMART-Seq Total RNA Library Prep with ZapR Depletion (with UMIs) を用いて、1 ngのヒト初代B細胞のtotal RNAからduplicateでライブラリーを調製し、3×106ペアエンドリードをCogentAPで解析して相関性を調べた。その結果、レプリケート間で高い相関性が示された。
SMART-Seq Total RNA Library Prep with ZapR Depletion (with UMIs) を用いて、1 ngのヒト初代B細胞のtotal RNAからduplicateでライブラリーを調製し、3×106ペアエンドリードをCogentAPで解析して相関性を調べた。その結果、レプリケート間で高い相関性が示された。
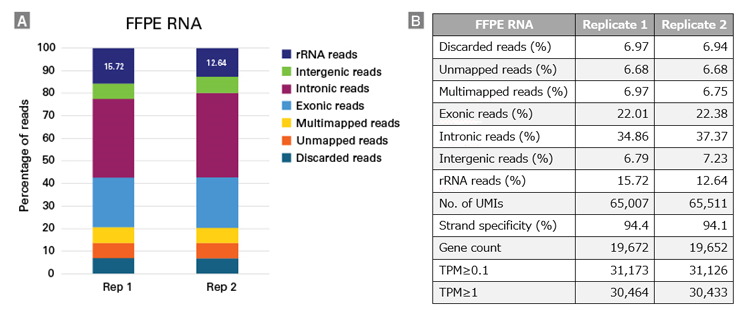
図7. 分解を受けたFFPE RNAサンプルから調製されたライブラリーの評価
SMART-Seq Total RNA Library Prep with ZapR Depletion (with UMIs) を用いて、10 ngのFFPE RNA(RIN=3、DV200=77%)からduplicateでライブラリーを調製し、3×106ペアエンドリードをCogentAPで解析し、その結果を棒グラフ(パネルA)と表(パネルB)に示した。その結果、分解を受けたサンプルからでもレプリケート間で高い再現性が示された。
SMART-Seq Total RNA Library Prep with ZapR Depletion (with UMIs) を用いて、10 ngのFFPE RNA(RIN=3、DV200=77%)からduplicateでライブラリーを調製し、3×106ペアエンドリードをCogentAPで解析し、その結果を棒グラフ(パネルA)と表(パネルB)に示した。その結果、分解を受けたサンプルからでもレプリケート間で高い再現性が示された。
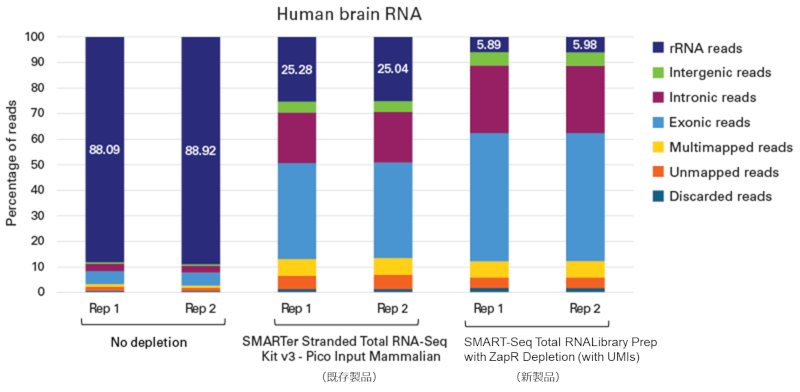
図8. rRNA除去試薬の新/既存製品比較
SMART-Seq Total RNA Library Prep (with UMIs)を用いて、250 ngのヒト脳total RNAからライブラリーを調製後、キットに含まれるZapR Mammalian rRNA Depletion KitによるrRNA由来cDNA除去処理(ZapR treatment)を実施し、PCR増幅により濃縮した。コントロールとしてZapR treatmentしていないライブラリーをそのまま用いた(No depletion)。更に、SMARTer Stranded Total RNA-Seq Kit v3 - Pico Input Mammalian(既存製品)を用いて、250 ngのヒト脳RNAからライブラリーを調製した。
それぞれのライブラリーについて、3×106ペアエンドリードをCogentAPで解析し、リード分布を比較した。
ZapR Mammalian rRNA Depletion Kitで処理したライブラリーのリード分布は、SMARTer Stranded Total RNA-Seq Kit v3 - Pico Input Mammalianで調製したライブラリーと比較したところ、rRNAのリードの割合が減少してExonのリードの割合が増加し、rRNA除去試薬の性能向上が示された。
SMART-Seq Total RNA Library Prep (with UMIs)を用いて、250 ngのヒト脳total RNAからライブラリーを調製後、キットに含まれるZapR Mammalian rRNA Depletion KitによるrRNA由来cDNA除去処理(ZapR treatment)を実施し、PCR増幅により濃縮した。コントロールとしてZapR treatmentしていないライブラリーをそのまま用いた(No depletion)。更に、SMARTer Stranded Total RNA-Seq Kit v3 - Pico Input Mammalian(既存製品)を用いて、250 ngのヒト脳RNAからライブラリーを調製した。
それぞれのライブラリーについて、3×106ペアエンドリードをCogentAPで解析し、リード分布を比較した。
ZapR Mammalian rRNA Depletion Kitで処理したライブラリーのリード分布は、SMARTer Stranded Total RNA-Seq Kit v3 - Pico Input Mammalianで調製したライブラリーと比較したところ、rRNAのリードの割合が減少してExonのリードの割合が増加し、rRNA除去試薬の性能向上が示された。
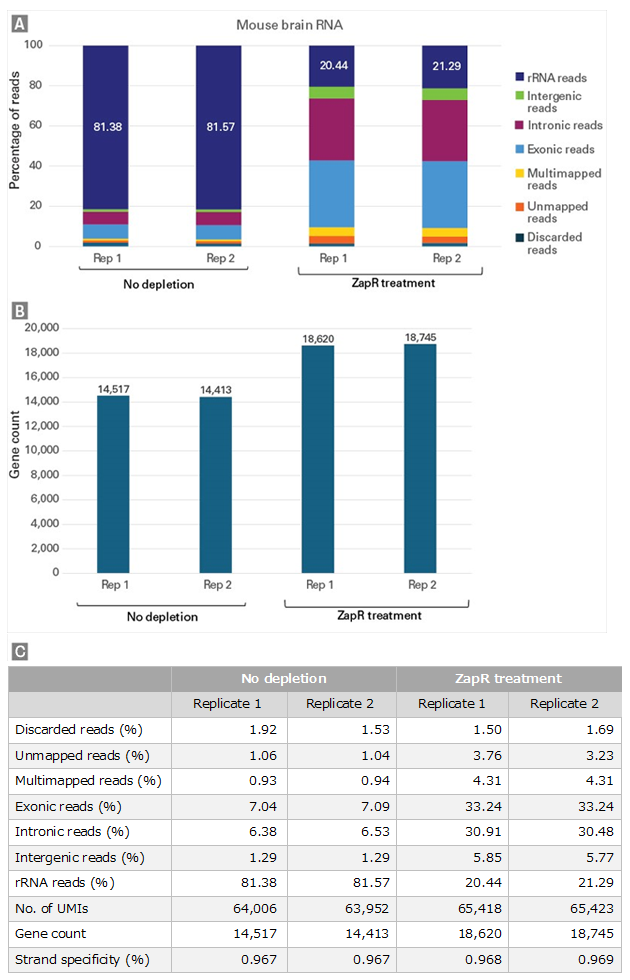
図9. マウス脳total RNA-Seq ライブラリーのrRNA除去効率と遺伝子数、およびトランスクリプト数
SMART-Seq Total RNA Library Prep (with UMIs)を用いて、250 ngのマウス脳total RNAからライブラリーを調製後、キットに含まれるZapR Mammalian rRNA Depletion KitによるrRNA由来cDNA除去処理(ZapR treatment)を実施したものと実施しないもので比較した。ZapR treatmentしたものはPCR増幅による濃縮後、ZapR treatmentしないものはそのままシーケンスに使用した。それぞれのライブラリーについて、3×106ペアエンドリードをCogentAPで解析し、リード分布(パネルAとパネルC)と遺伝子数(パネルBとパネルC)を示した。
その結果、ZapR treatmentしたライブラリーでは、遺伝子数が増加した。
SMART-Seq Total RNA Library Prep (with UMIs)を用いて、250 ngのマウス脳total RNAからライブラリーを調製後、キットに含まれるZapR Mammalian rRNA Depletion KitによるrRNA由来cDNA除去処理(ZapR treatment)を実施したものと実施しないもので比較した。ZapR treatmentしたものはPCR増幅による濃縮後、ZapR treatmentしないものはそのままシーケンスに使用した。それぞれのライブラリーについて、3×106ペアエンドリードをCogentAPで解析し、リード分布(パネルAとパネルC)と遺伝子数(パネルBとパネルC)を示した。
その結果、ZapR treatmentしたライブラリーでは、遺伝子数が増加した。
内容
SMART-Seq Total RNA Library Prep with ZapR Depletion (with UMIs)(製品コード 634354/634355/634356)
- SMART-Seq Total RNA Library Prep (with UMIs)*
Package 1
・SMART UMI-TSO Mix v3
Package 2
・SMART Pico Oligos Mix v3
・5X First-Strand Buffer
・SMARTScribe II Reverse Transcriptase
・RNase Inhibitor (40 U/µl)
・SeqAmp DNA Polymerase
・SeqAmp CB PCR Buffer (2X)
・Tris Buffer (5 mM)
・PCR2 Primers v3
・Nuclease-Free Water
・10X Lysis Buffer
- ZapR Mammalian rRNA Depletion Kit*
Package 1
・R-Probes
・Control Total RNA (1 µg/µl)
Package 2
・ZapR Enzyme
・ZapR Buffer (10X)
保存
- SMART-Seq Total RNA Library Prep (with UMIs)
- Package 1:-70℃
- Package 2:-20℃
- ZapR Mammalian rRNA Depletion Kit
- Package 1:-70℃
- Package 2:-20℃
本製品以外に必要な試薬、器具、機器(主なもの)
下記の製品は本キット中に含まれません。下記の製品はユーザーマニュアルに記載のProtocolで使用できることが確認されています。
Index Kit
- Unique Dual Index Kit (1-24)(製品コード 634756)
UDI No. U001-U024 - Unique Dual Index Kit (1-96)(製品コード 634752)
UDI No, U001-U096 - Unique Dual Index Kit (97-192)(製品コード 634753)
UDI No, U097-U192 - Unique Dual Index Kit (193-288)(製品コード 634754)
UDI No, U193-U288 - Unique Dual Index Kit (289-384)(製品コード 634755)
UDI No, U289-U384
サンプル調製(セルソーティング)
- 8-tube strips(Thermo Fisher Scientific社, Cat. No. AB0264)またはセルソーティングに適したPCR tube strips、96 well plates
- (オプション)BD FACS Pre-Sort Buffer(BD社 Cat. No. 563503)
- Microplate film(USA Scientific社, Cat. No. 2920-0010)for sealing tubes/plates before sorting
- Aluminum single tab foil seal(USA Scientific社, Cat. No. 2938-4100)または cap strips(Thermo Fisher Scientific社, Cat. No. AB0784/AB0850)for sealing tubes/plates after sorting
- ドライアイス(細胞の凍結用)
- (オプション)SMART-Seq HT Kit Lysis Components(製品コード 634439)または10× Lysis Buffer(製品コード 635013)
cDNA合成、PCR増幅
- PCRサーマルサイクラー 2台(cDNA合成用、PCR増幅用)
- Nuclease-free, PCR-grade, thin-wall PCR strips(0.2 ml PCR 8-tube strip; USA Scientific社, Cat. No.1402-4700)または同様のPCR-grade, thin-wall PCR tubes, strips、96-well plates
- Nuclease-free, low-adhesion 1.5 ml tubes(USA Scientific社, Cat. No. 1415-2600)、DNA LoBind tubes(Eppendorf社, Cat. No. 022431021)または同様の核酸低吸着性チューブ
- Thermo Scientific Adhesive PCR Plate Seals(Thermo Fisher Scientific社, Cat. No. AB0558)for 96-well plates or cap strips(Thermo Fisher Scientific社, Cat. No. AB0784/AB0850)for 8-tube strips
Beads精製
- NucleoMag NGS Clean-up and Size Select(5 ml size:製品コード 744970.5;50 ml size:製品コード 744970.50;500 ml size:製品コード 744970.500)
※NucleoMagの代わりにAMPure XP PCR purification kit(Beckman Coulter社 5 ml size:Cat. No. A63880;60 ml size:Cat. No. A63881)も使用可能 - 80%エタノール(用時調製)
- Magnetic Separation Device
- 24~96サンプルの場合:Magnetic Stand 96(Thermo Fisher Scientific社 Cat. No. AM10027)、V-bottom plates(500 μl; VWR, Cat. No. 47743-996)、adhesive PCR Plate Seals(Thermo Fisher Scientific社, Cat. No. AB0558)と組み合わせて使用
- 1.5 mlチューブ(ライブラリーのプール用)の場合:Magnetic Stand (2 tubes)(製品コード 631964)、8-tube strips(Thermo Fisher Scientific社, Cat. No. AB0264)、またはnuclease-free, PCR-grade, thin-wall PCR tubes、strips、96-well plates
cDNA定量、ライブラリー定量(必要に応じて選択)
- High Sensitivity DNA Kit(Agilent Technologies社, Cat. No. 5067-4626)、High Sensitivity D5000 ScreenTape(Agilent Technologies社, Cat. No. 5067-5592)、または同等の高感度電気泳動法
- Quant-iT PicoGreen dsDNA Assay Kit(Thermo Fisher Scientific社, Cat. No. P11496)または Qubit dsDNA HS Assay Kit(Thermo Fisher Scientific社, Cat. No. Q32851 or Q32854)
- Library Quantification Kit(製品コード 638324)
その他
- シングルチャンネルピペット:10 μl、20 μlおよび200 μl
- 8連、あるいは12連チャンネルピペット:10 μl
- フィルターピペットチップ:10 μl、20 μl、および200 μl
- 1.5 mlチューブ用微量遠心機
- 0.2 mlチューブまたはstrip用微量遠心機
この製品を見た人は、
こんな製品も見ています
-
SMARTer® Stranded Total RNA-Seq Kit v2 - Pico Input Mammalian
微量FFPEサンプルからもRNA-Seqが可能
-

SMART-Seq® Total RNA Mid Input
様々な生物種のRNAから方向性の情報をもつNGS用ライブラリー調製が可能
-

Unique Dual Index Kits
イルミナ社NGSライブラリー調製キット用インデックスキット
-

SMART-Seq® Stranded Kit
シングルセルまたはtotal RNA 10 pgからの方向性情報を持ったtotal RNA-Seq解析
-

SMART-Seq® Total RNA Single Cell Library Prep with ZapR® Depletion
シングルセルまたはtotal RNA 10 pgからの方向性情報を持ったtotal RNA-Seq解析
-

SMART-Seq® Total RNA Library Prep with ZapR® Depletion
微量total RNA(250 pg~1 μg)から方向性情報を持ったNGS用ライブラリー調製が可能
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。
