ハイスループットなシングルセルDNA-Seqキット
- ゴールドスタンダードのPicoPLEX技術を用いたWGAキット
- プレートベースのアッセイを大幅に上回る約1,500 細胞/反応のハイスループット解析を実現
- 単一細胞からのコピー数変異(CNV)解析、単一塩基変異(SNV)解析
- 一度の解析で最大8サンプルを解析
製品説明
Shasta WGA Kitは、illuminaプラットフォーム用の高品質なscDNA-Seqライブラリーを作製します。タカラバイオのPicoPLEX全ゲノム増幅(WGA)技術を用いることで、再現性の高いコピー数変異(CNV)解析、単一塩基変異(SNV)解析、インデル解析を行うことができます。 Shasta WGA Kit は、Shastaシングルセル自動調製システムを用いたハイスループットな自動化ワークフローに最適化されており、これまでのプレートベースのシングルセルWGAを大幅に上回る、1,000~1,500細胞からのscDNA-Seqライブラリー作製を可能にします。
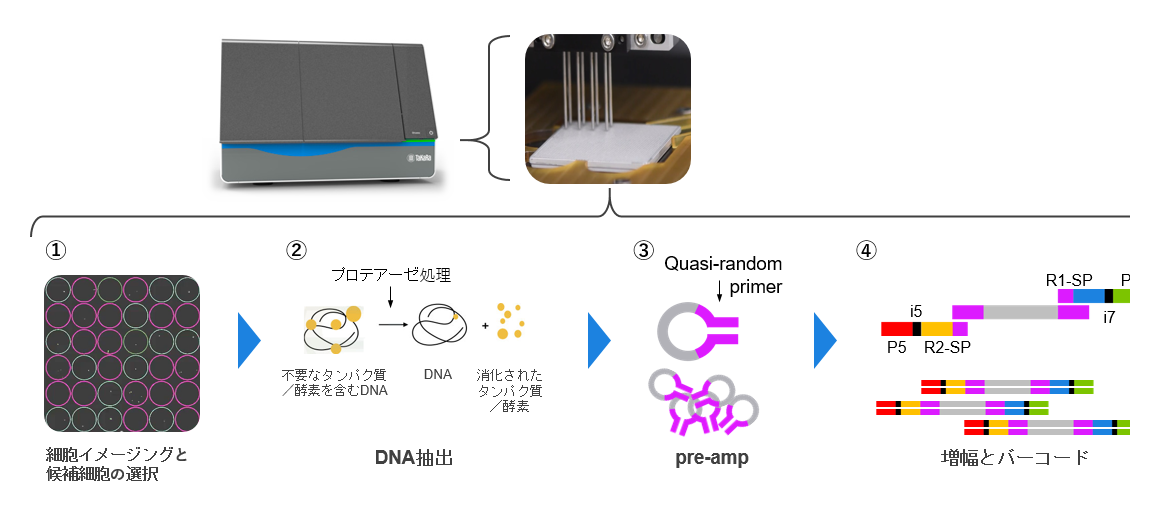
図1. Shasta WGA Kitワークフロー
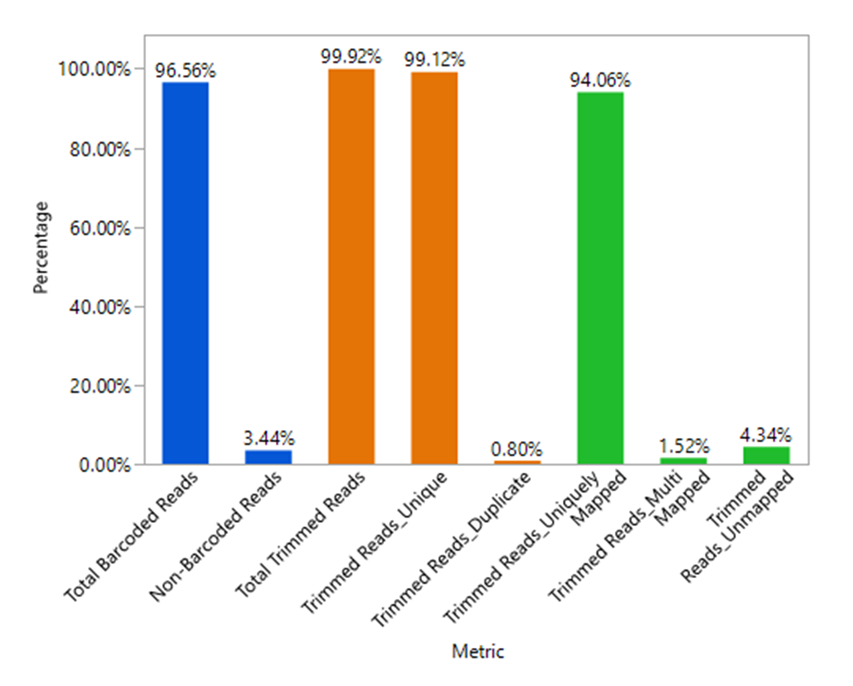
図2. 4つの異なる細胞株のシングルセルから作製したShasta WGAライブラリーのQC指標
ライブラリーのシーケンシングは、NextSeq 500/550 High Output Kitと150サイクルカートリッジ(リード長2 x 75 bp)を使用し、PhiXスパイクイン率は18.81%で実施した。96.56%のリードがバーコード化に成功し、バーコード化された単一細胞について、アダプタートリミング後、バーコード化されたリードの99.12%がユニークリードであり、バーコード化されたリードの94.06%がhg38参照ゲノムにユニークにマッピングされた。
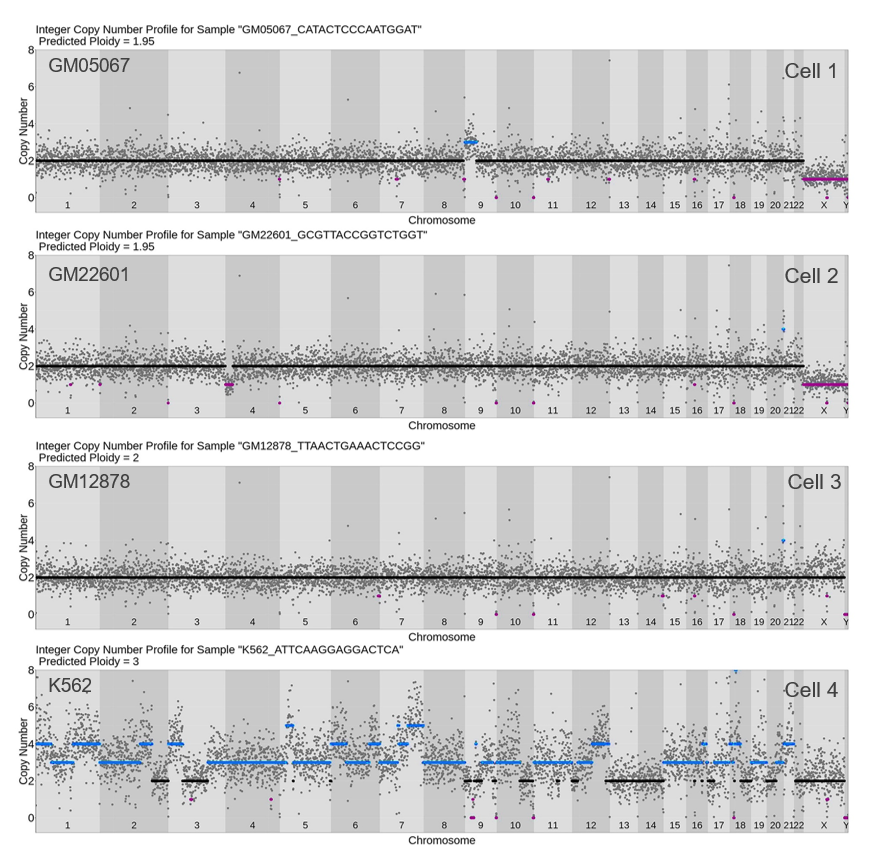
図3. Shasta WGAキットを用いてシングルセルから作成したCNP解析例
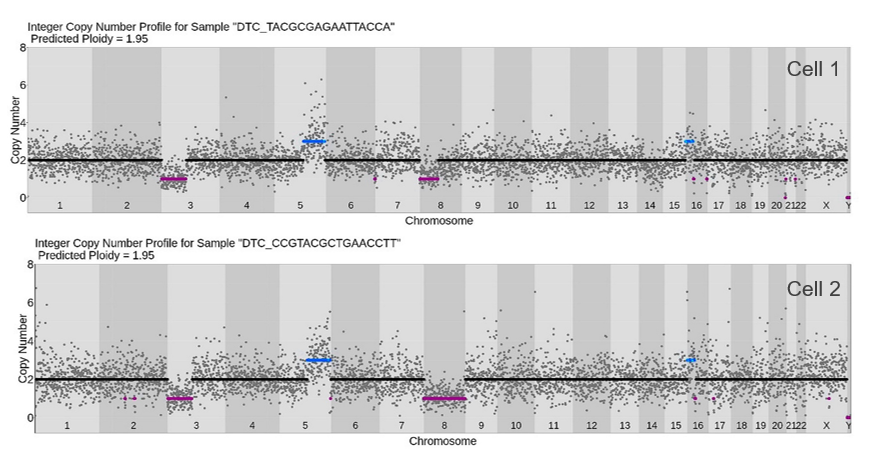
図4. 単一の淡明細胞型腎細胞がん(ccRCC)からShasta WGAキットを用いて作成したCNPから、ccRCCに一般的に関連するCNVを同定
Cell 1とCell 2はともに3番染色体のp-armの欠損(-3p)、5番染色体のq-armの獲得(+5q)、16番染色体のp-armの部分的獲得(+16pter)を有する。-3pはccRCCの細胞遺伝学的特徴であり、2013年の研究では417人の患者の91%が-3pの変異を有していた(Nature 499, 43-49 (2013))。この領域は、ccRCCで最もよく変異する4遺伝子を包含している(VHL、PBRM1、BAP1、SETD2)。また、+5qはccRCCでよくみられるCNVであり、2013年の研究では417人の患者の67%が+5qの変異を有していた。加えて、Cell 1では8番染色体のp-armが欠損(-8p)し、Cell 2では8番染色体全体が欠損していた。8番染色体の一部または全部の欠損は、以前に行われたccRCC患者の研究でも報告されており、典型的にはTCEB1異と関連している(Sato et al. Nature Genetics, (2013)45: 860-867)

図1. Shasta WGA Kitワークフロー
- ① ナノチップ上での細胞イメージングとシングルセルの選択
- ② オンチップでのDNA抽出
- ③ Quasi-random primerを含むpre-amplification mixの添加
- ④ forwardおよびreverseのindex primerを使用してDNAが増幅され、最終的なDNA-Seqライブラリーを構築

図2. 4つの異なる細胞株のシングルセルから作製したShasta WGAライブラリーのQC指標
ライブラリーのシーケンシングは、NextSeq 500/550 High Output Kitと150サイクルカートリッジ(リード長2 x 75 bp)を使用し、PhiXスパイクイン率は18.81%で実施した。96.56%のリードがバーコード化に成功し、バーコード化された単一細胞について、アダプタートリミング後、バーコード化されたリードの99.12%がユニークリードであり、バーコード化されたリードの94.06%がhg38参照ゲノムにユニークにマッピングされた。

図3. Shasta WGAキットを用いてシングルセルから作成したCNP解析例
- Cell 1(GM05067細胞): 9p染色体における約45 Mbの特徴的なセグメント増幅が特異的に検出された。
- Cell 2(GM22601細胞): 4p染色体での約25 Mbの欠失を検出した。
- Cell 3(GM12878細胞): CNVのない2倍体を示し、15 pgのNA12878 gDNAを用いて作製したWGAライブラリーと同等の性能を示した。
- Cell 4(K562細胞): 3倍体の予測された倍数性を示した。ハイパープロイディーは、急速に増殖するがん細胞において一般的な現象である。

図4. 単一の淡明細胞型腎細胞がん(ccRCC)からShasta WGAキットを用いて作成したCNPから、ccRCCに一般的に関連するCNVを同定
Cell 1とCell 2はともに3番染色体のp-armの欠損(-3p)、5番染色体のq-armの獲得(+5q)、16番染色体のp-armの部分的獲得(+16pter)を有する。-3pはccRCCの細胞遺伝学的特徴であり、2013年の研究では417人の患者の91%が-3pの変異を有していた(Nature 499, 43-49 (2013))。この領域は、ccRCCで最もよく変異する4遺伝子を包含している(VHL、PBRM1、BAP1、SETD2)。また、+5qはccRCCでよくみられるCNVであり、2013年の研究では417人の患者の67%が+5qの変異を有していた。加えて、Cell 1では8番染色体のp-armが欠損(-8p)し、Cell 2では8番染色体全体が欠損していた。8番染色体の一部または全部の欠損は、以前に行われたccRCC患者の研究でも報告されており、典型的にはTCEB1異と関連している(Sato et al. Nature Genetics, (2013)45: 860-867)
内容
Shasta Whole Genome Amplification Kit - 2 Chip(製品コード 640286)
| Shasta Whole-Genome Amplification Reagents | 1 Kit |
| Shasta Long Indexing Primer Set - A(製品コード 640283) | 2 Chip |
| Single-Cell 350v Chip(製品コード 640019) | 2 Chip |
| Single-Cell Loading Kit(製品コード 640206) | 2 Kit |
| Single-Cell Collection Kit(製品コード 640212) | 2 Kit |
保存
Shasta Whole-Genome Amplification Reagents -20℃
その他 室温
その他 室温
この製品を見た人は、
こんな製品も見ています
-

ICELL8 Blank Chip(cxシステム用)
-

SMART-Seq® Total RNA Single Cell Library Prep with ZapR® Depletion
シングルセルまたはtotal RNA 10 pgからの方向性情報を持ったtotal RNA-Seq解析
-

16S DNA Quantitative Standard for Microbiome
細菌叢解析用16S rDNA人工核酸標準物質
-

Shasta™ Total RNA-Seq Kit
ノンコーディングRNAを含むTotal RNA-Seqライブラリーを構築
-
SMART-Seq® Pro Application Kit - 2 Chip(cxシステム用)
ICELL8 cxシステム用シングルセル全長mRNA-Seq解析キット
-

PicoPLEX® Gold Single Cell DNA-Seq Kit
シングルセルからのDNAライブラリー調製
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。
