酵母、アスペルギルス菌用形質転換システム
概要
Aureobasidin A(オーレオバシジンA)耐性形質転換システム12)は、抗真菌抗生物質Aureobasidin A(AbA)とAbA耐性遺伝子を選択マーカーとして含むベクターを用いた、酵母または一部のAspergillus属糸状菌のための形質転換システムである。AbA耐性遺伝子は優性遺伝子のため、1または数コピーのプラスミドのみでも、形質転換体はAbAに対して耐性となる。また、栄養要求性変異を持たない野生型酵母や実用酵母(一倍体~多倍体)を宿主としたときも、高い効率で形質転換体が得られる。
<Aureobasidin A(オーレオバシジンA)耐性形質転換システムの概要>
* Aureobasidin A耐性酵母形質転換システムはS. cerevisiae株全般に使用できる形質転換系として開発された系であるが、S. cerevisiaeだけでなく、分類学上S. cerevisiaeの近縁種と考えられるKluyveromyces marxianusやK. lactis、Candida glabrataなどの形質転換系としても有用なシステムである(Application:pAUR101を用いたK. marxianus, K. lactis, およびC. glabrataの形質転換参照)。
Aureobasidin A 耐性形質転換システム取扱説明書
<Aureobasidin A(オーレオバシジンA)耐性形質転換システムの概要>
| 形質転換宿主 | AbA耐性遺伝子 | 大腸菌・酵母 シャトルベクター |
ベクターの特長 |
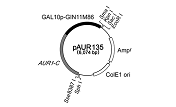
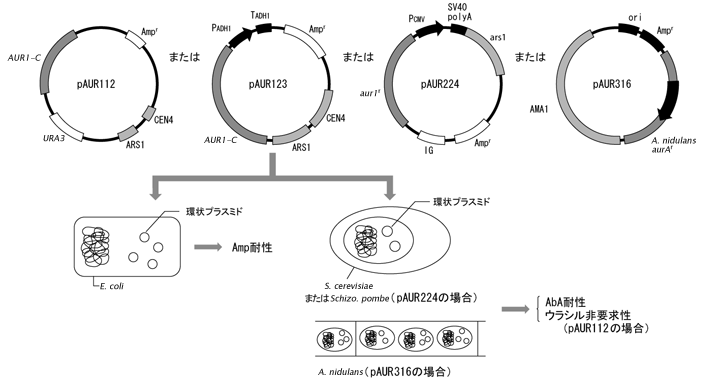
| Saccharomyces cerevisiae* | AUR1-C | pAUR101 | 染色体組込み型ベクター |
| pAUR112 | プラスミド状態で自律複製可能なベクター | ||
| pAUR123 | タンパク発現用プラスミドベクター | ||
| pAUR135 | マーカー除去型プラスミド | ||
| Schizosaccharomyces pombe | aur1r | pAUR224 | Schizo. pombe用発現ベクター |
| Aspergillus nidulans | aurAr | pAUR316 | プラスミド状態で自律複製可能なベクター |
* Aureobasidin A耐性酵母形質転換システムはS. cerevisiae株全般に使用できる形質転換系として開発された系であるが、S. cerevisiaeだけでなく、分類学上S. cerevisiaeの近縁種と考えられるKluyveromyces marxianusやK. lactis、Candida glabrataなどの形質転換系としても有用なシステムである(Application:pAUR101を用いたK. marxianus, K. lactis, およびC. glabrataの形質転換参照)。
Aureobasidin A 耐性形質転換システム取扱説明書
特長
●栄養要求性変異をもたない野生型酵母や実用酵母(酒酵母、パン酵母等)も効率良く形質転換できる。
●選択培地は完全培地(YPD等)にAureobasidin Aを添加するだけであり、煩雑な栄養要求性アミノ酸の添加は必要としないので、大腸菌のアンピシリン添加培地による選択と同様の感覚で形質転換体のスクリーニングが行える。
●S. cerevisiae(pAUR101, pAUR112, pAUR123, pAUR135)、Schizo. pombe(pAUR224)またはA. nidulans(pAUR316)への形質転換効率は栄養要求性選択マーカーを用いた場合と同程度である(酵母:酢酸リチウム法で1~5×104形質転換体/μg DNA;糸状菌:プロトプラスト-PEG法で102~103形質転換体/μg DNA)。
●選択培地は完全培地(YPD等)にAureobasidin Aを添加するだけであり、煩雑な栄養要求性アミノ酸の添加は必要としないので、大腸菌のアンピシリン添加培地による選択と同様の感覚で形質転換体のスクリーニングが行える。
●S. cerevisiae(pAUR101, pAUR112, pAUR123, pAUR135)、Schizo. pombe(pAUR224)またはA. nidulans(pAUR316)への形質転換効率は栄養要求性選択マーカーを用いた場合と同程度である(酵母:酢酸リチウム法で1~5×104形質転換体/μg DNA;糸状菌:プロトプラスト-PEG法で102~103形質転換体/μg DNA)。
Aureobasidin A耐性酵母形質転換システムの原理
Aureobasidin Aは黒色酵母Aureobasidium pullulans No. R106株の産生する抗真菌抗生物質である1)。
S. cerevisiae、Schizo. pombe、C. albicans等のCandida属、一部のAspergillus属に対して強い抗菌活性を持ち、清酒酵母、パン酵母等の実用酵母にも低濃度(0.1~0.4 μg/ml)で殺菌効果を示す。
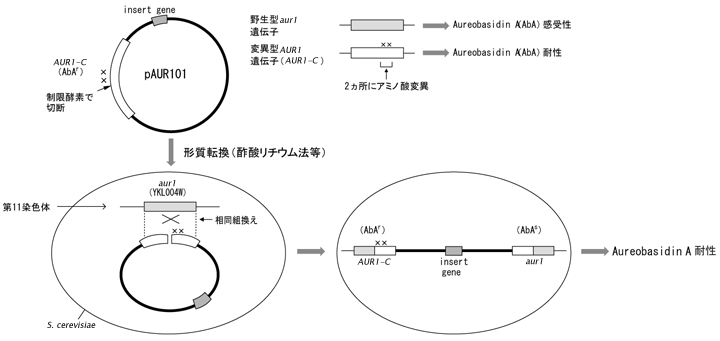
AUR1-C遺伝子は、タカラバイオがS. cerevisiaeより取得したAbA耐性遺伝子で、野生型の対立遺伝子aur1遺伝子(第11染色体YKL004W)の2ヵ所のアミノ酸を遺伝子工学的に置換した変異遺伝子である。この変異導入により、AUR1-C遺伝子はAbAに対して特異的な耐性を獲得する。同様に、aur1 r遺伝子はSchizo. pombeより、aurA r遺伝子はA. nidulansよりそれぞれ取得したAbA耐性遺伝子である。
これらAbA耐性遺伝子は優性的に働くため2)、細胞内に1コピー存在するだけで耐性形質を発現し、また、コピー数に応じてAbA耐性度が上昇する。AbA耐性遺伝子を選択マーカーとして含有するプラスミドで形質転換した場合、形質転換体はAbA耐性クローンとして得られる。
なお、近年、Nagiecら8)によりAureobasidin Aの作用機作が解明されている。それによれば、Aureobasidin Aは真菌のスフィンゴ脂質生合成系の酵素inositolphosphoryl-ceramide(IPC)synthaseの阻害剤であり、aur1遺伝子はIPC synthaseをコードしていると考えられる。
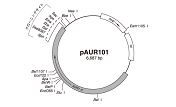
<pAUR101を用いた酵母染色体への組込み>
pAUR101は導入遺伝子を酵母内で安定に保持させるための染色体組込型ベクターで、酵母内で自律的には複製できず、相同組換えにより染色体DNAに組込まれた場合にのみ保持される。AUR1-C遺伝子内の制限酵素サイト(Stu I、BstP I、EcoO65 I、BsiW I)で1ヵ所切断した直鎖状プラスミドで形質転換することにより、第11染色体上のAUR1-C対立遺伝子の位置で効率よく酵母染色体への組込みが起こる。この方法により、染色体上の他の遺伝子に影響を及ぼすことなく外来遺伝子を導入した形質転換体が得られる。pAUR101はE. coliでの選択マーカーとしてアンピシリン耐性遺伝子 Ampr、S. cerevisiaeでの選択マーカーとしてAureobasidin A耐性遺伝子AUR1-Cを保持している。
<pAUR112を用いた形質転換>
pAUR112はS. cerevisiae由来のARS1/CEN4を有するため、分裂安定性があり、S. cerevisiaeの形質転換体中で安定にプラスミド状態(コピー数:1~数個)で保持される。
pAUR112はE. coliでの選択マーカーとしてアンピシリン耐性遺伝子 Ampr、S. cerevisiaeの選択マーカーとしてAureobasidin A耐性遺伝子AUR1-CおよびURA3遺伝子を保持している。
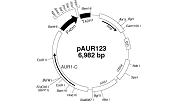
<pAUR123を用いた形質転換>
pAUR123は自律複製型ベクターpAUR112由来のタンパク発現用ベクターである。pAUR112と同様、S. cerevisiaeの形質転換体中で自律複製し、安定にプラスミド状態で保持される。発現用プロモーターとしてS. cerevisiaeのアルコール脱水素酵素遺伝子ADH1のプロモーターを含んでいるので、発現させたい遺伝子を開始コドンATGを含む形でpAUR123のマルチクローニングサイトに挿入すると、形質転換体は構成的に挿入遺伝子を発現する。ただしADH1プロモータは高発現タイプのプロモータではないので、タンパク質の高発現は期待できない。
pAUR123はE. coliでの選択マーカーとしてアンピシリン耐性遺伝子 Ampr、S. cerevisiaeでの選択マーカーとしてAureobasidin A耐性遺伝子AUR1-Cを保持している。
<pAUR135を用いた形質転換>
pAUR135は、ひとつの選択マーカーAUR1-Cで繰り返し形質転換を行うためのマーカー除去型ベクターであり、このベクターを用いて取得したS. cerevisiae形質転換体から簡便な方法で選択マーカーを含むベクター配列が除去されたクローンを選別することができる。
pAUR135には、形質転換体選択マーカーとしてのAUR1-C遺伝子と、マーカー除去株選別用としてのGAL10プロモーターに連結したGIN11M86配列が含まれている。GIN11M86は高発現にすると生育阻害を引き起こす自殺活性を有している。pAUR135による形質転換で得られたAbA耐性の形質転換体をガラクトース培地に移すと、GAL10プロモーター活性が誘導され、GIN11M86が高発現となる。このとき、GIN11M86を含むほとんどの形質転換体が生育阻害を受けるが、低頻度の相同組換えにより生じたAUR1-Cマーカーを含むベクター領域が除去された株のみが優先的に生育してくる。これには、AbA感受性の目的の形質転換体と元に復帰した株とが含まれる。したがって、pAUR135は再び形質転換に使用できる。また、AUR1-Cマーカーだけでなくベクター配列も除かれるため、実用酵母の育種において不用配列が残る不安を軽減できる。pAUR135は、変異導入や遺伝子機能の破壊などに使用できる。
<pAUR224を用いた形質転換>
pAUR224はSchizo. pombe由来のAbA耐性遺伝子であるaur1 r11)を選択マーカーとして含む、Shizo. pombeを宿主とするタンパク発現用ベクターである。発現用プロモーターとしてサイトメガロウイルス(CMV)由来のプロモーターとSV40由来のポリ(A)シグナル配列を含んでいる。
<pAUR316を用いた形質転換>
pAUR316は、Aspergillus nidulans内でプラスミド状態で維持できるAspergillus菌シャトルベクターである。大腸菌での選択マーカーとしてアンピシリン耐性遺伝子Amprを、また、A. nidulansでの選択マーカーとして薬剤耐性遺伝子aurArをもち、自律複製のためのAMA1配列を含んでいる。
<pAUR Primers>
pAUR123およびpAUR224にはそれぞれのベクターのクローニングサイト近傍にアニーリングするプライマーとして、pAUR123 F、同R、およびpAUR224 F、同Rが設定されており、クローニングされたDNA塩基配列のシーケンス確認やFとR Primerを組合せてPCR法によりインサート配列を増幅させることができる。
Aureobasidin A耐性酵母形質転換システムの原理図
| pAUR101 DNAを用いた酵母染色体への組込み |
|---|
| pAUR112, pAUR123, pAUR224, pAUR316 DNAを用いた形質転換 |
|---|
Protocols
酵母Saccharomyces cerevisiae の形質転換(酢酸リチウム法)
簡便な酢酸リチウム法(one-step transformation method)による酵母の形質転換
酢酸リチウム法によるSchizo. pombeの形質転換法
Application
プラスミドpAUR101を用いたADE1遺伝子のgene disruption
pAUR112による実用酵母の形質転換効率
pAUR101を用いたKluyveromyces marxianus、K. lactis、およびCandida glabrataの形質転換
pAUR123ベクターを用いたlacZ遺伝子の発現(Candida glabrataを宿主として)
pAUR224ベクターを用いたlacZ遺伝子の発現
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。