【ユーザー様実施例】ThruPLEX® DNA-Seq Kitを使用したネッタイツメガエル初期胚で発現する発生制御因子に対するChIP-Seq解析手法の確立
データご提供:理化学研究所生命医科学研究センター 応用ゲノム解析技術研究チーム 安岡有理様
■実験の背景・目的
ChIP-Seqとは、クロマチン共免疫沈降(ChIP)によって目的タンパク質が結合しているゲノムDNA領域を濃縮し、その領域を次世代シーケンサー(NGS)で解読することで網羅的に決定する手法である。
動物の胚発生において、細胞の運命決定を担う転写因子と呼ばれる一連のDNA結合タンパク質が、どのようにゲノムDNAに作用して細胞の発生運命を制御しているのかを解き明かす上で、動物胚を用いたChIP-Seq解析は非常に重要な意味を持つ。
しかし、動物胚の中でも一部の細胞にのみ発現する発生制御因子(転写因子)を標的としたChIP-Seq解析を実施するためには、限られた発現細胞からChIPによって濃縮されたごく微量のDNAを用いてNGS解析を行う必要がある。
我々が研究に用いているネッタイツメガエルは、同調的に発生する胚を一度に多量に入手できる優れた実験動物であり、特に初期発生における遺伝子発現制御ネットワークの解析に適している。
そこで我々は、少量のネッタイツメガエル胚からChIPによって濃縮された微量DNAをタカラバイオ社のThruPLEX DNA-Seq KitによってNGSライブラリー化することで、効率よく発生制御因子のChIP-Seq解析を行う手法を確立した。
■使用製品
| 製品名 | 容量 | 製品コード |
|---|---|---|
| SMARTer® ThruPLEX® DNA-Seq Kit | 24回 | R400674 |
■実験ワークフロー
①胚の固定
ネッタイツメガエル原腸胚約250個を1%ホルマリンで固定し、-80℃で冷凍保存↓
②細胞溶解と核の濃縮
界面活性剤入りのLysis bufferで固定胚をhomogenizeし、核を濃縮↓
③クロマチン断片化
超音波破砕装置Picoruptor(Diagenode社)でクロマチンを断片化↓
④免疫沈降
あらかじめanti-Otx2抗体を結合させたProtein A Magnetic Beadsと上記クロマチン画分を混合し、4℃で一晩転倒撹拌後、RIPA Bufferでビーズを洗浄↓
⑤脱クロスリンク
溶出Buffer(1% SDS含)で65℃一晩インキュベート後、RNase A処理、Proteinase K処理により脱クロスリンク ※ビーズが含まれた状態で実施↓
⑥DNA精製
MinElute PCR Purification Kit(QIAGEN社)でDNA精製 ※溶出液は2倍に薄めて10 μl×2回で溶出↓
⑦ChIP DNAのQC
蛍光光度計(Qubit dsDNA HS kit)を用いてDNAを定量qPCRによって既知のゲノム領域の濃縮を確認
↓
⑧NGS用ライブラリー調製
10 μlのChIP DNAを用いてThruPLEX DNA-Seq Kitでライブラリー調製↓
⑨シーケンス
NextSeq550(基礎生物学研究所、新学術領域「進化制約方向性」大規模解析支援班 http://constrained-evo.org/support.html)75 bpペアエンド↓
⑩情報解析
ネッタイツメガエルゲノム(v9)にBWAを用いてマッピングし、IGVで可視化■実験データ
・マッピング結果
・Otx2 ChIP-seq結果の可視化例
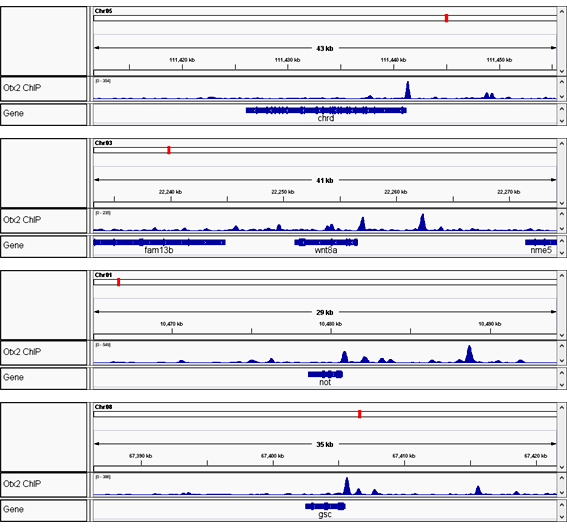
1 ng程度のChIP DNAから十分量のシーケンスライブラリーが作成できた(PCRサイクル=12)。
マッピングデータを見る限り、以前約2,000個のネッタイツメガエル原腸胚を用いて行ったChIP-Seq解析(Yasuoka et al., Nature Communications 2014, 5:4322)と同等の結果が得られている。
| サンプル | total reads | mapped reads(%) |
|---|---|---|
| Input | 74,426,066 | 70,219,181(94.35%) |
| Otx2 ChIP | 82,794,598 | 75,710,767(91.44%) |
・Otx2 ChIP-seq結果の可視化例

1 ng程度のChIP DNAから十分量のシーケンスライブラリーが作成できた(PCRサイクル=12)。
マッピングデータを見る限り、以前約2,000個のネッタイツメガエル原腸胚を用いて行ったChIP-Seq解析(Yasuoka et al., Nature Communications 2014, 5:4322)と同等の結果が得られている。
■今回の実験結果を受けての今後のご計画・展望
ThruPLEX DNA-Seq Kitを用いることで微量DNAからのライブラリー作製が可能となったので、同一転写因子に対するChIP-Seq解析を繰り返し行い、転写因子がどの程度のばらつき(揺らぎ)を持って結合しているのかを今後明らかにしたい(新学術領域「進化制約方向性」公募研究班 http://constrained-evo.org/proposed_projects.html)。
標的とする転写因子や使用する抗体にもよるが、ChIPに用いる胚の数を50~100個ほどに減らせる可能性もあり、今後さらなる条件検討によってより効率よく研究を進められると期待できる。
標的とする転写因子や使用する抗体にもよるが、ChIPに用いる胚の数を50~100個ほどに減らせる可能性もあり、今後さらなる条件検討によってより効率よく研究を進められると期待できる。
■ThruPLEXを使ってChIP解析をしたことがない方へのコメント
ThruPLEX DNA-Seq Kitは蛍光光度計でも測定できないほどの微量DNAからライブラリー作製が可能なので、少数細胞のChIP-Seq解析を行う方にはオススメです。
実験の工程が少なく簡便で、キット内の試薬のチューブラベルも色分けされていてわかりやすく、とても使いやすい仕様になっているので、ライブラリー作製に慣れていない方にもオススメです。
実験の工程が少なく簡便で、キット内の試薬のチューブラベルも色分けされていてわかりやすく、とても使いやすい仕様になっているので、ライブラリー作製に慣れていない方にもオススメです。
