AAVベクターゲノムのNGS解析(ロングリード解析)
遺伝子治療製品として注目されるアデノ随伴ウイルス(AAV)ベクターに対して、Sequel IIロングリードシーケンサーを用いた解析を行い、ベクターゲノム構造の確認およびベクターゲノム領域内の変異を確認することで、AAVベクターの品質確認におけるNGSロングリード解析の有効性を評価しました。
実験方法:
<ZsGreen1発現AAVベクターの作製> AAVpro Helper Free System(製品コード 6673、6230、6650、6651)、pAAV-ZsGreen1 Vector(製品コード 6231)を用い、AAV1の血清型のCapsidを持つAAV-ZsGreen1ウイルスベクターを作製した。作製したウイルスは、弊社の研究受託の標準手順であるCsCl超遠心法により精製を行い、Amiconにより濃縮した。
<ウイルスゲノムDNAの抽出とNGS解析>
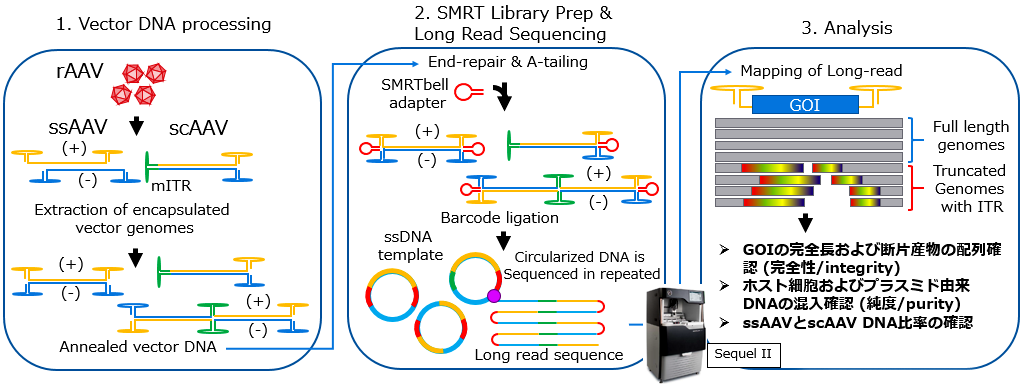
ロングリードシーケンサーを用いたAAVベクターゲノム構造の確認手法の概略図
結果:
<AAVベクターの完全長および断片化産物の配列確認>
NGSの全シーケンスリード配列をpAAV-ZsGreen1ベクタープラスミドにマッピングを実施した結果、シーケンスリード配列の96%以上がITR間のAAVベクター領域にマップされ、88%以上はITRを含む完全長のAAVベクターのゲノム配列を持つことが確認できた。
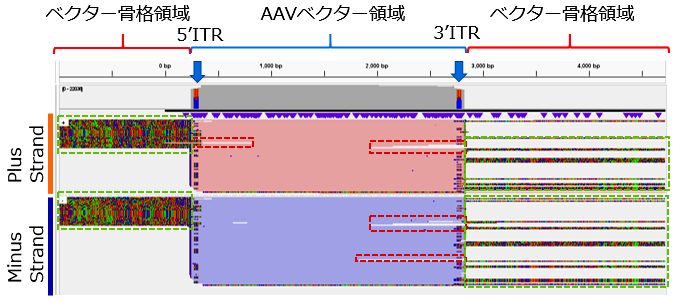
AAV_QC_AAV1のIGV図
<全シーケンスリードの分類結果>
NGSの全シーケンスリード配列について分類した結果(表1)、AAVベクター由来のリードは全体の96%以上を占め、さらに約2/3は一本鎖DNAゲノム構造をとるsingle-strand AAV(ssAAV)、約1/3はヘアピンDNAゲノム構造をとるself-complementary AAV(scAAV)に分類された。AAVベクター以外のリードは3%以下であり、ヒトゲノム、pRC、pHelperプラスミド由来のシーケンスリード配列やその複合産物等に分類された。またAAVベクター由来のリードのうち88%以上は完全長を含んでおり、一部欠損のあるリードは12%以下であった(表2)。これらの結果より、弊社のCsCl超遠心法で精製されたAAVの純度は、非常に高いことが示された。
表1. AAVベクターシーケンスリード配列の分類結果1
表2. AAVベクターシーケンスリード配列の分類結果2
実験方法:
<ZsGreen1発現AAVベクターの作製> AAVpro Helper Free System(製品コード 6673、6230、6650、6651)、pAAV-ZsGreen1 Vector(製品コード 6231)を用い、AAV1の血清型のCapsidを持つAAV-ZsGreen1ウイルスベクターを作製した。作製したウイルスは、弊社の研究受託の標準手順であるCsCl超遠心法により精製を行い、Amiconにより濃縮した。
<ウイルスゲノムDNAの抽出とNGS解析>
- NucleoSpin Virusを用いて、AAVベクターDNAを抽出精製し、アニーリングにより二本鎖を形成させた。
- ロングリードライブラリーを構築し、PacBio社のSequel IIロングリードシーケンサーを用いてHiFi Read(CCS:Circular Consensus Sequencing)解析を行った。
- 得られた全てのリードをベクタープラスミド、AAVベクター産生に用いたヘルパープラスミドおよびホストゲノムの参照配列にマッピングし、マッピングの結果よりAAVベクターのDNA配列確認、およびシーケンスリードの分類によるAAVベクター以外のDNA配列の確認を行った。

ロングリードシーケンサーを用いたAAVベクターゲノム構造の確認手法の概略図
結果:
<AAVベクターの完全長および断片化産物の配列確認>
NGSの全シーケンスリード配列をpAAV-ZsGreen1ベクタープラスミドにマッピングを実施した結果、シーケンスリード配列の96%以上がITR間のAAVベクター領域にマップされ、88%以上はITRを含む完全長のAAVベクターのゲノム配列を持つことが確認できた。

AAV_QC_AAV1のIGV図
- 88%は完全長のAAV構造
- 赤枠 一部欠損したものも含む(12%未満)
- 緑枠 secondary alignmentもAAVベクター領域にマップされるリードはscAAV由来と思われる(1/3程度)
- 青矢印
 ITR配列に高頻度に生じるミスマッチは、ITR配列の逆位構造で説明可(Flip/Flop)
ITR配列に高頻度に生じるミスマッチは、ITR配列の逆位構造で説明可(Flip/Flop)
<全シーケンスリードの分類結果>
NGSの全シーケンスリード配列について分類した結果(表1)、AAVベクター由来のリードは全体の96%以上を占め、さらに約2/3は一本鎖DNAゲノム構造をとるsingle-strand AAV(ssAAV)、約1/3はヘアピンDNAゲノム構造をとるself-complementary AAV(scAAV)に分類された。AAVベクター以外のリードは3%以下であり、ヒトゲノム、pRC、pHelperプラスミド由来のシーケンスリード配列やその複合産物等に分類された。またAAVベクター由来のリードのうち88%以上は完全長を含んでおり、一部欠損のあるリードは12%以下であった(表2)。これらの結果より、弊社のCsCl超遠心法で精製されたAAVの純度は、非常に高いことが示された。
表1. AAVベクターシーケンスリード配列の分類結果1
| Assign Type | Count | Frequency (%) | Assign Subtype | Frequency (%) |
|---|---|---|---|---|
| AAV | 16,478 | 97.91 | ssAAV(一本鎖AAV) | 61.5 |
| scAAV(自己相補的AAV) | 36.41 | |||
| Other(その他) | 352 | 2.09 | other(未分類) | 1.6 |
| repcap(pRCプラスミド) | 0.26 | |||
| chimeric(複合) | 0.17 | |||
| host(ヒトゲノム) | 0.05 | |||
| helper(pHelperプラスミド) | 0.02 | |||
| Total | 16,830 | 100 | Total | 100 |
| Assign Type | Count | Frequency (%) |
|---|---|---|
| full(完全長) | 14,519 | 88.11 |
| partial(一部欠損) | 1,959 | 11.89 |
| 16,478 | 100 |
