10 kb以上の長鎖マルチプレックスPCRを用いたロングリードNGS解析
マルチプレックスPCRは、複数のプライマー対を一つのPCR反応系に使用することで、複数の遺伝子領域を同時に増幅する方法です。マルチプレックスPCRを行うことで、試薬や消耗品の節約によるコスト削減や、同時検出による迅速性のメリットに加え、貴重なサンプルの有効利用が可能です。しかし、マルチプレックスPCRは非特異的な増幅が生じやすく、プライマー設計の複雑さや反応の最適化が必要といったデメリットがあります。
今回、世界最高クラスの高い特異性を持つPCR酵素であるPrimeSTAR LongSeq DNA Polymerase(製品コード R055S/A/B)を用い、長鎖マルチプレックスPCRが可能か評価を行いました(実施例1)。
また、PrimeSTAR LongSeq DNA Polymeraseは高い正確性を特長とするDNA Polymeraseであり、正確性の高い増幅が求められる次世代シーケンサー(NGS)解析に適しています。そこで、長鎖マルチプレックスPCRで得られた増幅産物についてロングリードシーケンス(Oxford Nanopore Technologies)を用いた解析を行いました(実施例2)。

図1.解析ワークフロー
今回、世界最高クラスの高い特異性を持つPCR酵素であるPrimeSTAR LongSeq DNA Polymerase(製品コード R055S/A/B)を用い、長鎖マルチプレックスPCRが可能か評価を行いました(実施例1)。
また、PrimeSTAR LongSeq DNA Polymeraseは高い正確性を特長とするDNA Polymeraseであり、正確性の高い増幅が求められる次世代シーケンサー(NGS)解析に適しています。そこで、長鎖マルチプレックスPCRで得られた増幅産物についてロングリードシーケンス(Oxford Nanopore Technologies)を用いた解析を行いました(実施例2)。

図1.解析ワークフロー
実施例1:20ターゲットの長鎖マルチプレックスPCR
●方法
使用酵素:
鋳型:
ヒトゲノムDNA
ターゲット:
全20ターゲット(鎖長: 10~12 kb, GC含量: 35~60%)
反応系:
50 μl反応系に鋳型とする100 ngのヒトゲノムDNAを添加し、各プライマーは最終濃度0.2 μMで使用
PCR条件:
PrimeSTAR LongSeq DNA Polymerase
※他社PCR酵素:メーカー推奨PCR条件
※PCR反応はN=2で実施
使用装置:
Clontech PCR Thermal Cycler GP(製品コード WN400)
●結果
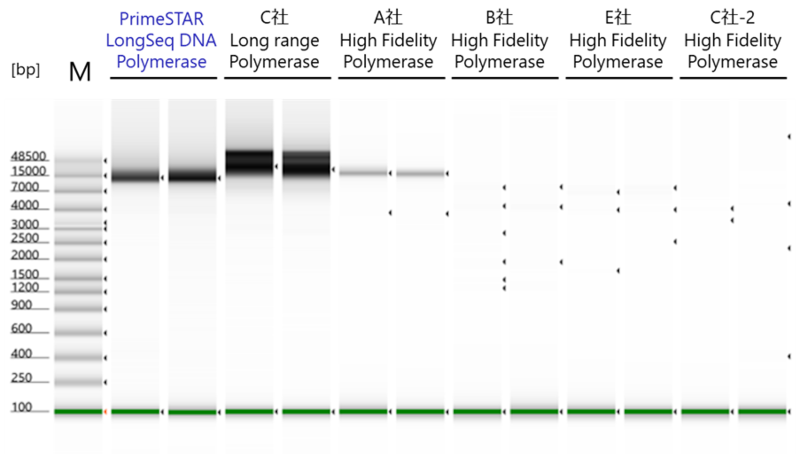
図2.20ターゲットのマルチプレックスPCR
マルチプレックスPCR産物を精製後、4200 TapeStation System(Agilent Technologies)を用いて解析しました。PrimeSTAR LongSeq DNA Polymeraseの増幅産物は、目的の増幅サイズである10~12 kb付近に特異的な増幅が確認できました。
使用酵素:
- PrimeSTAR LongSeq DNA Polymerase
- 長鎖増幅を得意としている他社PCR酵素
鋳型:
ヒトゲノムDNA
ターゲット:
全20ターゲット(鎖長: 10~12 kb, GC含量: 35~60%)
反応系:
50 μl反応系に鋳型とする100 ngのヒトゲノムDNAを添加し、各プライマーは最終濃度0.2 μMで使用
PCR条件:
PrimeSTAR LongSeq DNA Polymerase
| 94℃ | 1 | min. | ||
| ↓ | ||||
| 98℃ | 10 | sec. | 30 cycles | |
| 68℃ | 30 | sec./kb | ||
※他社PCR酵素:メーカー推奨PCR条件
※PCR反応はN=2で実施
使用装置:
Clontech PCR Thermal Cycler GP(製品コード WN400)
●結果

図2.20ターゲットのマルチプレックスPCR
マルチプレックスPCR産物を精製後、4200 TapeStation System(Agilent Technologies)を用いて解析しました。PrimeSTAR LongSeq DNA Polymeraseの増幅産物は、目的の増幅サイズである10~12 kb付近に特異的な増幅が確認できました。
実施例2:NGS解析
●方法
実施例1で10~12 kb付近の増幅バンドが確認できた3つの酵素のサンプルについて、Native Barcoding Kit 96 V14(Oxford Nanopore Technologies)を用いてNGSライブラリーを作製し、GridION (Oxford Nanopore Technologies)、R10.4.1 flow cells (Oxford Nanopore Technologies) を用たNGS解析を行いました。
ロングリードNGSから得られた塩基配列はリード数を揃えて解析を行いました。また、本実験ではプライマー濃度の最適化を行わず、等モル混合したプライマーセットを使用しています。
●結果
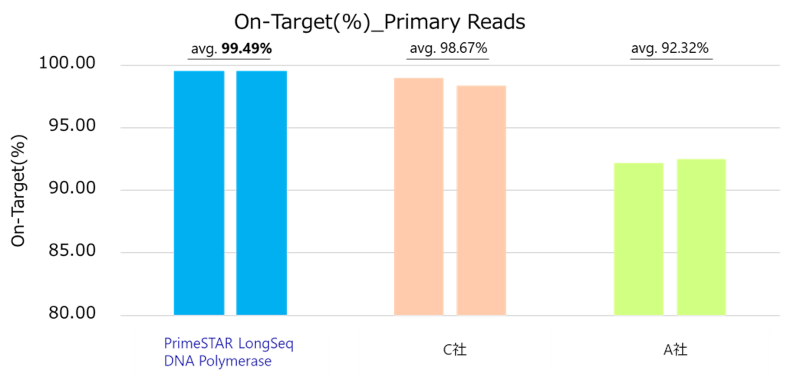
図3.各酵素によるOn-Target率
得られたシーケンスリードから、各酵素によるOn-Target率を算出しました。PrimeSTAR LongSeq DNA Polymeraseの増幅産物は、On-Target率99%以上を示し、長鎖マルチプレックスPCRにおいて特異性の高い増幅ができていることが示唆されました。
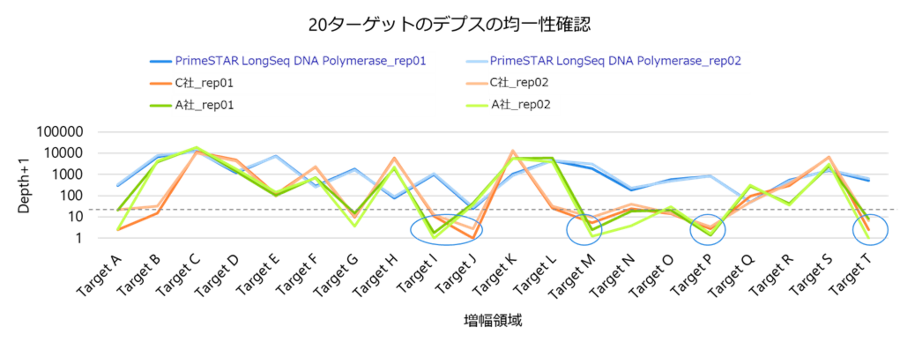
図4.20ターゲットのデプスの均一性確認
マルチプレックスPCRでは各PCRターゲットの増幅効率の違いが課題となります。各PCR酵素の増幅産物中に、20ターゲットの配列が含まれているかを確認しました。PrimeSTAR LongSeq DNA Polymeraseを用いることでより均一な増幅を得ることができました。他社PCR酵素では複数のターゲットでまったくデプスが得られていない、もしくは極端に少ないデプスが得られたのに対し、PrimeSTAR LongSeq DNA Polymeraseではよりバイアスの少ない均一なデプスを得ることができました。
増幅が困難な領域についてはシーケンス量を増やしても改善は難しく、シーケンスコストが増大します。PrimeSTAR LongSeq DNA Polymeraseを用いることでマルチプレックス反応PCRの最適化作業を軽減でき、より少ないシーケンス量での解析可能となることが期待できます。
(A)

(B)
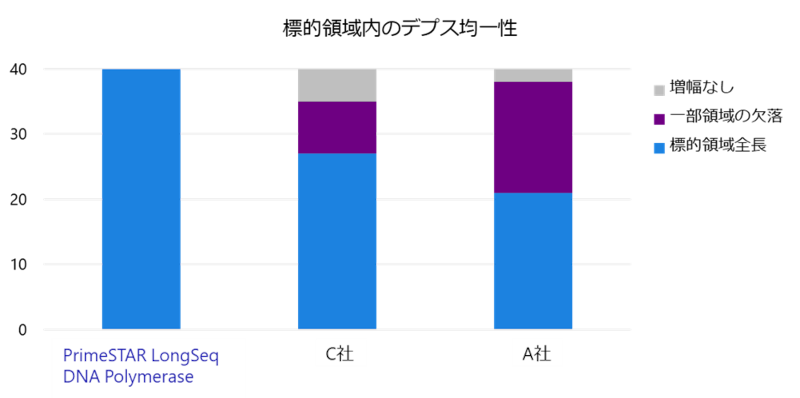
図5.ターゲット領域全長の増幅確認
各酵素によるマルチプレックスPCR増幅産物中に、20ターゲットの配列が全長含まれているかをIntegrative Genomics Viewer(IGV)を用いて確認しました。PrimeSTAR LongSeq DNA Polymeraseは20ターゲットすべてで全長増幅ができましたが、他社のPCR酵素では一部領域が欠落し、68~78%のターゲットでしか全長配列を取得できませんでした。
※Target Lのリードが得られていない領域は、検証に使用したヒトゲノムDNAに存在する既知の大きな欠失領域(976 bp)に該当します。
実施例1で10~12 kb付近の増幅バンドが確認できた3つの酵素のサンプルについて、Native Barcoding Kit 96 V14(Oxford Nanopore Technologies)を用いてNGSライブラリーを作製し、GridION (Oxford Nanopore Technologies)、R10.4.1 flow cells (Oxford Nanopore Technologies) を用たNGS解析を行いました。
ロングリードNGSから得られた塩基配列はリード数を揃えて解析を行いました。また、本実験ではプライマー濃度の最適化を行わず、等モル混合したプライマーセットを使用しています。
●結果

図3.各酵素によるOn-Target率
得られたシーケンスリードから、各酵素によるOn-Target率を算出しました。PrimeSTAR LongSeq DNA Polymeraseの増幅産物は、On-Target率99%以上を示し、長鎖マルチプレックスPCRにおいて特異性の高い増幅ができていることが示唆されました。

図4.20ターゲットのデプスの均一性確認
マルチプレックスPCRでは各PCRターゲットの増幅効率の違いが課題となります。各PCR酵素の増幅産物中に、20ターゲットの配列が含まれているかを確認しました。PrimeSTAR LongSeq DNA Polymeraseを用いることでより均一な増幅を得ることができました。他社PCR酵素では複数のターゲットでまったくデプスが得られていない、もしくは極端に少ないデプスが得られたのに対し、PrimeSTAR LongSeq DNA Polymeraseではよりバイアスの少ない均一なデプスを得ることができました。
増幅が困難な領域についてはシーケンス量を増やしても改善は難しく、シーケンスコストが増大します。PrimeSTAR LongSeq DNA Polymeraseを用いることでマルチプレックス反応PCRの最適化作業を軽減でき、より少ないシーケンス量での解析可能となることが期待できます。
(A)

(B)

図5.ターゲット領域全長の増幅確認
各酵素によるマルチプレックスPCR増幅産物中に、20ターゲットの配列が全長含まれているかをIntegrative Genomics Viewer(IGV)を用いて確認しました。PrimeSTAR LongSeq DNA Polymeraseは20ターゲットすべてで全長増幅ができましたが、他社のPCR酵素では一部領域が欠落し、68~78%のターゲットでしか全長配列を取得できませんでした。
※Target Lのリードが得られていない領域は、検証に使用したヒトゲノムDNAに存在する既知の大きな欠失領域(976 bp)に該当します。
●まとめ
本検証では、6つのPCR酵素を評価し、3つのPCR酵素で20ターゲットのマルチプレックスPCRにより特異的な増幅バンドが確認できました。その3つのPCR酵素のうち、20ターゲットすべての領域の全長配列を得られたPCR酵素は、PrimeSTAR LongSeq DNA Polymeraseのみでした。長鎖マルチプレックスPCRによりターゲット領域の全長をNGS解析するには、PCR酵素の選択が重要であることが示唆されました。
