~Cell-free DNA~ 他社NGSライブラリー作製キットとの比較
Cell-free DNAは細胞の死滅や細胞からの分泌等によって過度に断片化されたDNAであるため、シーケンスライブラリー作製が難しいサンプルの一つです。ヒト血漿から抽出したCell-free DNA 10 ngを使用して各キットのプロトコールに従い、ライブラリーを作製しその性能を比較評価しました。NextSeq 2000(Illumina)でペアエンドシーケンス(2×150 bp)を行い、リード数は合計100万リードにダウンサンプリングした後に、Bowtie2とPicardを用いて解析を実施しました。アライメントした参照配列はGRCh38を使用しました。ssDNA-Seq Kit(製品コード NN0003/NN0004)はCell-free DNAのサンプルにおいて高いマッピング率(%Mapping)と低い重複率(%Duplicate)を達成し、他社キットによりも優れたシーケンス結果およびライブラリー収量を示しました。
表1.ssDNA-Seq Kitの優れたシーケンス結果
Mapping(%): 参照ゲノム配列にアライメントされたリードの割合を示す。アダプターダイマー(インサートDNAをもたず、アダプター配列同士が結合)などが多く含まれるライブラリーをシーケンスした場合、この数値が低下する。
Duplicate(%) : 同じ配列のDNAフラグメントが複数回読み取られたリードの割合を示す。PCR増幅などにより、特定の分子が繰り返し読まれているものが多いとこの数値が高くなる。ヘテロ変異などを高感度に検出するためには、この数値が低いほど望ましい。

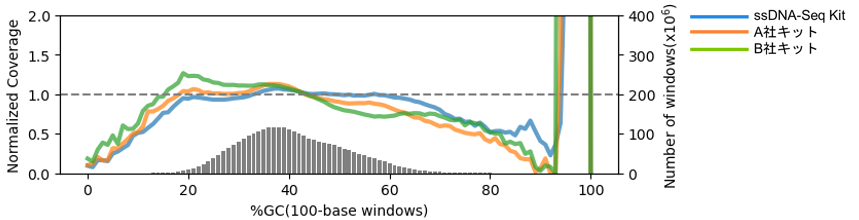 図1.Cell-free DNA由来ライブラリーのシーケンス解析におけるGCカバレッジ
図1.Cell-free DNA由来ライブラリーのシーケンス解析におけるGCカバレッジ
ヒト血漿(BioIVT)から抽出したCell-free DNA 10 ngを使用して各キットのプロトコールに従い、ライブラリーを調製した。NextSeq 2000(Illumina)でペアエンドシーケンス(2×150 bp)を行い、リード数は合計400万リードにダウンサンプリングした後に、Picardを用いて解析を実施した。縦の灰色のバーは、100 bp windowsを用いて予想されるGC含量分布を表している。ssDNA-Seq KitはCell-free DNAにおいて、GC含有量に依らず最も均一なカバレッジを示した。
表1.ssDNA-Seq Kitの優れたシーケンス結果
| Library yield(ng) | Mapping(%) | Duplicate(%) | |
| ssDNA-Seq Kit | 468 | 98.8 | 0.92 |
| A社キット | 40 | 96.2 | 2.39 |
| B社キット | 82 | 94.6 | 1.69 |
Duplicate(%) : 同じ配列のDNAフラグメントが複数回読み取られたリードの割合を示す。PCR増幅などにより、特定の分子が繰り返し読まれているものが多いとこの数値が高くなる。ヘテロ変異などを高感度に検出するためには、この数値が低いほど望ましい。

ヒト血漿(BioIVT)から抽出したCell-free DNA 10 ngを使用して各キットのプロトコールに従い、ライブラリーを調製した。NextSeq 2000(Illumina)でペアエンドシーケンス(2×150 bp)を行い、リード数は合計400万リードにダウンサンプリングした後に、Picardを用いて解析を実施した。縦の灰色のバーは、100 bp windowsを用いて予想されるGC含量分布を表している。ssDNA-Seq KitはCell-free DNAにおいて、GC含有量に依らず最も均一なカバレッジを示した。
Cell-free DNA の解析には高品質なシーケンス結果を得られるssDNA-Seq Kitが最適です。
