※色文字での表示は、キャンペーン価格およびそのキャンペーン期間です。
※本キットにはIndexが含まれていません。Unique Dual Index Kit(製品コード 634752/634753/634754/634755/634756)を別途ご購入ください。
※本キットにはIndexが含まれていません。Unique Dual Index Kit(製品コード 634752/634753/634754/634755/634756)を別途ご購入ください。
カートにいれる
WEB会員ログインが必要です
製品説明
ssDNA-Seq Kitは、一本鎖DNA(single strand DNA:ssDNA)、および二本鎖DNA(double strand DNA:dsDNA)からイルミナ社の次世代シーケンサー(NGS)ライブラリーを調製する製品です。
本製品は、ssDNA同士を結合できるSingle strand DNA Ligase(SDL)を用いたNGSライブラリー調製法を採用しています。サンプルに含まれるすべてのDNAを熱変性によりssDNAとし、それらのssDNAはSDLによりNGSライブラリー調製に用いられます。
従来のdsDNAのNGSライブラリー調製で失われていたssDNAや損傷分解DNAにも対応可能で、以下のような様々なssDNAおよびdsDNAからのライブラリー調製と解析に最適です。高効率なssDNAライゲーション技術により、超微量DNA(10 pg~)からの解析が可能であり、Ultra-short ssDNA(50 base)のNGS解析も可能です。
本製品は、ssDNA同士を結合できるSingle strand DNA Ligase(SDL)を用いたNGSライブラリー調製法を採用しています。サンプルに含まれるすべてのDNAを熱変性によりssDNAとし、それらのssDNAはSDLによりNGSライブラリー調製に用いられます。
従来のdsDNAのNGSライブラリー調製で失われていたssDNAや損傷分解DNAにも対応可能で、以下のような様々なssDNAおよびdsDNAからのライブラリー調製と解析に最適です。高効率なssDNAライゲーション技術により、超微量DNA(10 pg~)からの解析が可能であり、Ultra-short ssDNA(50 base)のNGS解析も可能です。
- ssDNA
- ゲノムDNA
- 環境DNA
- ウイルスゲノム
- dsDNA
- FFPE由来DNA
- 古代DNA
- 合成オリゴ
- ssDNAとdsDNAの混合物
- cell-free DNA
- Ultra-short ssDNA
- DNAアプタマー
表1.ssDNA-Seq Kitの対象サンプル一覧

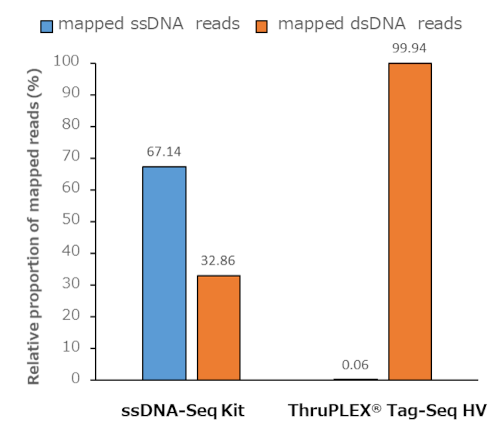
図1.ssDNA-Seq KitはssDNAとdsDNAからライブラリー調製が可能
ssDNA合成オリゴ200 baseとdsDNA合成オリゴ200 bpの混合産物を使用してDNAライブラリーをssDNA-Seq KitとThruPLEX Tag-Seq HV(製品コード R400743)で調製した。NextSeq 2000でペアエンドシーケンス(2×150 bp)を行った。解析はBowtie2を使用し、それぞれの既知配列にアライメントした。
ThruPLEX Tag-Seq HVはdsDNAからライブラリー調製が可能であるのに対し、ssDNA-Seq KitはssDNAとdsDNA両方からライブラリー調製が可能であることが示された。
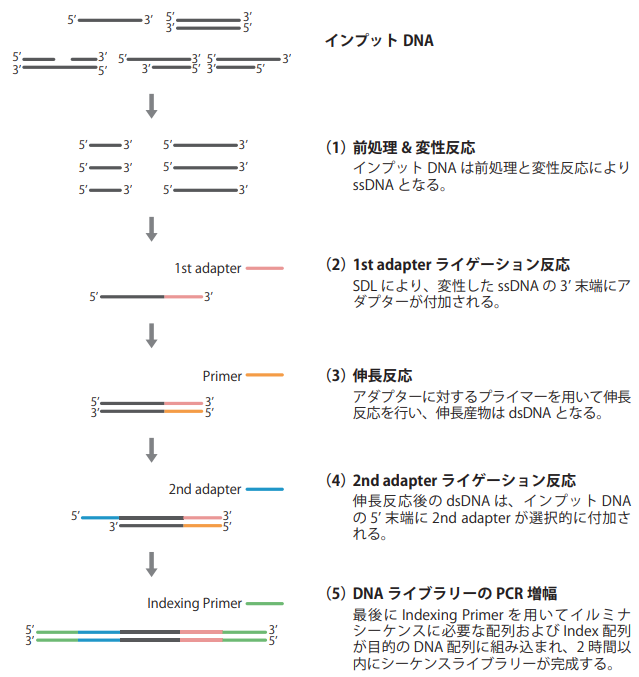
図2.ライブラリー調製フローの概略図
本製品は、ssDNAに直接アダプターをライゲースする技術により、断片化の進んだDNAであっても安定したライブラリー収量を得ることが可能である。
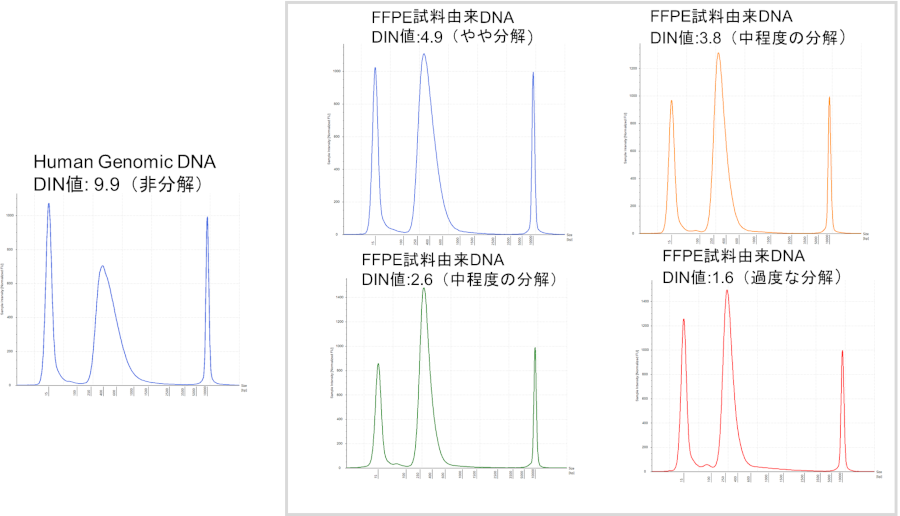
図3.断片化度合いの異なるヒトゲノムから調製したDNAライブラリーの波形
Human Genomic DNA、および断片化スコアが異なるFFPE試料由来DNAから、本製品を用いてDNAライブラリーを調製した。超音波発生装置(Covaris)で断片化処理されたHuman Genomic DNAとFFPE試料由来DNAをそれぞれ10 ng使用した。調製したライブラリーをTapeStation HD5000(Agilent Technologies)でサイズを評価したところ、断片化度合いの異なるいずれのサンプルにおいても良好なDNAライブラリーの波形が確認された。NextSeq 2000でペアエンドシーケンス(2×150 bp)を行い、Bowtie2を使用してGRCh38にアライメントしたところ、いずれの断片化度合いのサンプルからも97%以上のマッピングが確認された。
DIN値はTapeStation Genomic DNA(Agilent Technologies)により算出される断片化を示す値であり、DIN値の低いDNAサンプルは断片化の進んでいることを示す。各サンプルの断片化度合いをTakara FFPE DNA QC All-in-One Kit(製品コード NN0001)を用いてLong/Short比で評価した結果は以下の通りであり、L/S比が0.02と過度に損傷したDNAからでもライブラリー調製が可能であることが確認された。
- DIN: 9.9=L/S: 0.95
- DIN: 4.9=L/S: 0.30
- DIN: 3.8=L/S: 0.10
- DIN: 2.6=L/S: 0.04
- DIN: 1.6=L/S: 0.02
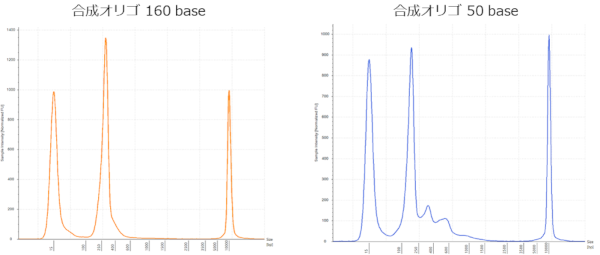
図4.合成オリゴのDNAライブラリーの波形
ssDNA合成オリゴ160 base 10 ngおよび合成オリゴ50 base 100 ngから、本製品を用いてDNAライブラリーを調製した。TapeStation(Agilent)でDNAライブラリーのサイズを評価し、各サンプルにおいて良好な波形が確認された。合成オリゴ50 baseの高分子側のピークはバブル構造によると考えられる。
NextSeq 2000でペアエンドシーケンス(2×150 bp)を行い、BWA-MEMを使用して160 baseおよび50 baseの既知配列にアライメントした。合成オリゴ160 baseでは97%以上のマッピングが確認された。一方で、合成オリゴ50 baseでは89%以上のマッピングが確認された。
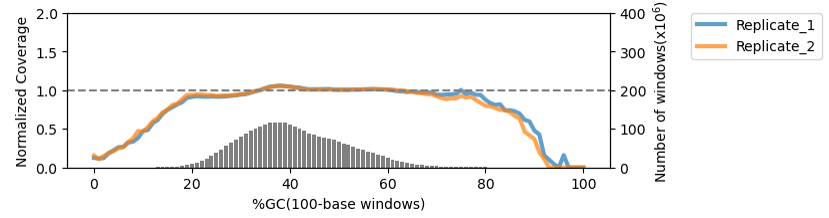
図5.カバレッジの均一性
超音波発生装置(Covaris)で断片化されたHuman Genomic DNA 10 ng (n=2)から、本製品を用いてDNAライブラリーを調製した。NextSeq 2000でペアエンドシーケンス(2×150 bp)を行い、リード数はtotal 900万リードにダウンサンプリングした。縦の灰色のバーは、100 bp windowsを用いて予想されるGC含量分布を表している。この結果から、本製品はGC含有量に依らず均一なカバレッジを示すことが確認された。
表2.ssDNA-Seq Kitは断片化の進んだDNAに最適
Human Genomic DNA 10 ng、断片化の程度が異なるFFPE試料由来DNA 10 ngおよび血漿から抽出したcell-free DNA 10 ngから、本製品を用いてDNAライブラリーを調製した。NextSeq 2000でペアエンドシーケンス(2×150 bp)を行い、リード数はtotal 100万リードにダウンサンプリングした。解析はBowtie2とPicardを使用した。
断片化の進んでいないDNA(Human Genomic DNA)と断片化の進んだDNA(FFPE試料とCell-free DNA)はいずれも97%以上の高いマッピング率を示し、高品質なシーケンス結果を示した。
| 試料名 | Total reads aligned | Mapping(%) | Duplicate(%) | Chimera(%) | Library yield (ng) |
| Human Genomic DNA | 9.79×105 | 98.59 | 0.77 | 0.79 | 1,088 |
| FFPE試料由来DNA (DIN:4.9) | 9.78×105 | 98.39 | 0.95 | 0.85 | 560 |
| FFPE試料由来DNA (DIN:3.8) | 9.75×105 | 98.18 | 0.88 | 1.10 | 395 |
| FFPE試料由来DNA (DIN:2.6) | 9.76×105 | 98.22 | 0.94 | 1.21 | 394 |
| FFPE試料由来DNA (DIN:1.6) | 9.62×105 | 97.14 | 0.95 | 1.25 | 101 |
| Cell-free DNA | 9.82×105 | 98.82 | 0.92 | 1.61 | 468 |
Mapping(%)は参照ゲノム配列にアラインメントされたリードの割合を示す。
Duplicate(%)は、PCRによるライブラリー調製の際に生じる、同じ配列のDNAフラグメントを由来とするリードの割合を示す。
Chimera(%)は、ゲノムの二つの異なる部位にアライメントされたリードの割合を示す。
内容
| NN0003 24反応 | NN0004 96反応 | |
1 Preparation enzyme | 24 μl | 96 μl |
2 Preparation buffer | 48 μl | 192 μl |
(*) TE buffer | 1 ml×3 | 6 ml×2 |
3 SDL enzyme | 48 μl | 192 μl |
4 SDL buffer | 600 μl | 1.2 ml×2 |
5 SDL adapter | 24 μl | 96 μl |
6 Extension polymerase Premix | 600 μl×2 | 1.2 ml×4 |
7 Extension primer | 24 μl | 96 μl |
8 VDC enzyme | 24 μl | 96 μl |
9 VDC buffer | 150 μl | 600 μl |
10 PCR polymerase Premix | 600 μl | 1.2 ml×2 |
NN0003:(白色)
NN0004:(コンポーネントカラー無し)
保存
-20℃
本製品以外に必要な試薬、機器(主なもの)
- Unique Dual Index Kit(製品コード 634752/634753/634754/634755/634756)
- NucleoMag NGS Clean-up and Size Select(製品コード 744970.5/744970.50/744970.500)
- サーマルサイクラー(例:Clontech PCR Thermal Cycler GP(製品コード WN400))
- 80%(v/v)エタノール(用時調製)
この製品を見た人は、
こんな製品も見ています
-
NucleoMag® NGS Clean-up and Size Select
NGSライブラリーの精製、サイズ分画に
-

Single strand DNA Ligase
一本鎖DNA同士の連結に適した酵素
-

SMART-Seq® mRNA Long Read
微量サンプル対応!ONT社ロングリードシーケンサー用cDNA合成キット
-

Unique Dual Index Kits
イルミナ社NGSライブラリー調製キット用インデックスキット
-

ThruPLEX® DNA-Seq Kit
簡単操作、微量(50 pg)対応の、ChIP-seqに最適なDNAライブラリー調製キット
-

SMART-Seq® Total RNA High Input (RiboGone™ Mammalian)
total RNA 100 ng~1 μgからイルミナ社NGS用ライブラリーを迅速に調製
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。

