短鎖DNAからの高品質シーケンスを可能にするライブラリー調製法
■ はじめに
サンプルに含まれる短鎖DNAの解析が基礎研究あるいは臨床研究の分野において注目されるようになり、短鎖DNAからのシーケンスライブラリー調製に対するニーズが高まっています。しかし、短鎖DNAの解析においては、ライブラリー調製の過程で生じるアダプターダイマー等のサイズが短鎖DNAに近いため、解析が困難となります。このため、短鎖DNAからの解析において良好な結果を得るためには、シーケンスライブラリー調製の過程におけるアダプターダイマーの形成そのものを抑えることが必要となります。
ssDNA-Seq Kit(製品コード NN0003/NN0004)は、その高効率なライゲース技術により、アダプターダイマーの発生を抑えながらシーケンスライブラリーを調製することで、短鎖DNAからの高品質な解析を可能にしています。
本検討では、短鎖DNAに適したライブラリー調製プロトコールの性能を評価するため、合成オリゴDNAを用いたライブラリー収量、サイズ分布、シーケンス結果の比較を行いました。また、他社製品との比較も実施し、短鎖DNAからのライブラリー調製における本製品の優位性を検証しました。
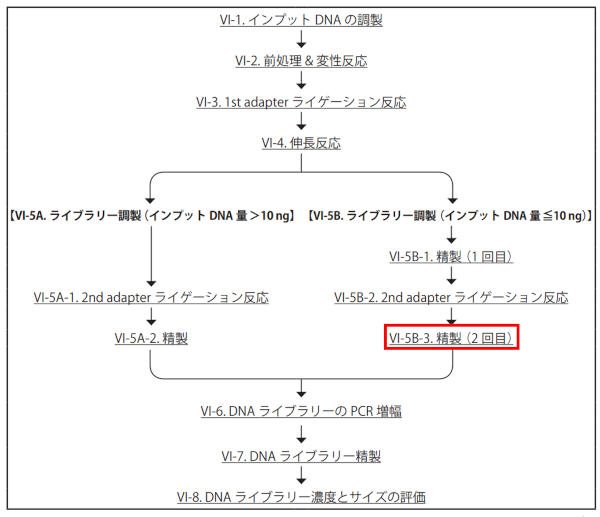
図1.ssDNA-Seq Kitのプロトコール(本製品ユーザーマニュアルより引用)
本製品を使用したライブラリー調製においてはVI-5B-3. 精製(2回目)の工程で磁気ビーズ量をA.基本プロトコール 30 μlより変更したB.短鎖DNA最適プロトコール 60 μlが用意されています。インサートサイズが50~160 bpの断片を含むDNAからライブラリー調製を行う場合にはインプット量に関わらずB.短鎖DNA最適プロトコールをご使用いただくことをお勧めします。
ssDNA-Seq Kit(製品コード NN0003/NN0004)は、その高効率なライゲース技術により、アダプターダイマーの発生を抑えながらシーケンスライブラリーを調製することで、短鎖DNAからの高品質な解析を可能にしています。
本検討では、短鎖DNAに適したライブラリー調製プロトコールの性能を評価するため、合成オリゴDNAを用いたライブラリー収量、サイズ分布、シーケンス結果の比較を行いました。また、他社製品との比較も実施し、短鎖DNAからのライブラリー調製における本製品の優位性を検証しました。

図1.ssDNA-Seq Kitのプロトコール(本製品ユーザーマニュアルより引用)
本製品を使用したライブラリー調製においてはVI-5B-3. 精製(2回目)の工程で磁気ビーズ量をA.基本プロトコール 30 μlより変更したB.短鎖DNA最適プロトコール 60 μlが用意されています。インサートサイズが50~160 bpの断片を含むDNAからライブラリー調製を行う場合にはインプット量に関わらずB.短鎖DNA最適プロトコールをご使用いただくことをお勧めします。
■ プロトコール比較:基本プロトコール vs 短鎖DNA最適プロトコール
プロトコール同士の比較を行うため、異なるサイズの合成オリゴDNA 2種類からなる混合物(160 base : 100 pg/50 base : 300 pg)を使用し、それぞれのプロトコールに基づき、本製品を使用してライブラリーを調製しました。
B.短鎖DNA最適プロトコールを使用してライブラリー調製を行ったところ、A.基本プロトコールの約2倍のライブラリー量が得られました(表1)。TapeStation HD5000(Agilent社)でライブラリーのサイズを評価したところ、B.短鎖DNA最適プロトコールにおいては160 base および50 base由来の2種類の波形がともに確認されました(図2)。また、いずれのプロトコールにおいてもアダプターダイマー由来のピークは確認されませんでした。
表1. 合成オリゴDNAの混合物*より調製されたライブラリーの収量
※合成オリゴDNAを2種類混合したもの(160 base : 100 pg/50 base : 300 pg)
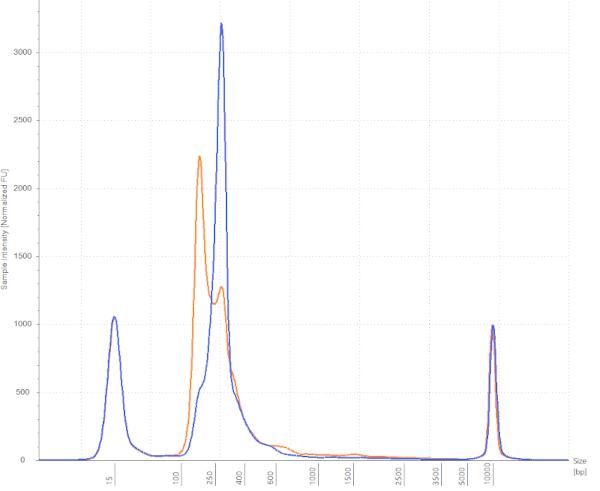
図2.合成オリゴDNAの混合物*より調製されたライブラリーのサイズ分布
A.基本プロトコール、B.短鎖DNA最適プロトコール
それぞれのプロトコールで調製されたライブラリーを使用し、シーケンス解析を行いました(図3)。Total Mapping(%)についてはいずれのプロトコールにおいても98%以上となり、アダプターダイマーの形成を抑制できていることをシーケンス結果からも確認できました。マッピングされた全リードに対して合成オリゴDNA 50 baseに由来するリードが占める割合を比較したところ、B.短鎖DNA最適プロトコールはA.基本プロトコールの約5倍となりました。この結果はB.短鎖DNA最適プロトコールを使用することにより、50 baseという極めて短いフラグメントからもシーケンス結果を得ることが可能であることを示唆します。
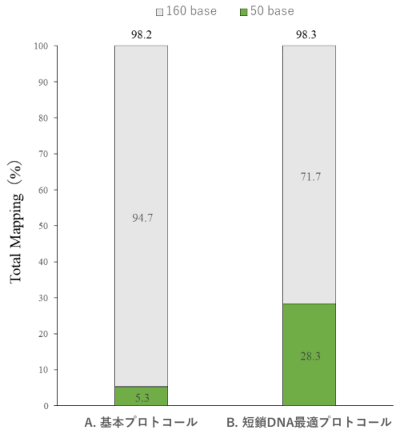
図3.合成オリゴDNAの混合物*より調製されたライブラリーのシーケンス結果
*合成オリゴDNAを2種類混合したもの(160 base : 100 pg/50 base : 300 pg)
NextSeq 2000(Illumina)でペアエンドシーケンス(2×150 bp)を行い、BWA-MEMを使用して160 baseおよび50 baseの既知配列にアライメントした。Total Mapping(%)は160 baseおよび50 baseの既知配列にアライメントされたリードの割合を示す。Total Mapping(%)の内訳は160 baseまたは50 base由来のリードの比率を色別に示す。
B.短鎖DNA最適プロトコールを使用してライブラリー調製を行ったところ、A.基本プロトコールの約2倍のライブラリー量が得られました(表1)。TapeStation HD5000(Agilent社)でライブラリーのサイズを評価したところ、B.短鎖DNA最適プロトコールにおいては160 base および50 base由来の2種類の波形がともに確認されました(図2)。また、いずれのプロトコールにおいてもアダプターダイマー由来のピークは確認されませんでした。
表1. 合成オリゴDNAの混合物*より調製されたライブラリーの収量
| ライブラリー収量(ng) | |
| A.基本プロトコール | 440 |
| B.短鎖DNA最適プロトコール | 848 |

図2.合成オリゴDNAの混合物*より調製されたライブラリーのサイズ分布
A.基本プロトコール、B.短鎖DNA最適プロトコール
それぞれのプロトコールで調製されたライブラリーを使用し、シーケンス解析を行いました(図3)。Total Mapping(%)についてはいずれのプロトコールにおいても98%以上となり、アダプターダイマーの形成を抑制できていることをシーケンス結果からも確認できました。マッピングされた全リードに対して合成オリゴDNA 50 baseに由来するリードが占める割合を比較したところ、B.短鎖DNA最適プロトコールはA.基本プロトコールの約5倍となりました。この結果はB.短鎖DNA最適プロトコールを使用することにより、50 baseという極めて短いフラグメントからもシーケンス結果を得ることが可能であることを示唆します。

図3.合成オリゴDNAの混合物*より調製されたライブラリーのシーケンス結果
*合成オリゴDNAを2種類混合したもの(160 base : 100 pg/50 base : 300 pg)
NextSeq 2000(Illumina)でペアエンドシーケンス(2×150 bp)を行い、BWA-MEMを使用して160 baseおよび50 baseの既知配列にアライメントした。Total Mapping(%)は160 baseおよび50 baseの既知配列にアライメントされたリードの割合を示す。Total Mapping(%)の内訳は160 baseまたは50 base由来のリードの比率を色別に示す。
■ 他社製品との比較
短鎖DNAからのライブラリー調製における各社製品の性能を比較するため、合成オリゴDNA 50 base 10 ngより、本製品およびA社キット、B社キット(それぞれssDNAからのライブラリー調製を可能とするキット)を使用してライブラリー調製を行いました。
各社キットで調製されたライブラリーの収量を比較したところ、本製品を使用した場合が最も多い結果となりました(表2)。また、得られたライブラリーより各1 ngを使用してそのサイズをTapeStation HD5000(Agilent社)で確認しました(図4)。その結果、本製品においては目的のライブラリー由来とみられる非常にシャープなピーク(約170 bp)が得られました。一方で、A社キットあるいはB社キットを使用した場合は、目的のライブラリー由来およびアダプターダイマー(100 bp未満)とみられるピークが確認されました。
表2. 各社キットにおける合成オリゴ 50 baseのライブラリー収量
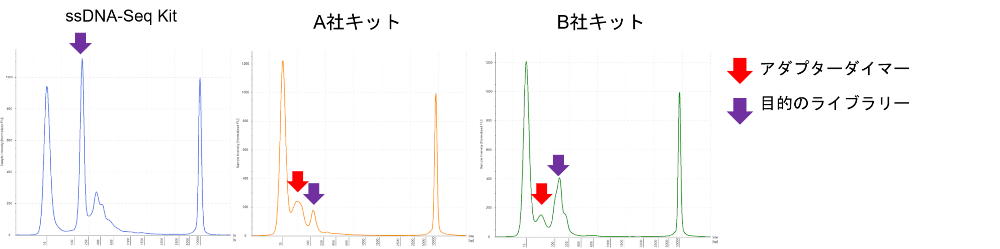 図4.各社キットにおける合成オリゴ50 baseのライブラリーのサイズ
図4.各社キットにおける合成オリゴ50 baseのライブラリーのサイズ
各社キットで調製されたライブラリーを使用してペアエンドシーケンス(2×150 bp)を行い、合計 1万リードとなるようダウンサンプリングした後に、BWA-MEMを使用して50 baseの既知配列にアライメントしました。本製品はMapping(%)が最も高い結果となり、アダプターダイマーの形成を抑制できていることがシーケンス結果からも示されました(図5)。
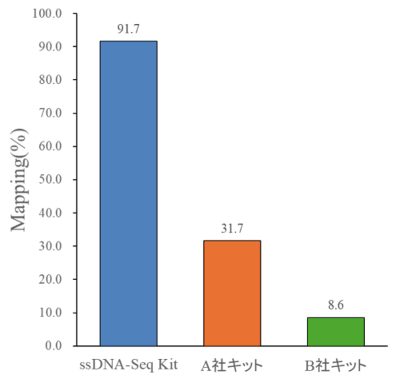
図5.各社キットで調製されたライブラリーのMapping(%)
各社キットのシーケンス結果に合成オリゴ50baseの全長が含まれているかをIntegrative Genomics Viewer(IGV)を用いて確認しました(図6)。本製品では合成オリゴ50 baseの全長に対して均一なカバレッジを得ることができました。一方で、A社およびB社キットでは末端領域のカバレッジが低下する結果となりました。

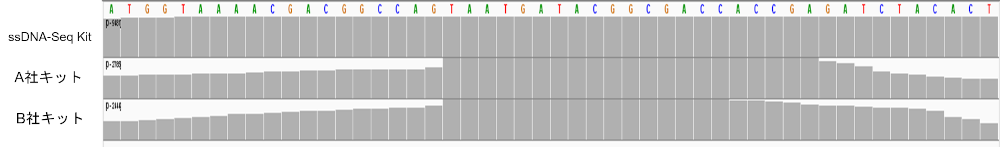 図6.各社キットにおける短鎖DNAの全長のシーケンスカバレッジ
図6.各社キットにおける短鎖DNAの全長のシーケンスカバレッジ
各社キットで調製されたライブラリーの収量を比較したところ、本製品を使用した場合が最も多い結果となりました(表2)。また、得られたライブラリーより各1 ngを使用してそのサイズをTapeStation HD5000(Agilent社)で確認しました(図4)。その結果、本製品においては目的のライブラリー由来とみられる非常にシャープなピーク(約170 bp)が得られました。一方で、A社キットあるいはB社キットを使用した場合は、目的のライブラリー由来およびアダプターダイマー(100 bp未満)とみられるピークが確認されました。
表2. 各社キットにおける合成オリゴ 50 baseのライブラリー収量
| ライブラリー収量(ng) | |
| ssDNA-Seq Kit | 171.6 |
| A社キット | 6.4 |
| B社キット | 7.0 |

各社キットで調製されたライブラリーを使用してペアエンドシーケンス(2×150 bp)を行い、合計 1万リードとなるようダウンサンプリングした後に、BWA-MEMを使用して50 baseの既知配列にアライメントしました。本製品はMapping(%)が最も高い結果となり、アダプターダイマーの形成を抑制できていることがシーケンス結果からも示されました(図5)。

図5.各社キットで調製されたライブラリーのMapping(%)
各社キットのシーケンス結果に合成オリゴ50baseの全長が含まれているかをIntegrative Genomics Viewer(IGV)を用いて確認しました(図6)。本製品では合成オリゴ50 baseの全長に対して均一なカバレッジを得ることができました。一方で、A社およびB社キットでは末端領域のカバレッジが低下する結果となりました。

■ まとめ
本製品ssDNA-Seq Kitは、短鎖DNAに特化したライブラリー調製プロトコールを備えており、従来法では困難だった短鎖フラグメントからも高効率かつ高品質なシーケンスデータの取得を可能にします。
短鎖DNA最適プロトコールでは、基本プロトコールとの比較において約2倍のライブラリー収量、約5倍の短鎖DNA由来リードの取得率を示し、さらに他社製品と比較しても圧倒的な収量・精度・カバレッジを達成しました。
短鎖DNAの解析において、アダプターダイマーの影響を最小限に抑え、信頼性の高いデータを得るためには、ssDNA-Seq Kitの使用が最適な選択肢です。
短鎖DNA最適プロトコールでは、基本プロトコールとの比較において約2倍のライブラリー収量、約5倍の短鎖DNA由来リードの取得率を示し、さらに他社製品と比較しても圧倒的な収量・精度・カバレッジを達成しました。
短鎖DNAの解析において、アダプターダイマーの影響を最小限に抑え、信頼性の高いデータを得るためには、ssDNA-Seq Kitの使用が最適な選択肢です。
