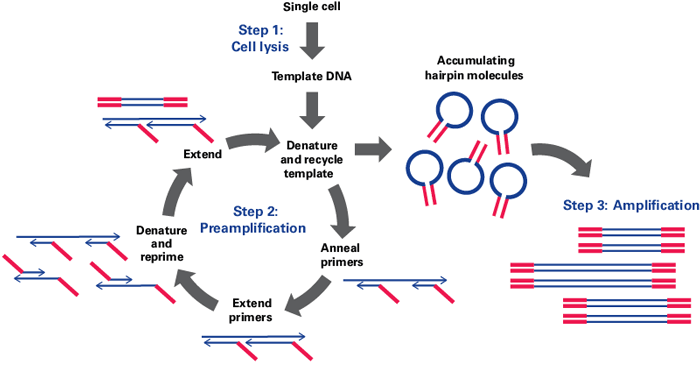
製品説明
従来のPicoPLEX WGA Kitで可能であった再現性の高いコピー数多型(CNV)解析に加えて、本キットでは、正確性の高い増幅用酵素を使用することにより増幅エラーを最小限に抑え、一塩基変異体(SNV)の検出が可能である。
使用できるインプットの種類;
- 哺乳類細胞(1~10細胞)
- 精製DNA(15~60 pg DNA)
- ソートされた染色体
- インタクトまたは断片化された一本鎖または二本鎖DNA
| Depth of SNV position ≧10 Allele frequency ≧20% |
PicoPLEX WGA v3 1 cell |
PicoPLEX WGA v3 5 cells |
A社 1 cell |
A社 5 cells |
B社 1 cell |
B社 5 cells |
|||||||
| Bulk | Rep1 | Rep2 | Rep1 | Rep2 | Rep1 | Rep2 | Rep1 | Rep2 | Rep1 | Rep2 | Rep1 | Rep2 | |
| Number of SNVs called | 74 | 57 | 67 | 69 | 67 | 34 | 57 | 62 | 67 | Failed | Failed | 40 | Failed |
| Number of false positives | 3 | 1 | 0 | 1 | 5 | 1 | 0 | 7 | Failed | Failed | 0 | Failed | |
| Average false positives | 0.02% | 0.005% | 0.03% | 0.035% | Failed | 0 | Failed | ||||||
| Call rate | 78% | 92% | 95% | 92% | 47% | 78% | 85% | 92% | Failed | Failed | 55% | Failed | |
| Average call rate | 85% | 93% | 62% | 88% | Failed | 55% | Failed | ||||||
| Missed | 17 | 7 | 5 | 7 | 40 | 17 | 12 | 7 | Failed | Failed | 34 | Failed | |
| Average locus dropouts | 16.2% | 8.1% | 38.5% | 12.8% | Failed | 45.9% | Failed | ||||||
| Number of heterozygous SNVs called | 45 | 45 | 38 | 45 | 45 | 36 | 31 | 38 | 41 | Failed | Failed | 32 | Failed |
| Average allele dropouts | 7.8% | 0.0% | 25.6% | 12.2% | Failed | 71.1% | Failed | ||||||
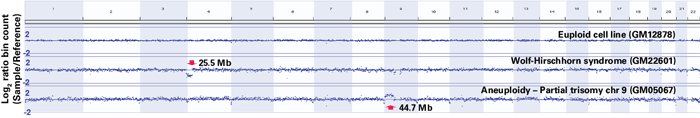
PicoPLEX Single Cell WGA Kit v3を使用して、さまざまな細胞株(GM22601、GM05067、GM12878)のシングルセルからゲノムを増幅した。精製した増幅産物 1 ngをIllumina Nextera XT Kitのインプットサンプルとして使用し、2×75 bpの読み取り長でIllumina MiSeqでシーケンスを行った。Fastqファイルは、アダプター配列を除去するためにトリミングされ、human genome assembly GRCh37にアライメントされた。常染色体のみが報告された。アライメントは1Mリードにノーマライズされ、1 MBのbinあたりのリード数はbedtools 2.25.0を使用して計算された。bin数のlog2比(サンプル/リファレンス)は、Integrative Genomics Viewer(IGV)を使用してプロットされた。PicoPLEX Single Cell WGA Kit v3は染色体の部分的異数性も高解像度で解析できることが示された。
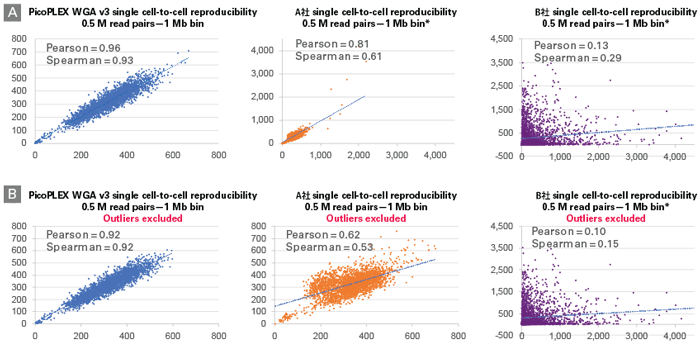
PicoPLEX Single Cell WGA Kit v3、A社、およびB社(MDA)キットを使用して、GM12878のシングルセルサンプルから2連で増幅産物を調製した。Illumina Nextera XT Kit のインプットサンプルとして増幅産物1 ngを使用し、ライブラリーを作製後、2×75 bpの読み取り長でIllumina MiSeqでシーケンスを行った。Human genome assembly GRCh37へのリードアライメントと100万リード(50万リードペア)へのノーマライズ後、1 Mbのbinあたりのリード数をbedtools 2.25.0を使用して計算した。2つのシングルセルライブラリーからの各ウィンドウの合計読み取り値をプロットし、ピアソン相関とスピアマン相関を計算して各グラフに示した。
Panel A.外れ値を含むサンプル
注)比較した各社キットのバイアスが異なり、相関ポイントが除外されていないため、グラフのスケールは異なる。
Panel B.外れ値を除外したサンプル
PicoPLEX WGA v3は、A社およびB社キットと比較して高い再現性と堅牢なカバレッジを示した。
内容
- Cell Extraction Buffer
- Extraction Enzyme Dilution Buffer
- Cell Extraction Enzyme
- Pre-Amplification Buffer
- Pre-Amplification Enzyme
- Amplification Buffer
- Amplification Enzyme
- Nuclease-Free Water
保存
用途
- NGS、oligonucleotide aCGH、BAC aCGH、またはqPCRによるコピー数多型(CNV)解析
- 変異検出のためのターゲット濃縮またはアンプリコン濃縮
- SNPジェノタイピング
本製品以外に必要な試薬、機器(主なもの)
- サーマルサイクラー
- 微量高速遠心機
- PCRチューブまたは 96-well PCRプレートとシール
- ピペットチップ
- PBS (1×PBS free of Mg2+、Ca2+) 細胞洗浄用
- 80% (v/v) エタノール
- Agencourt AMPure XP beads (Beckman Coulter社, Cat No.A63880)
- 0.2 mlチューブ用マグネットスタンド
この製品を見た人は、
こんな製品も見ています
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。


