製品説明
従来品に比べてv2キットは、イルミナ社シーケンサーにおけるシーケンスのパフォーマンスを改良する工夫がされており、また、より使いやすいライブラリー精製のワークフローになっている。
total RNA中に含まれるリボソームRNA(rRNA)(および一部ミトコンドリアRNA)は、rRNA特異的プローブを使用した独自技術を採用することにより、効率的に除かれる。その結果、シーケンスコストを下げ、マッピング精度(mapping statistics)を上げることができる。
v2キットで作製されたライブラリーは従来のキットで作製されたライブラリーとは異なる設計になっており、イルミナ社のNGS装置を使用する際、大量のPhiXを添加しなくても高いClusters passing filter(% PF)を得ることができる。その結果、シーケンスの1ランあたり、より生物学的に有意義なリードを得ることができるので、シーケンスコストを下げることができる。v2キットを用いたデータはrRNAやmtRNAにマッピングされるリードが少なく、duplicateの割合も低いため、よりユニークなtranscriptsを識別する傾向がある。さらにv2キットはPCR Bufferの組成を改良したことにより、ライブラリー精製の効率が増大した。
本キットには、12、48、96および192回用の製品があり、96回用および192回用(製品コード 634413、634414)の製品には12種類のReverseプライマーと8種類のForwardプライマーが含まれており、ハイスループット解析用に最大96インデックスが設定できる。
また、本キットにはインデックスプライマーが含まれているが、インデックスホッピング低減のためのユニークデュアルインデックスキット(別売)も使用可能である。
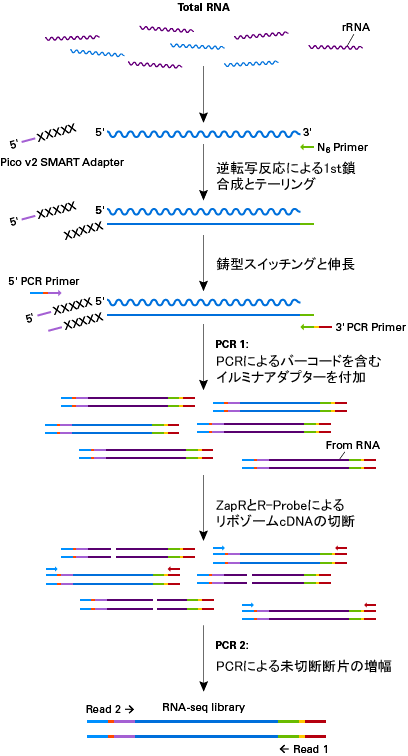
図1. 実験フローチャート

5' PCR Primer HTおよび3' PCR Primer HTにより付加されたアダプターは、イルミナ社シーケンサーのフローセルへの結合部位(P7、P5)およびイルミナ社 TruSeq HT Index(Index 1 [i5]、Index 2 [i7])の配列を含んでおり、シーケンスプライマーRead 2およびRead 1を認識する領域も含んでいる。
Read 1では元のRNAのアンチセンス側の配列が得られ、Read 2では元のRNAのセンス側の配列が得られる(元のRNAの方向は 5’→3’になる)。
Read 2を用いた場合の最初の3塩基 (XXX)はPico v2 SMART Adapter由来の配列になるので、paired-end sequencingを行った場合、この3塩基はマッピングの前にトリミングする必要がある。
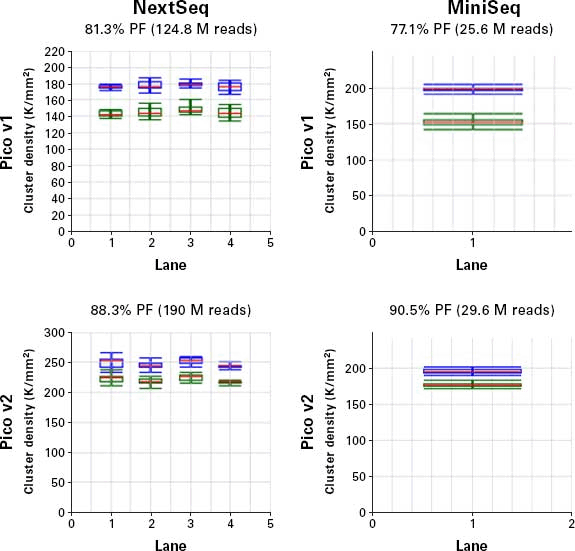
従来のキット(Pico v1)と本キット(Pico v2)を用いて作製したライブラリーを、NextSeq 500またはMiniSeqを用いてシーケンスを行った。それぞれのグラフにおいて、青い枠の部分はunfiltered (raw) リードのクラスター密度の分布を示している。一方、緑の枠の部分はfilteringを行ったリードのクラスター密度の分布を示している。
Passing filterのリード数(M:million)と%PFの値をグラフの上に表記した。
イルミナ社の規格では、予想されるPassing filterのリード数は、NextSeqの場合は130 M、MiniSeqの場合は25 Mである。PhiXはPico v2キットでは1%、Pico v1キットでは10%添加している。
Pico v2キットの場合、PhiXにアライメントされたリードの割合はすべてのシーケンスランにおいて、0.5%から1.15%の間である。
上記のグラフの結果により、Pico v2キットで作製されたライブラリーは、Pico v1キットに比べて高い% PFの値が得られた。
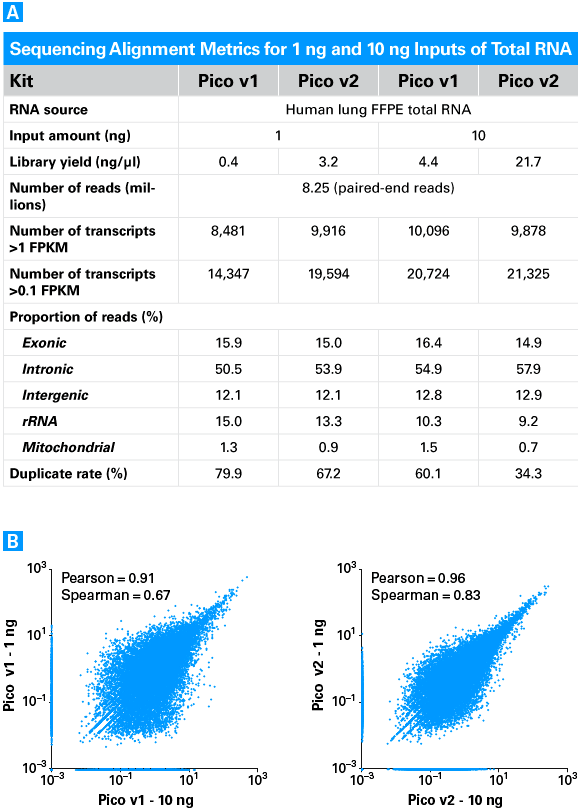
1 ngまたは10 ngのヒト肺FFPE組織から抽出したTotal RNAから、従来のキット(Pico v1)および Pico v2キットを用いてライブラリーを調製し、NextSeq 500でシーケンスを行った。
Panel A:1 ngおよび10 ng inputから調製したライブラリーの評価
どちらのinput量においてもPico v2を用いた場合、ライブラリーの収量が多く、rRNAやmtRNAにマッピングされるリード数の割合や、Duplicateの割合が低かった。1 ngのinputではPico v2のライブラリーから得られたシーケンスデータでは、Pico v1の場合に比べて数千以上多くのTranscript数が得られ、Pico v2では高い感度での解析が可能になった。
Panel B : 1 ngおよび10 ng inputから調製したライブラリーのFPKMs(Fragments Per Kilobase of Exon Per Million Fragments Mapped)の相関性
Pico v2キットを用いた場合、Pico v1キットに比べて1 ngと10 ngのinputのデータにおいて高い再現性が見られた。FPKMの値はlog10 scaleで表示している。
内容
- Package 1:
- SMART TSO Mix v2
- R-Probes v2
- Control Total RNA (1 μg/μl)
- Package 2
- ZapR v2
- SMART Pico Oligos Mix v2
- 5X First-Strand Buffer
- SMARTScribe RT (100 U/μl)
- RNase Inhibitor (40 U/μl)
- 10X ZapR Buffer
- SeqAmp DNA Polymerase
- SeqAmp CB PCR Buffer (2X):
- Tris Buffer (5 mM)
- PCR2 Primers v2
- Nuclease-Free Water
- Indexing Primer Set:
- 12回用(製品コード 634411)
- Indexing Primer Set HT for Illumina v2-12
- 3’ PCR Primer HT Index 2 (3’ 2; 12.5 μM)
- 5’ PCR Primer HT Index 1~12 (5’ 1~12; 12.5 μM)
- 48回用(製品コード 634412)
- Indexing Primer Set HT for Illumina v2- 48
- 3’ PCR Primer HT Index 1~4 (3’ 1~4 ; 12.5 μM)
- 5’ PCR Primer HT Index 1~12 (5’ 1~12; 12.5 μM)
- 96回用(製品コード 634413)、192回用(製品コード 634414)
- Indexing Primer Set HT for Illumina v2-96(192回用は2個含まれる。)
- 3’ PCR Primer HT Index 1~8 (3’ 1~8; 12.5 μM)
- 5’ PCR Primer HT Index 1~12 (5’ 1~12; 12.5 μM)
保存
Package 2:-20℃
Indexing Primer Set:-20℃
本製品以外に必要な試薬、機器(主なもの)
- シングルチャンネルピペット:10 μl、20 μlおよび200 μl
- 8連チャンネルピペット: 20 μl、200 μl
- Pre-PCR amplification用のPCRサーマルサイクラー
- PCR amplification用のPCRサーマルサイクラー
(Final RNA-seq Library Amplificationのステップを100μlの反応液でPCRを行うため、100μlの反応液量に対応したPCRサーマルサイクラーを使用する。50μlの反応液量に対応したサーマルサイクラーの場合は、2本のチューブにわけてPCRを行う。) - フィルターピペットチップ: 10 μl、20 μlおよび200 μl
- 0.2 ml tube minicentrifuge
- 96-well PCR chiller rack: IsoFreeze PCR Rack (MIDSCI社 Code 5640-T4) or 96 Well Aluminum Block(Light Labs社 Code A-7079)
PCR増幅およびバリデーション
- Agilent High Sensitivity DNA Kit(Agilent社 Code 5067-4626)
- Qubit dsDNA HS kit(Thermo Fisher Scientific社 Code Q32851)
- Nuclease-free thin-wall PCR tube(USA Scientific社;0.2 ml Code 1402-4700)
- Nuclease-free nonsticky 1.5 ml tubes (USA Scientific社 Code 1415-2600)
SPRI Bead精製
- Agencourt AMPure XP PCR Purification Kit(Beckman Coulter社; 5 ml, Code A63880、60 ml, Code A63881)
- 0.2 mlチューブ用マグネットスタンド
- 80% Ethanol(用時調製)
Technical Notes
Improved ease of use and sequencing performance for whole transcriptome analysis of high-quality or degraded samples
Stranded NGS libraries from FFPE samples
RNA-seqを成功させるためのコツ(英語)
ヒト生体液および細胞外小胞からのtotal RNA-seq(英語)
RNA-seqのヒント(英語)
無細胞核酸のシーケンス解析(英語)
Total RNA-seqの概要(英語)
SMARTer® Stranded Total RNA-Seq Kit v2 Pico Input Mammalianを用いたRNAバイオマーカー探索(英語)
この製品を見た人は、
こんな製品も見ています
-
次世代シーケンス(NGS)関連試薬選択ガイド RNAシーケンス
-

SMART-Seq® Stranded Kit
シングルセルまたはtotal RNA 10 pgからの方向性情報を持ったtotal RNA-Seq解析
-

SMART-Seq® Total RNA Mid Input
様々な生物種のRNAから方向性の情報をもつNGS用ライブラリー調製が可能
-

SMARTer® Stranded Total RNA-Seq Kit v3 - Pico Input Mammalian
UMI付き、微量FFPEサンプルからもRNA-Seqが可能
-

SMART-Seq® mRNA / SMART-Seq® mRNA LP
イルミナ社NGS装置用の超微量mRNA-Seq用ライブラリー調製キット
-

SMART-Seq® Total RNA Library Prep with ZapR® Depletion
微量total RNA(250 pg~1 μg)から方向性情報を持ったNGS用ライブラリー調製が可能
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。