断片化からライブラリー調製まで、1チューブ・わずか約2時間で完了
- 100 pg~200 ng(インプット量 30 μl)の二本鎖DNAが使用可能
- サイズ調整可能なDNA断片化酵素モジュールが含まれており、断片化装置は不要
- サンプルのロスを防ぎ、作業時間を短縮できる1チューブワークフロー
- GCリッチ領域も均一にカバーでき、高品質なシーケンスデータを取得可能
- Unique Dual Index Kit(製品コード 634752~634756)との組み合わせにより、384サンプルまでのマルチプレックス解析に対応可能
- コアキットであるThruPLEX DNA-Seq FLEXを個別に利用することで、断片化済みのDNAサンプルに対応可能
製品説明
ThruPLEX DNA-Seq FLEXは、ThruPLEXケミストリーを用いて、最大30 μlの二本鎖DNA(100 pg~200 ng)から、高品質なNGSライブラリーを調製できるキットです。エンドリペア、アダプターライゲーション、ライブラリー増幅の3ステップのワークフローを1本のチューブまたはウェル内で連続して行うことができ、約2時間(ハンズオンタイム15分)でライブラリー調製が完了します。
ThruPLEX技術により、ライゲーション後に余分なアダプターを分解するため、アダプター希釈や中間洗浄ステップは不要となります。精製やサンプルの移し替えも必要なく、サンプルロスや操作ミスを防ぎます。
ThruPLEX DNA-Seq FLEX EFは、インタクトな二本鎖DNAを断片化するThruPLEX FLEX Enzymatic Fragmentation Moduleとライブラリー調製用コアキットThruPLEX DNA-Seq FLEXで構成されています。
断片化サイズを調整可能なEnzymatic Fragmentation Moduleを用いることにより、機械的断片化や別の酵素による断片化を行っていた時間を省略することができ、効率的なライブラリー構築が可能です。
別売りのUnique Dual Index(UDI)キット(製品コード 634752~634756)との組み合わせにより、最大384サンプルのマルチプレックス解析に対応可能です。
本製品で調製したライブラリーは、イルミナ社次世代シーケンサーに対応しており、標準的な試薬・プロトコールでそのまま使用できます。また、全ゲノム解析(WGS)、全エクソーム解析(WES)、de novoシーケンス、ターゲットエンリッチメントなど、高カバレッジのディープシーケンスに最適です。
ThruPLEX DNA-Seq FLEXとThruPLEX DNA-Seq FLEX EFは、従来のThruPLEX DNA-Seq Kitよりも幅広いインプットDNA量(≦200 ng)とインプット液量(≦30 μl)に対応できるように最適化されています。
注意)ThruPLEX DNA-Seq FLEX EFは、ThruPLEX DNA-Seq HV PLUS Kit(製品コード R400783)のインデックスを別売りとしたリニューアル品になります。別売りのUnique Dual Index(UDI)キット(製品コード 634752~634756)と組み合わせてご使用ください。なお試薬の組成に変更はございませんが、インデックスの変更に伴いプロトコールが一部変更となっております。
ThruPLEX技術により、ライゲーション後に余分なアダプターを分解するため、アダプター希釈や中間洗浄ステップは不要となります。精製やサンプルの移し替えも必要なく、サンプルロスや操作ミスを防ぎます。
ThruPLEX DNA-Seq FLEX EFは、インタクトな二本鎖DNAを断片化するThruPLEX FLEX Enzymatic Fragmentation Moduleとライブラリー調製用コアキットThruPLEX DNA-Seq FLEXで構成されています。
断片化サイズを調整可能なEnzymatic Fragmentation Moduleを用いることにより、機械的断片化や別の酵素による断片化を行っていた時間を省略することができ、効率的なライブラリー構築が可能です。
別売りのUnique Dual Index(UDI)キット(製品コード 634752~634756)との組み合わせにより、最大384サンプルのマルチプレックス解析に対応可能です。
本製品で調製したライブラリーは、イルミナ社次世代シーケンサーに対応しており、標準的な試薬・プロトコールでそのまま使用できます。また、全ゲノム解析(WGS)、全エクソーム解析(WES)、de novoシーケンス、ターゲットエンリッチメントなど、高カバレッジのディープシーケンスに最適です。
ThruPLEX DNA-Seq FLEXとThruPLEX DNA-Seq FLEX EFは、従来のThruPLEX DNA-Seq Kitよりも幅広いインプットDNA量(≦200 ng)とインプット液量(≦30 μl)に対応できるように最適化されています。
注意)ThruPLEX DNA-Seq FLEX EFは、ThruPLEX DNA-Seq HV PLUS Kit(製品コード R400783)のインデックスを別売りとしたリニューアル品になります。別売りのUnique Dual Index(UDI)キット(製品コード 634752~634756)と組み合わせてご使用ください。なお試薬の組成に変更はございませんが、インデックスの変更に伴いプロトコールが一部変更となっております。
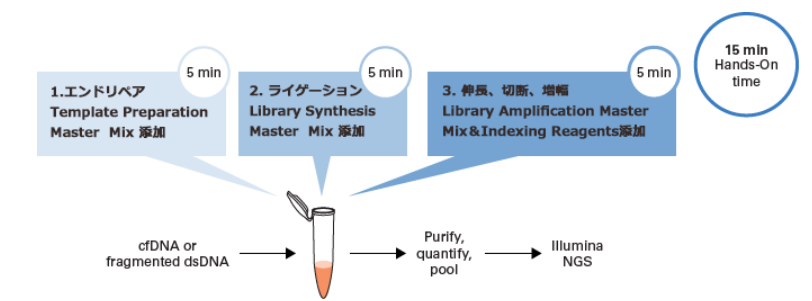
図1.ThruPLEX DNA-Seq FLEX EFの1チューブライブラリー調製のワークフロー
ThruPLEX DNA-Seq FLEX EFのワークフローは、同じPCRチューブまたはウェル内で行われる3つのシンプルなステップで構成されており、サンプルの精製や移し替えを行う必要がない。
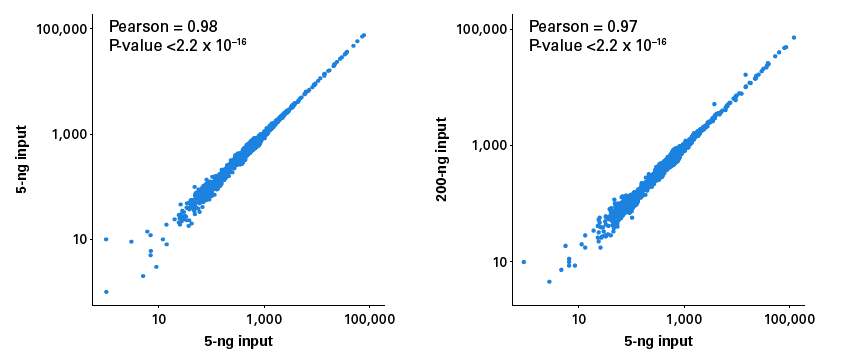
図2.ThruPLEX DNA-Seq FLEXで調製されたシーケンスライブラリーのインプット量に応じたライブラリーの均一なカバレッジ
ThruPLEX DNA-Seq FLEXを使用して、5 ng、50 ng、200 ngのヒトゲノムDNA(MerckMillipore社)からライブラリーを調製し、それぞれN=2で比較した。得られたシーケンス結果から、それぞれ2,000万リードを抽出してヒトゲノム(hg19)をリファレンスとしたマッピング解析を行い、各10 kb領域のカバレッジ(読み取りの均一性)を以下について比較した。
そのうち、5 ngのDNAから独立して調製した2つライブラリーの比較(左図)と5 ngと200 ngの異なるインプット量の比較(右図)を示す。いずれも高い再現性を示しており、ThruPLEX DNA-Seq FLEX は少量のDNAでも高い再現性と均一なカバレッジを実現できる優れたライブラリー調製システムであることが示された。
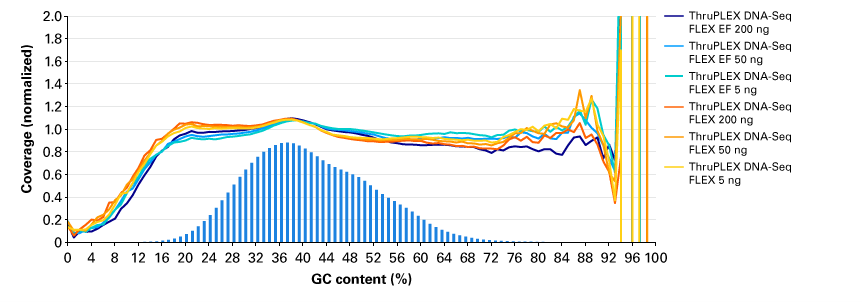
図3.ThruPLEX DNA-Seq FLEXおよびThruPLEX DNA-Seq FLEX EFでの各インプット量におけるGCカバレッジ
ThruPLEX DNA-Seq FLEX EF (DNAの断片化にはThruPLEX FLEX Enzymatic Fragmentation Moduleを使用)を用いて、NA12878 gDNAを5 ng、50 ng、200 ngのインプット量からそれぞれN=3でライブラリーを調製した。同様に、ThruPLEX DNA-Seq FLEX(DNAの断片化にはCovaris M220を使用)でも同じ条件でライブラリーを調製し、それぞれのサンプルについてIllumina NextSeq 500/550 Mid Output Kit v2.5(150サイクル)を用いてペアエンドシーケンスを実施した。得られたシーケンスデータは、各サンプルあたり500万リードにダウンサンプリングし、GC含量ごとのカバレッジ分布を比較した。青色の縦棒グラフは、100 bp windowsで算出された理論上のGC含量分布を示している。
どちらの結果もインプット量にかかわらず一貫したGCカバレッジを示しており、特にGCリッチ領域でも均等なカバレッジが得られることが確認された。
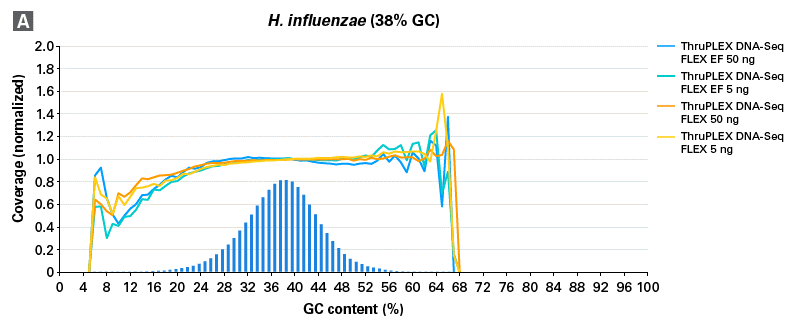
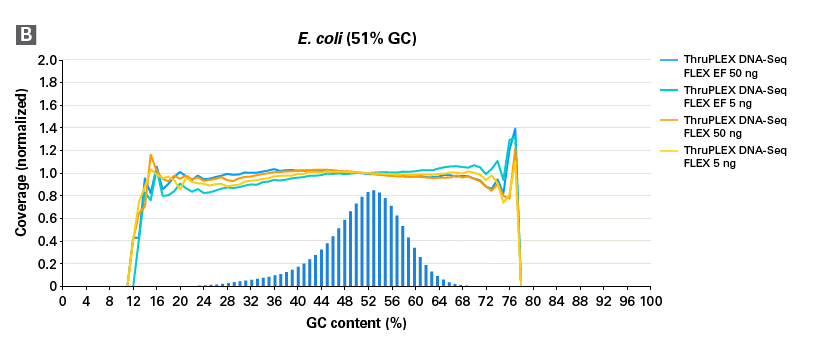
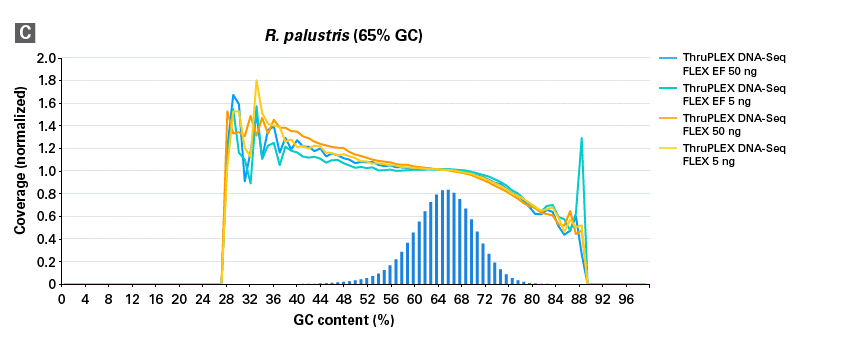
図4.ThruPLEX DNA-Seq FLEXおよびThruPLEX DNA-Seq FLEX EFにおける微生物サンプルのGCカバレッジ
ThruPLEX DNA-Seq FLEX(DNAの断片化にはCovaris M220を使用)およびFLEX EF(DNAの断片化にはThruPLEX FLEX Enzymatic Fragmentation Moduleを使用)を用いて、GC含量が異なる3種の微生物(Haemophilus influenzae(GC含量38%)、Escherichia coli(GC含量51%)、Rhodopseudomonas palustris(GC含量65%))のゲノムDNA(ATCC)から、5 ngおよび50 ngのインプット量でライブラリーをそれぞれN=3で調製した。AMPure beadsで精製後、Illumina NextSeq(150サイクル、2×75 bp)でペアエンドシーケンスを実施し、得られたデータを正規化して、GC含量ごとのカバレッジ分布として可視化した。
FLEX(橙、黄)とFLEX EF(青、緑)の結果は、いずれの微生物においてもGC含量の違いにかかわらず、均一なカバレッジを示した。青色の棒グラフは、各GC%における理論上の100 bp windowの分布を示している。
ThruPLEX DNA-Seq FLEXを使用してライブラリー調製を行った場合、酵素的断片化法を採用した1チューブワークフローのFLEX EFにおいても、物理的断片化法(FLEX)と同等の高い性能を発揮することが、多様なGC含量のサンプルに対して示された。
FLEX EFは、FLEXと同等の性能を示した。
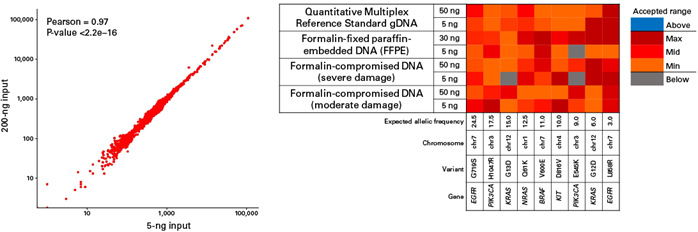
図5.FFPEサンプルの解析例
がんをはじめとする疾患の研究に欠かせないFFPEサンプルにおいては、DNAの劣化あるいは分解が進んでいることも多く、そのシーケンス解析は困難である。ThruPLEX FLEXを使用することにより、分解の進んだFFPEサンプルからも幅広いインプット量において高品質なデータを再現性良く取得することが可能となりる。
内容
- ThruPLEX DNA-Seq FLEX EF(製品コード 635101)
-
- ThruPLEX DNA-Seq FLEX(製品コード R400736)
- ThruPLEX FLEX Enzymatic Fragmentation Module*
Control Human gDNA (5 ng/μl)
10X PDF1
PBF1
PEF1
Nuclease-Free Water
- ThruPLEX DNA-Seq FLEX(製品コード R400736)
- Control Fragmented Human gDNA (5 ng/μl)
PBD1
PED1
SBD1
SED1
ABD1
AED1
Nuclease-Free Water
保存
-20℃
本製品以外に必要な試薬、機器(主なもの)
- Index Kit
- Unique Dual Index Kit (1-24)(製品コード 634756)
UDI No. U001-U024 - Unique Dual Index Kit (1-96)(製品コード 634752)
UDI No, U001-U096 - Unique Dual Index Kit (97-192)(製品コード 634753)
UDI No, U097-U192 - Unique Dual Index Kit (193-288)(製品コード 634754)
UDI No, U193-U288 - Unique Dual Index Kit (289-384)(製品コード 634755)
UDI No, U289-U384
- Unique Dual Index Kit (1-24)(製品コード 634756)
- Low TE緩衝液(10 mM Tris、0.1 mM EDTA、pH 8.0)
- 80%エタノール(v/v)
- NucleoMag NGS Clean-up and Size Select(製品コード 744970.5、744970.50、744970.500)またはAgencourt AMPure XP beads(Beckman Coulter社、Cat. No.A63880)
- ホットリッド付きPCRサーマルサイクラー(リアルタイム装置でも可)
- 遠心機
- PCR tube、または96-well nuclease-free thin-wall PCR plate
- 1.5 ml低吸着マイクロ遠心チューブ
- PCRプレート用シール(プレート使用時)
- シングルチャンネルピペット:10 μl、20 μl、200 μl
- マルチチャンネルピペット:20 μl、200 μl
- 低吸着フィルター付きピペットチップ:10 μl、20 μl、200 μl
- 低吸着エアロゾルバリア付きチップ
- マグネットスタンド
- フルオロメーター(例:Thermo Fisher Scientific Qubit)—ライブラリー定量用
他にあると便利な試薬や機器
- Illumina NGSライブラリーの定量用qPCRキット: Library Quantification Kit(製品コード 638324)
- ライブラリーサイズ分布の確認用:
Agilent BioanalyzerまたはTapeStation
技術情報
(Takara Bio USAのサイトへジャンプします)
- 様々なGC含量のゲノム解析(ヒト、微生物)における解析例
NGS library preparation with integrated enzymatic fragmentation—ThruPLEX DNA-seq FLEX EF
Streamlined DNA-seq from challenging samples - 他社製品との比較データ
Comparing ThruPLEX DNA-Seq FLEX EF to KAPA HyperPlus and NEBNext Ultra II FS
ThruPLEX FLEX outperforms NEBNext Ultra II for DNA-seq of cell-free and FFPE DNA
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。