ノンコーディングRNAを含むTotal RNA-Seqライブラリーを構築
- 1回の反応で5,000~100,000細胞から全長RNA-Seqライブラリを調製
- 一度の解析で最大12サンプルを解析
- non-coding RNAを含む全RNAの解析を実現
- 全長転写産物をカバーし、バリアントを同定(スプライシング、遺伝子融合など)
- 簡単な2日間のワークフロー
製品説明
Shastaシステムは、革新的なナノディスペンシング技術を採用し、より高精度で堅牢なシステムを実現しました。さらに、新しいサンプルバーコーディングフローにより、最大10万個の細胞をシングルセル解析することができます。ナノディスペンシング技術を使用した細胞の取得では、マイクロ流路タイプの手法と比較して、より広範囲な細胞サイズのサンプルの解析が可能です。Shastaシステムのテクノロジーにより、スプライシングアイソフォーム、遺伝子融合、染色体アームレベルのCNVなどのイベントを、これまで以上に多くの細胞で検出することができるようになりました。
Shasta Total RNA-Seq Kit は、市場初の大規模な全長scRNA-Seqソリューションであり、最大100,000個の単一細胞の全長遺伝子の包括的なプロファイリングを簡便かつ自動化されたプロトコルで実現します。ランダムプライミングベースの逆転写反応により、long non-coding RNA(lncRNA)を含む全RNAからの新規バイオマーカーの発見をサポートします。
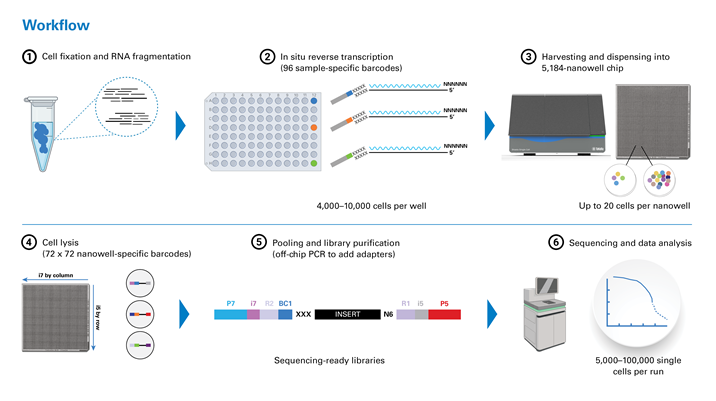
図1. Shasta Total RNA-Seqワークフロー
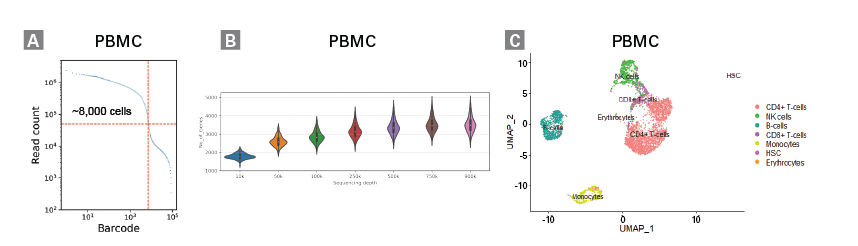
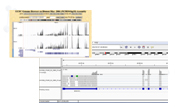
図2. Shasta Total RNA-Seq Kitを用いた不均一なPBMCサンプル中の異なる細胞集団の同定
Panel A: ~8,000個のシングルPBMCがQC閾値をパスしていることを示すバーコードランクプロット。赤い破線は、単一細胞Callの閾値を示す。
Panel B: 異なるシーケンシング深度で単一細胞ごとに検出された遺伝子数を示したバイオリンプロット図。ライブラリーは、Illumina NovaSeq 6000で単一細胞あたり平均490,000リードでシーケンスし、得られたデータはCogent NGSソフトウェアを用いた遺伝子数解析のために、X軸に示されている異なるシーケンシング深度にダウンサンプリングを行った。
Panel C: 遺伝子発現プロファイルに基づいてヒトPBMC内の異なる細胞集団を表示するUMAPプロット図
Shasta Total RNA-Seq Kit は、市場初の大規模な全長scRNA-Seqソリューションであり、最大100,000個の単一細胞の全長遺伝子の包括的なプロファイリングを簡便かつ自動化されたプロトコルで実現します。ランダムプライミングベースの逆転写反応により、long non-coding RNA(lncRNA)を含む全RNAからの新規バイオマーカーの発見をサポートします。

図1. Shasta Total RNA-Seqワークフロー
- ① 細胞固定およびRNA断片化
- ② 透過処理された細胞をin situ逆転写反応の容器とし、サンプル固有のバーコードを付加するため、96種類のバーコードを持つtemplate switching oligo (TSO)プレートにサンプルをロード
- ③ サンプルを回収し、Shasta Single-Cell Systemを用いて5,184のナノウェルに分注
- ④ 細胞溶解と72 i5および72 i7プライマーによるバーコードの組込み
- ⑤ ライブラリーをチップから回収。オフチップPCRおよび精製後にシーケンシング可能なライブラリーへの変換と、rRNA由来の配列をZapRテクノロジーにより除去
- ⑥ シーケンスとCogent NGSツールを用いたデータ解析

図2. Shasta Total RNA-Seq Kitを用いた不均一なPBMCサンプル中の異なる細胞集団の同定
Panel A: ~8,000個のシングルPBMCがQC閾値をパスしていることを示すバーコードランクプロット。赤い破線は、単一細胞Callの閾値を示す。
Panel B: 異なるシーケンシング深度で単一細胞ごとに検出された遺伝子数を示したバイオリンプロット図。ライブラリーは、Illumina NovaSeq 6000で単一細胞あたり平均490,000リードでシーケンスし、得られたデータはCogent NGSソフトウェアを用いた遺伝子数解析のために、X軸に示されている異なるシーケンシング深度にダウンサンプリングを行った。
Panel C: 遺伝子発現プロファイルに基づいてヒトPBMC内の異なる細胞集団を表示するUMAPプロット図
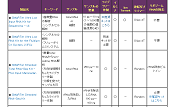
内容
Shasta Total RNA-Seq Kit - 2 Chip (製品コード 640288)
| Shasta Total RNA-Seq Reagents | 1 Kit |
| Shasta Long Indexing Primer Set - A (製品コード 640283) | 2 Chip |
| Single-Cell 350v Chip(製品コード 640019) | 2 Chip |
| Single-Cell Loading Kit(製品コード 640206) | 2 Kit |
| Single-Cell Collection Kit(製品コード 640212) | 2 Kit |
保存
Shasta Total RNA-Seq Reagents
Package 1: -70℃
Package 2: -20℃
その他 室温
Package 1: -70℃
Package 2: -20℃
その他 室温
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。