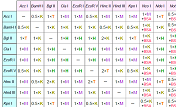
活性の定義
制限酵素活性の1 Uは、各酵素反応液50 μl中、原則として37℃で1時間に1 μgのλDNAを完全に分解する酵素量とする。なお、活性測定に用いた基質および反応温度については、酵素ごとに記載している。他のUniversal Bufferでの相対活性は、「Universal Buffer・Basal Bufferによる制限酵素活性表示システム」に一括して記載している。
純度検定
各酵素Lotごとに下記の項目を基本にチェックしている。
1. Overdigestion Test
各精製酵素について、基質DNA(通常λDNA)1 μgと過剰量の酵素とを16時間反応させる。その後、アガロースゲル電気泳動のDNA泳動パターンによりnon-specific DNaseの有無を判定している。
2. Genome DNA Analysis
選定した制限酵素(酵素ごとに明記)についてチェックする。適当なBacterial Genome DNA(Agarose embedded、0.5μg DNA/50 μl Gel)に対して、20~150 Uの制限酵素を加え、16時間反応させる。その後パルスフィールド電気泳動を行い、DNA泳動パターンに乱れがないことを確認している。
概要については「Genome DNA Analysis Gradeについて」に一括して記載している。
3. Ligation-Recutting Test
基質DNAに対して、まず、2~50倍過剰の酵素を作用させる。切断されたDNAを回収し、5'末端濃度が0.1~1.0 μMになるようにT4 DNA Ligase Buffer[66 mM Tris-HCI(pH7.6)、6.6 mM MgCl2、10 mM DTT、0.4 mM ATP]に溶解させる。適当量のT4 DNA Ligaseを加え、16℃で1時間あるいは16~18時間反応させる。DNA回収後、制限酵素反応液に溶解させ、同じ制限酵素でDNAを再切断する。以上の結果により、ligase inhibitor、phosphatase、exonucleaseの有無を判定している。なお、Ligation効率、Recutting効率について、タカラバイオの品質規格を「Ligation-Recutting効率品質規格について」に一括して記載している。
4. Labeled oligonucleotide assay (LOA) testまたはExonuclease Contamination Test※
LOA testは、酵素および試薬溶液のNuclease汚染の有無を確認するテストである。
5’末端または3’末端を蛍光標識したオリゴDNA(制限酵素切断部位を持たない)を制限酵素とともに1時間反応後、反応液を電気泳動しオリゴDNAの長さを測定する。
検出されるDNA断片長の変化から、各種Nuclease汚染(5'-exonuclease、3'-exonuclease、endonuclease)の有無を判定する。
Exonuclease Contamination Testは、試薬溶液のExonuclease汚染の有無を確認するテストである。
検出対象とは異なる制限酵素を用いて、3'末端が突出したDNA断片と5'末端が突出したDNA断片をそれぞれ作製し、その両断片を検出対象の制限酵素と混合してライゲーション反応後、電気泳動を行う。検出対象の酵素にExonucleaseの汚染がない場合、DNA断片が効率よくライゲーションされる。ライゲーションされていないDNA断片の有無により、検出対象酵素のExonuclease汚染の有無を判断する。
※試験方法は各ロットのCoAをご確認ください。
1. Overdigestion Test
各精製酵素について、基質DNA(通常λDNA)1 μgと過剰量の酵素とを16時間反応させる。その後、アガロースゲル電気泳動のDNA泳動パターンによりnon-specific DNaseの有無を判定している。
2. Genome DNA Analysis
選定した制限酵素(酵素ごとに明記)についてチェックする。適当なBacterial Genome DNA(Agarose embedded、0.5μg DNA/50 μl Gel)に対して、20~150 Uの制限酵素を加え、16時間反応させる。その後パルスフィールド電気泳動を行い、DNA泳動パターンに乱れがないことを確認している。
概要については「Genome DNA Analysis Gradeについて」に一括して記載している。
3. Ligation-Recutting Test
基質DNAに対して、まず、2~50倍過剰の酵素を作用させる。切断されたDNAを回収し、5'末端濃度が0.1~1.0 μMになるようにT4 DNA Ligase Buffer[66 mM Tris-HCI(pH7.6)、6.6 mM MgCl2、10 mM DTT、0.4 mM ATP]に溶解させる。適当量のT4 DNA Ligaseを加え、16℃で1時間あるいは16~18時間反応させる。DNA回収後、制限酵素反応液に溶解させ、同じ制限酵素でDNAを再切断する。以上の結果により、ligase inhibitor、phosphatase、exonucleaseの有無を判定している。なお、Ligation効率、Recutting効率について、タカラバイオの品質規格を「Ligation-Recutting効率品質規格について」に一括して記載している。
4. Labeled oligonucleotide assay (LOA) testまたはExonuclease Contamination Test※
LOA testは、酵素および試薬溶液のNuclease汚染の有無を確認するテストである。
5’末端または3’末端を蛍光標識したオリゴDNA(制限酵素切断部位を持たない)を制限酵素とともに1時間反応後、反応液を電気泳動しオリゴDNAの長さを測定する。
検出されるDNA断片長の変化から、各種Nuclease汚染(5'-exonuclease、3'-exonuclease、endonuclease)の有無を判定する。
Exonuclease Contamination Testは、試薬溶液のExonuclease汚染の有無を確認するテストである。
検出対象とは異なる制限酵素を用いて、3'末端が突出したDNA断片と5'末端が突出したDNA断片をそれぞれ作製し、その両断片を検出対象の制限酵素と混合してライゲーション反応後、電気泳動を行う。検出対象の酵素にExonucleaseの汚染がない場合、DNA断片が効率よくライゲーションされる。ライゲーションされていないDNA断片の有無により、検出対象酵素のExonuclease汚染の有無を判断する。
※試験方法は各ロットのCoAをご確認ください。
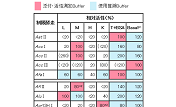
保存温度・保存形状
各酵素とも-20℃で保存(ただし、Aat II については-80℃凍結保存)。
各酵素とも一回程度の凍結溶解であれば失活はみられない。保存形状については、「制限酵素形状一覧表」にその保存緩衝液および組成を一括して記載している。
各酵素とも一回程度の凍結溶解であれば失活はみられない。保存形状については、「制限酵素形状一覧表」にその保存緩衝液および組成を一括して記載している。
添付Bufferについて
タカラバイオでは、各制限酵素に活性測定に用いた10×Bufferを添付している。また、全制限酵素に10×Loading Bufferを添付している。
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。