微量サンプル対応!ONT社ロングリードシーケンサー用cDNA合成キット
- 微量サンプルから優れた感度でOxford Nanopore Technologies(ONT)社ロングリードシーケンスが可能
- 細胞(1~1,000個)、またはtotal RNA (10 pg~100 ng)からバーコード付きの長鎖NGS用cDNAを合成
- 高品質のロングリードシーケンスデータを取得可能 - 平均リード長約2 kb(N50)、8 kbを超える完全長転写産物を検出
- シームレスにバーコードを付加 - マルチプレックスシングルチューブロングリードライブラリー用のcDNAを最大96サンプル調製可能
- 簡単で効率的なデマルチプレックスプロトコールによりONT社のベースコーラーと互換性がある正確なデータを取得可能
※色文字での表示は、キャンペーン価格およびそのキャンペーン期間です。
※ライブラリー調製には、ONT社製Ligation Sequencing Kit V14(Cat. No. SQK-LSK114)等が別途必要です。
※ライブラリー調製には、ONT社製Ligation Sequencing Kit V14(Cat. No. SQK-LSK114)等が別途必要です。
カートにいれる
WEB会員ログインが必要です
製品説明
SMART-Seq mRNA Long Readは、微量サンプルからロングリードシーケンス用のcDNAを合成するキットで、特に微量サンプルからの全トランスクリプトーム解析に適しています。10 pg~100 ngのtotal RNA(RIN>8)、または1~1,000個のインタクトな細胞から、Oligo dTプライマーを用いて完全長cDNAを合成し、続いてバーコードプライマーを用いてcDNA増幅することにより、最大96個のバーコード付きcDNAを合成できます。その後、Oxford Nanopore Technologies(ONT)社のプラットフォームでアダプターライゲーション、ライブラリー調製を行い、ロングリードシーケンスを実施します。一連の操作により、平均リード長約2 kb(N50)、8 kbを超える完全長転写産物の検出が可能です。
cDNA合成、バーコード付加、濃縮、バーコード付きcDNAのプールを含む一連の操作は1日半で完了し、本製品のワークフローは自動化、小型化にも適しています。本製品によって取得されたデータはタカラバイオが提供するプロトコールとファイルを用いて簡単にデマルチプレックスでき、ONT社のDoradoベースコーラーで使用することができます。
本製品のcDNA合成にはSMART(Switching Mechanism At 5’End of RNA Template)法を用いており、高い効率で完全長cDNA合成が可能です。転写産物の全長情報を効率的に取得することができ、転写産物アイソフォーム、遺伝子融合、点変異などの分析を可能にします。さらに、SMART法は感度と再現性が非常に高いため、サンプルのインプット量が少ない場合でも、他の方法に比べて、より多くの遺伝子を識別できます。本製品を用いることで、高い再現性、均一なカバレッジ、およびGCリッチ転写産物の正確な検出が可能です。
cDNA合成、バーコード付加、濃縮、バーコード付きcDNAのプールを含む一連の操作は1日半で完了し、本製品のワークフローは自動化、小型化にも適しています。本製品によって取得されたデータはタカラバイオが提供するプロトコールとファイルを用いて簡単にデマルチプレックスでき、ONT社のDoradoベースコーラーで使用することができます。
本製品のcDNA合成にはSMART(Switching Mechanism At 5’End of RNA Template)法を用いており、高い効率で完全長cDNA合成が可能です。転写産物の全長情報を効率的に取得することができ、転写産物アイソフォーム、遺伝子融合、点変異などの分析を可能にします。さらに、SMART法は感度と再現性が非常に高いため、サンプルのインプット量が少ない場合でも、他の方法に比べて、より多くの遺伝子を識別できます。本製品を用いることで、高い再現性、均一なカバレッジ、およびGCリッチ転写産物の正確な検出が可能です。
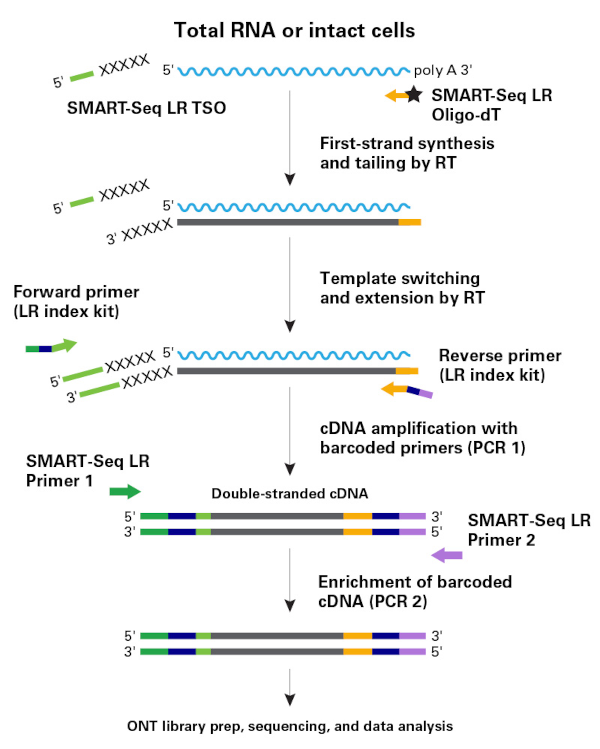
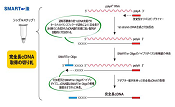
図1.ワークフロー
total RNA、または細胞からSMART-Seq LR Oligo-dTプライマーとMMLV由来の逆転写酵素(SMARTScribe Reverse Transcriptase)を用いて逆転写反応を行う。
各mRNAの5’末端まで逆転写が進むと5’末端への塩基のテ―リングが起こり、そこにSMART-Seq LR TSOがアニーリングして鋳型スイッチングにより5’末端にSMART-Seq LR TSOを鋳型とした配列が付加される。
SMART-Seq LR Index Plateに含まれるプライマーを用いた1回目のPCRによってバーコードを付加したcDNAが増幅される。
2回目のPCRによってバーコード付加されたcDNAが濃縮される。
サンプルをプールし、ONT社のLigation Sequencing Kit V14(Cat. No. SQK-LSK114)を用いて末端処理、シーケンスアダプター付加を行い、シーケンスを実施する。シーケンス後は、ONT社のGuppyを使用してベースコール、デマルチプレックスを行い、Cutadapt、Minimap2、SAMtools、Bedtools、Salmon等を用いて解析する。
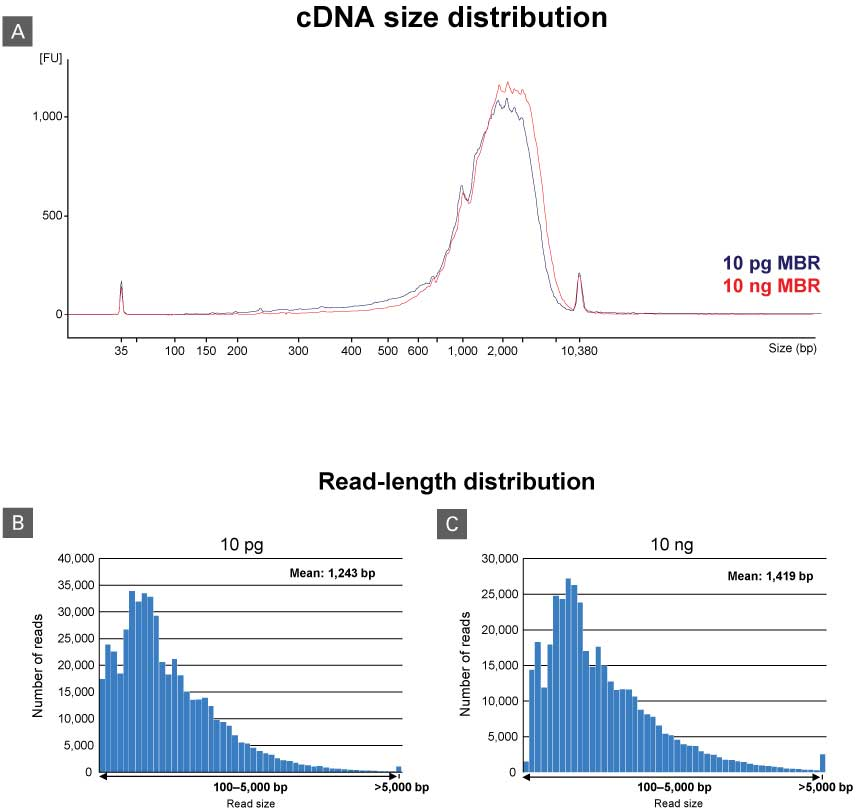
図2.SMART-Seq mRNA Long Readで合成されたバーコード付きcDNAのサイズ分布
SMART-Seq mRNA Long Readを用いて10 pgまたは10 ngマウス脳total RNA(n=8)からcDNAを合成し、Agilent High Sensitivity DNA Kitを用いて2100 Bioanalyzer(Agilent Technologies)でcDNAのサイズ分布を測定した。
パネルA:バーコード付加cDNAをプールして、ONT社のLigation Sequencing Kit V14を用いてライブラリーを調製し、MinION Flow Cellで72時間シーケンスした。Guppyを用いてベースコール、デマルチプレックスし、MS-Excelを用いてリード長の分布をプロットした。
パネルB、C:10 pg(パネル B)および 10 ng(パネル C)のマウス脳total RNAの代表的なサンプルのリード長分布を示した。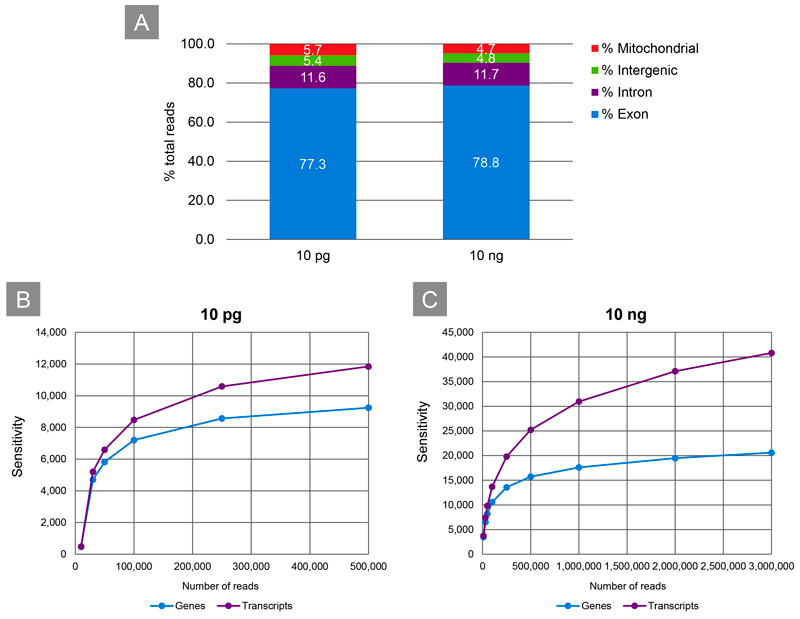
図3. SMART-Seq mRNA Long Readによって調製されたライブラリーのリード分布と感度
SMART-Seq mRNA Long Readを用いて10 pgまたは10 ngのマウス脳total RNAからcDNAを合成し、ライブラリーを調製した。シーケンス後、ONT社のGuppyを用いてベースコール、デマルチプレックスし、それぞれのバーコードあたり400,000リードにダウンサンプリングした。
パネルA:ライブラリーのリード分布。いずれのインプット量でも一貫して優れた結果が得られた(10 ngの場合はn=8、10 pgの場合はn=7)。
パネルB、C:遺伝子および転写産物の検出感度を評価するためにダウンサンプリングして比較した。いずれの条件(10 pg、10 ng)も飽和状態まではシーケンスされていないが、リード深度全体にわたって高感度に遺伝子(Gene)と転写産物(Transcript)が検出された。
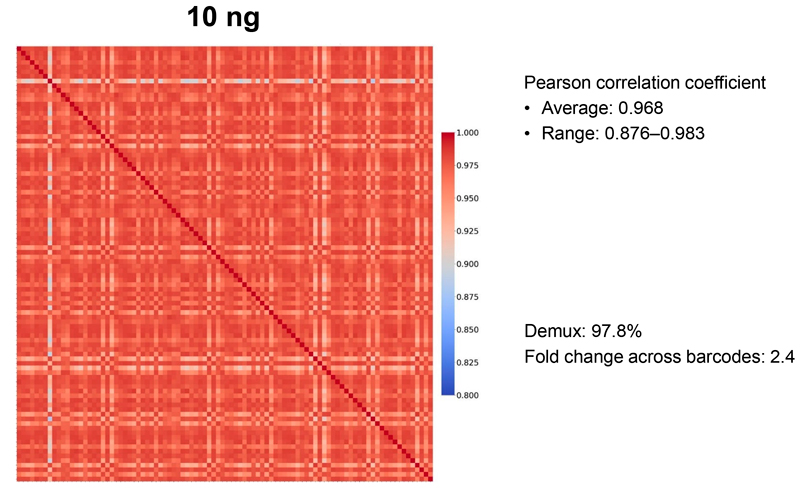
図4. SMART-Seq mRNA Long Readの再現性
SMART-Seq mRNA Long Readを用いて10 ngマウス脳total RNAの96個のレプリケートサンプルからcDNAを合成した。96サンプル全てのバーコード付きcDNAをプールしてライブラリーを調製しシーケンスを実施した結果、高い再現性が確認された。
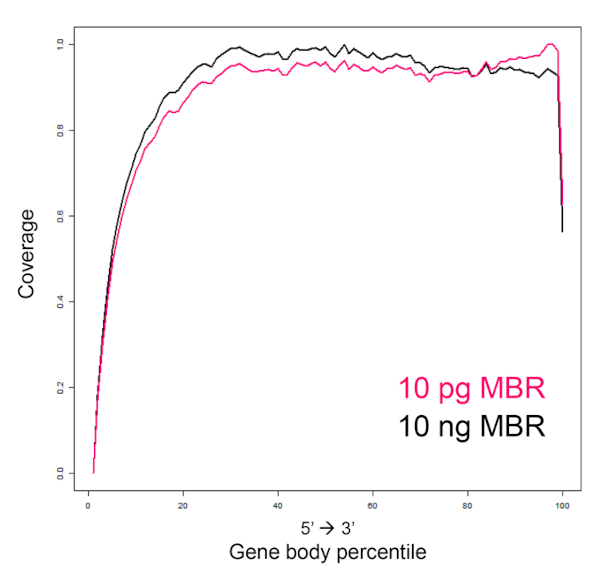
図5. SMART-Seq mRNA Long Readのカバレッジ
SMART-Seq mRNA Long Readを用いて10 pgまたは10 ngのマウス脳total RNAからcDNAを合成し、ライブラリー調製、シーケンスを行った。n=8の平均値を用いて遺伝子全体のカバレッジを評価した結果、10 pgというごく少量のインプット量でも高いカバレッジが得られた。
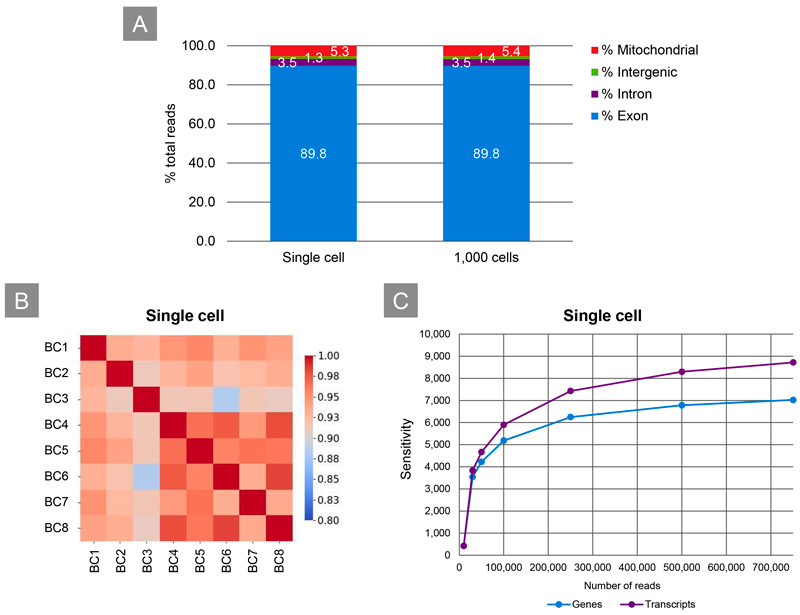
図6. 細胞サンプルからのロングリードシーケンス
SMART-Seq mRNA Long Readを用いて、シングルセル、または1,000個のK562細胞からcDNAを合成し、ライブラリー調製、シーケンスを実施した。Guppyを使用してベースコール、デマルチプレックスし、バーコードあたり300,000リードにダウンサンプリングした。
パネル A:平均遺伝子数(シングルセルはn=8、1,000個の細胞はn=2)
パネル B:ピアソン相関(シングルセルn=8の遺伝子マトリックスから算出)
パネル C:シングルセルのダウンサンプリング分析(リード深度あたりの遺伝子数、転写産物数を表示)
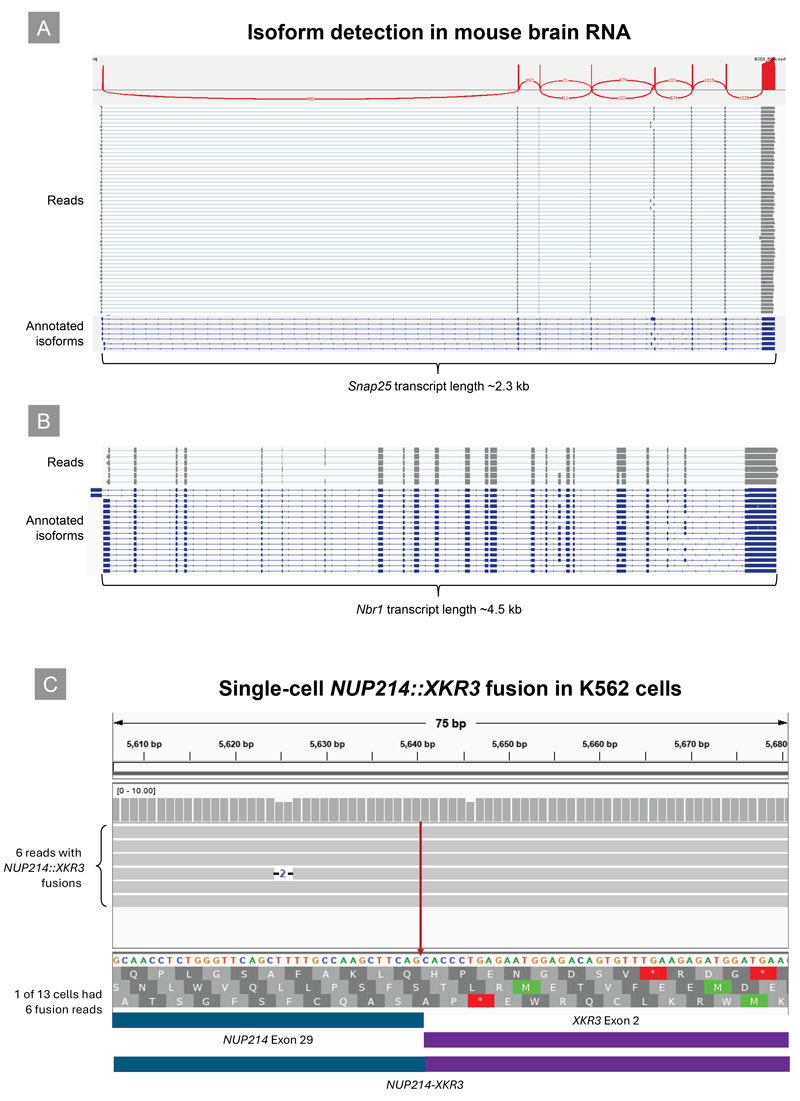
図7. 完全長アイソフォームと遺伝子融合の検出
SMART-Seq mRNA Long Readのワークフローに従って10 pgマウス脳total RNA、またはK562細胞のシングルセルからcDNA合成し、ライブラリー調製、シーケンスを実施した。ONT社のGuppyを用いてベースコール、デマルチプレックスし、Minimap2を用いてアライメントを実施した。
パネル A、B:10 pgマウス脳total RNAから検出された Snap25(パネル A)とNbr1(パネルB)のアイソフォーム、IVGで可視化
パネル C:K562細胞のシングルセルから検出されたNUP214::XKR3 遺伝子融合、IVGで可視化
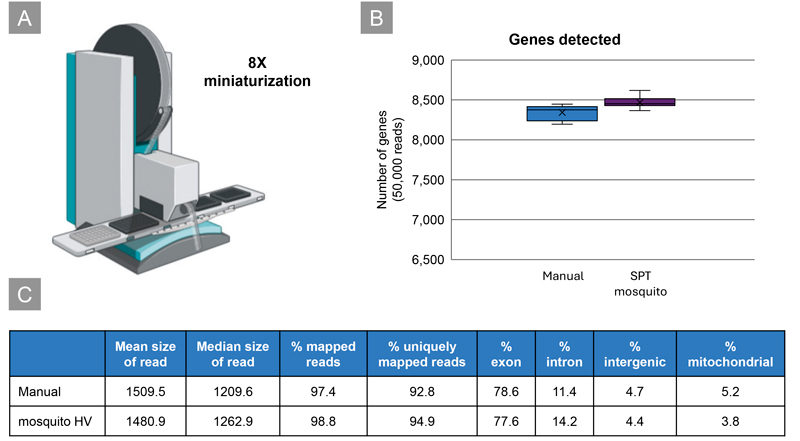
図8. SMART-Seq mRNA Long Readワークフローの自動化および小型化の検討
10 ngマウス脳total RNAの96個のレプリケート、およびシングルセルをサンプルとして、SMART-Seq mRNA Long Readのワークフローの自動化、および小型化を検討した。一方はベンチトップ上のマニュアル操作によって標準液量でcDNA合成を実施し、もう一方は自動化装置mosquito HV(SPT Labtech社)を用いて1/8の反応液量でcDNA合成を実施した。それぞれ96サンプル全てのバーコード付きcDNAをプールしてライブラリーを調製し、 72時間シーケンスした。ONT社のGuppyを用いてベースコール、デマルチプレックスし、Minimap2を用いてアラインメントした。
パネル A:mosquito HV 液体ハンドラー
パネル B:Salmonで評価した遺伝子数(マウス脳total RNA)
パネル C:マニュアル操作で合成したcDNAとmosquito HVで自動化、小型化して合成したcDNAの比較(シングルセル)
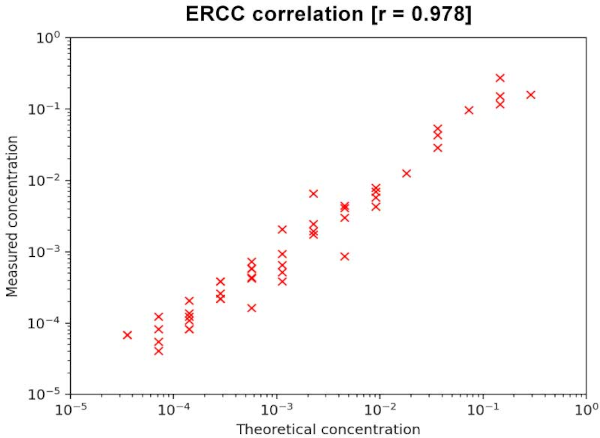
図9. mRNA Reference配列を用いた性能評価(測定濃度)
ERCC(External RNA Control Consortium)定量化コントロールとLong SIRV mRNA standardの両方を含む SIRV-Set 4(Lexogen社)をmRNA reference standardとして用い、SMART-Seq mRNA Long Readの性能を評価した。10 ngのマウス脳RNAにSIRV-Set 4をリードの約5%を占める量となる様にスパイクしてcDNAを合成し、ライブラリーを調製した。ONT社のMinIONでシーケンスし、Restranderツールを使用してFASTQデータのリード-ストランド補正を行い、minimap2で整列させてERCCスタンダードの理論濃度に対して測定濃度をプロットした。

図10. mRNA reference standardを用いた性能評価(IGVプロット)
ERCC(External RNA Control Consortium)定量化コントロールとLong SIRV mRNA standardの両方を含むSIRV-Set 4(Lexogen社)をmRNA reference standardとして用い、SMART-Seq mRNA Long Readの性能を評価した。10 ngのマウス脳RNAにSIRV-Set 4をリードの約5%を占める量となる様にスパイクしてcDNAを合成し、ライブラリー調製した。ONT社のMinIONでシーケンスし、Restranderツールを使用してFASTQデータのリード-ストランド補正を行い、minimap2で整列させた。
ERCCおよびSIRVアイソフォームセットに含まれる1 kb、4 kb、6 kb、8 kbのデータについてIGVプロットを示した。赤色のリードはプラス鎖リード、青色は非常に稀なマイナス鎖リード、小さな紫色または赤色のマークはONTシーケンスで一般的に見られる小さなインデル/変異を示している。なお、予想されるmRNA配列の少なくとも90%をカバーするリードを全長として定義した。
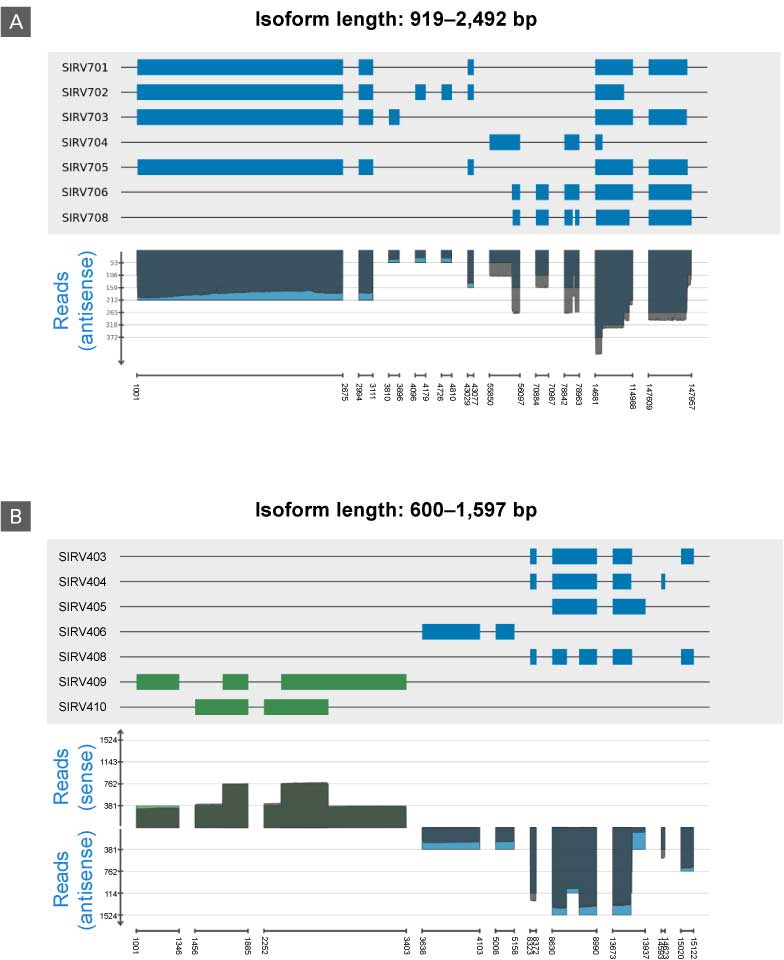
図11. SMART-Seq mRNA Long Readで合成したcDNAの完全性と方向性
Spike-In RNA Variant(SIRV)コントロール(600~2,492 bp)を用い、高い確率で転写産物(Transcript)全長を含むと考えられる全長cDNAの検出を行った。また、SIRVsuiteを用いてSIRV-spike-inに含まれる全てのアイソフォームの方向性を識別し可視化した。
パネル A:完全長cDNAのカバレッジ分析
パネル B:SIRVsuiteで可視化されたSIRV-spike-inの鎖方向の識別結果
内容
- SMART-Seq mRNA Long Read
-
Package 1
- Control Total RNA (1 μg/μl)
Package 2- SMART-Seq LR TSO (100 μM)
- SMART-Seq LR Oligo-dT (10 μM)
- 5X Ultra Low First-Strand Buffer
- SMARTScribe Reverse Transcriptase (100 U/μl)
- TRH (200 U/μl)
- SMART-Seq LR Primer 1 (12 μM)
- SMART-Seq LR Primer 2 (12 μM)
- RNase Inhibitor (40 U/μl)
- Nuclease-Free Water
- 10X Lysis Buffer
- Elution Buffer (10 mM Tris-HCl, pH 8.5)
- SeqAmp DNA Polymerase
- SeqAmp CB PCR Buffer (2X)
- SMART-Seq LR Index Plate (12.5 μM)
保存
Package 1:-70℃
Package 2:-20℃
10X Lysis Buffer:解凍後は4℃保存
Elution Buffer:解凍後は室温保存
(SMART-Seq LR Index Plateは12回以上凍結融解しないでください。)
Package 2:-20℃
10X Lysis Buffer:解凍後は4℃保存
Elution Buffer:解凍後は室温保存
(SMART-Seq LR Index Plateは12回以上凍結融解しないでください。)
本製品以外に必要な試薬、機器(主なもの)
下記の製品は本キット中に含まれません。下記の製品はユーザーマニュアルに記載のProtocolで使用できることが確認されています。
- サンプル調製(セルソーティング)
-
- 8-tube strips(Thermo Fisher Scientific社 Cat. No. AB0264)またはセルソーティングに適したPCR tube strips、96 well plates
- Microplate film(USA Scientific社 Cat. No. 2920-0010)for sealing tubes/plates before sorting
- Aluminum single tab foil seal(USA Scientific社 Cat. No. 2938-4100)または cap strips(Thermo Fisher Scientific社 Cat. No. AB0784/AB0850)for sealing tubes/plates after sorting
- ドライアイス(細胞の凍結用)
- (オプション)BD FACS Pre-Sort Buffer (BD Biosciences社 Cat. No. 563503)
- (オプション)10X Lysis Buffer(製品コード 635013)
- cDNA合成、PCR増幅
-
- PCRサーマルサイクラー 2台(cDNA合成用、PCR増幅用)
- Nuclease-free, PCR-grade, thin-wall PCR strips(0.2 ml PCR 8-tube strip; USA Scientific社 Cat. No.1402-4700)または同様のPCR-grade, thin-wall PCR tubes, strips, 96-well plates
- Nuclease-free, low-adhesion 1.5 ml tubes(USA Scientific社 Cat. No. 1415-2600)、DNA LoBind tubes(Eppendorf社 Cat. No. 022431021)または同様の核酸低吸着性チューブ
- Thermo Scientific Adhesive PCR Plate Seals(Thermo Fisher Scientific社 Cat. No. AB0558)for 96-well plates or cap strips(Thermo Fisher Scientific社 Cat. No. AB0784/AB0850)for 8-tube strips
- Beads精製
-
- NucleoMag NGS Clean-up and Size Select(5 ml size:製品コード 744970.5;50 ml size:製品コード 744970.50;500 ml size:製品コード 744970.500)
※NucleoMagの代わりにAMPure XP PCR purification kit(Beckman Coulter社 5 ml size:Cat. No. A63880;60 ml size:Cat. No. A63881)も使用可能 - 80%エタノール(用時調製)
- Magnetic Separation Device
〇24~96サンプルの場合:Magnetic Stand 96(Thermo Fisher Scientific社 Cat. No. AM10027)、V-bottom plates(500 μl; VWR, Cat. No. 47743-996)、adhesive PCR Plate Seals(Thermo Fisher Scientific社 Cat. No. AB0558)と組み合わせて使用
〇1.5 mlチューブ(ライブラリーのプール用)の場合:Magnetic Stand (2 tubes)(製品コード 631964)、8-tube strips(Thermo Fisher Scientific社 Cat. No. AB0264)、またはnuclease-free、PCR-grade、thin-wall PCR tubes、strips、96-well plates
- NucleoMag NGS Clean-up and Size Select(5 ml size:製品コード 744970.5;50 ml size:製品コード 744970.50;500 ml size:製品コード 744970.500)
- cDNA定量、ライブラリー定量(必要に応じて選択)
-
- High Sensitivity DNA Kit(Agilent Technologies社 Cat. No. 5067-4626)、High Sensitivity D5000 ScreenTape(Agilent Technologies社 Cat. No. 5067-5592)、または同等の高感度電気泳動法
- Quant-iT PicoGreen dsDNA Assay Kit(Thermo Fisher Scientific社 Cat. No. P11496)またはQubit dsDNA HS Assay Kit(Thermo Fisher Scientific社 Cat. No. Q32854)
- その他
-
- シングルチャンネルピペット:10 μl、20 μlおよび200 μl
- 8連、あるいは12連チャンネルピペット:10 μl
- フィルターピペットチップ:10 μl、20 μl、および200 μl
- 1.5 mlチューブ用微量遠心機
- 0.2 mlチューブまたはstrip用微量遠心機
この製品を見た人は、
こんな製品も見ています
-

ssDNA-Seq Kit
ssDNAを直接繋げる独自技術により、わずか10 pgの断片化の進んだDNAや短鎖ssDNAからでもNGS解析が可能
-
次世代シーケンス(NGS)関連試薬選択ガイド RNAシーケンス
-
PrimeSTAR® LongSeq DNA Polymerase
世界最高レベルの高特異性で超長鎖増幅(50 kb)と長鎖マルチプレックスPCRに対応
-

ロングリードRNA-Seq解析(Iso-Seq)
ロングリードシーケンサーによる網羅的な全長アイソフォームの検出
-
SMART法を用いたcDNA合成の概要
-

SMART-Seq® mRNA / SMART-Seq® mRNA LP
イルミナ社NGS装置用の超微量mRNA-Seq用ライブラリー調製キット
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。
