高正確性PCR酵素【PrimeSTARシリーズ】
クルードサンプルに対応!パワフルなMightyAmp DNA Polymeraseシリーズ
高正確性PCR酵素
PrimeSTAR®シリーズ
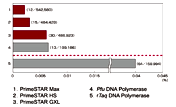
「高い正確性」と「良好な反応効率」を両立するPrimeSTAR PCR酵素シリーズは様々な分野で活躍しています。
本ページではその一部をご紹介いたします。
クルードサンプルに対応!パワフルなMightyAmp DNA Polymeraseシリーズ
高正確性PCR酵素
PrimeSTAR®シリーズ
「高い正確性」と「良好な反応効率」を両立するPrimeSTAR PCR酵素シリーズは様々な分野で活躍しています。
本ページではその一部をご紹介いたします。
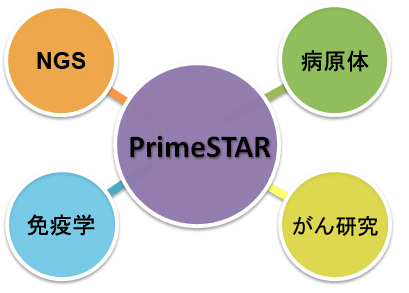
さまざまな分野での活躍(使用文献)
各分野において、PrimeSTARを使用した文献が続々と報告されています。
クルードサンプルに対応!パワフルな
MightyAmp™シリーズ
通常のPCR酵素では増幅が困難な、クルードなサンプルからでも良好に増幅できるMightyAmp DNA Polymeraseシリーズの使用例をご紹介いたします。

- PrimeSTAR HSを使用した文献:
- https://www.takarabio.com/learning-centers/pcr/citations/primestar-hs
- PrimeSTAR Maxを使用した文献:
- https://www.takarabio.com/learning-centers/pcr/citations/primestar-max
- PrimeSTAR GXLを使用した文献:
- https://www.takarabio.com/learning-centers/pcr/citations/primestar-gxl
■病原体研究での使用例
- SARS-CoV-2機能解析法の研究において、ウイルスcDNA断片の増幅にPrimeSTAR Max DNA Polymeraseを使用
Liu,G., et al.(2023). An optimized circular polymerase extension reaction-based method for functional analysis of SARS-CoV-2. Virol J. 7;20(1). - SARS-CoV-2のオミクロン株の研究において、SARS-CoV-2関連配列の増幅にPrimeSTAR GXL DNA Polymeraseを使用
Chen, D., et al.,(2023). Spike and nsp6 are key determinants of SARS-CoV-2 Omicron BA.1 attenuation. Nature, 615.143-150. -
マダニの病原性Rickettsia属菌感染率の研究において、ompB, sca4, gltA, 16S ribosomal DNA(rrs), およびompA遺伝子増幅にPrimeSTAR GXL Premix Fast, Dye plusを使用
Qi, Y., et al.,(2022). High prevalence of Rickettsia spp. in ticks from wild hedgehogs rather than domestic bovine in Jiangsu province, Eastern China. Front Cell Infect Microbiol. 12. 954785.
■がん研究での使用例
- EZH2を治療標的としたがん研究において、truncated EZH2のクローニングにPrimeSTAR Max DNA Polymeraseを使用
Mei,H., et al.(2023). Discovery of IHMT-337 as a potent irreversible EZH2 inhibitor targeting CDK4 transcription for malignancies. Signal Transduct Target Ther. 2023; 8: 18. -
p63アイソフォームTA-p63の研究において、ゲノムDNAから目的領域の増幅(0.5-2.4 kbp)にPrimeSTAR Max DNA Polymeraseを使用
Katoh, I., et al.(2023). ΔNp63 silencing, DNA methylation shifts, and epithelial-mesenchymal transition resulted from TAp63 genome editing in squamous cell carcinoma. Neoplasia, 45: 100938.
■遺伝性疾患・エピゲノム研究での使用例
- エピゲノムプロファイリングの研究において、シークエンス解析を行うためのライブラリー調製にPrimeSTAR Max DNA Polymeraseを使用
Llorens-Bobadilla,E., et al.(2023). Solid-phase capture and profiling of open chromatin by spatial ATAC. Nat Biotechnol, 41(8), 1085-1088. -
遺伝性疾患の治療アプローチの研究において、ATM断片のクローニングにPrimeSTAR GXL DNA Polymeraseを使用
Kim, J., et al.,(2023). A framework for individualized splice-switching oligonucleotide therapy. Nature, 619.828-836. - 遺伝子量補償 Dosage compensation(DC)の調節因子に関する研究において、SOA遺伝子のクローニングにPrimeSTAR GXL DNA Polymeraseを使用
Kalita et al., (2023). The sex-specific factor SOA controls dosage compensation in Anopheles mosquitoes. Nature, 623(7985).
■CRISPR・Cas9構造研究での使用例
- CRISPR–Csm複合体を用いた真核生物のRNA標的化ツールに関する研究において、培養細胞ゲノムDNAからCKB遺伝子座の増幅にPrimeSTAR GXL DNA Polymeraseを使用
Colognori, D., et al.(2023). Precise transcript targeting by CRISPR-Csm complexes. Nat. Biotechnol. 41.1256-1264. -
Cas9に近縁のIsrBの構造解析研究において、TAM(target-adjacent motif)のクローニングにPrimeSTAR Max DNA Polymeraseを使用
Hirano,S., et al.(2022). Structure of the OMEGA nickase IsrB in complex with ωRNA and target DNA. Nature, (610), 575-581.
■Long-read sequencing(PacBio)での使用例
- 長鎖PCR条件の探索研究において、FLG exon 3(12,753 bp)の増幅にPrimeSTAR GXL DNA Polymeraseを使用
Mareso, C., et al.(2023). Optimization of long-range PCR protocol to prepare filaggrin exon 3 libraries for PacBio long-read sequencing. Mol Biol Rep. 50(4).
■NGSにおけるAmplicon調製での使用例
- PrimeSTAR GXL DNA Polymeraseを用いてPKD1およびPKD2領域から2.3-10.8 kbのAmpliconを調製。常染色体優性多発性嚢胞腎の原因とされる突然変異をMiseqで解析
Tan, Adrian Y., et al. (2014). Molecular diagnosis of autosomal dominant polycystic kidney disease using next-generation sequencing. The Journal of Molecular Diagnostics, 16(2), 216-228. - CAGの3塩基繰り返し配列を含む1.5 kb断片をPrimeSTAR GXL DNA Polymeraseで増幅しライブラリ調製に使用。PacBioでlong-read sequencing解析を実施
Liu, Qian, et al. (2017). Interrogating the“unsequenceable”genomic trinucleotide repeat disorders by long-read sequencing. Genome medicine, 9(1), 65. - Neuronal mtDNA(ミトコンドリアDNA)の特定領域9 kbをPrimeSTAR GXL DNA Polymeraseで増幅、HiSeq 2500でdeep sequencingし、これまでにない高感度でmtDNAの欠失を検出した。
Nido, Gonzalo S., et al. (2018). Ultradeep mapping of neuronal mitochondrial deletions in Parkinson’s disease. Neurobiology of aging, 63, 120-127. - PrimeSTAR GXL DNA PolymeraseとTaKaRa LA Taqを用いてHLA class I領域の全長およびHLA class IIの特定領域を増幅、PacBioで配列決定し、HLA typingを行った。
Turner, Thomas R., et al. (2017). Single molecule real‐time DNA sequencing of HLA genes at ultra‐high resolution from 126 International HLA and Immunogenetics Workshop cell lines. HLA. - Red-Eared Slider Turtle(ミシシッピアカミミガメ)のAromatase遺伝子領域をPrimeSTAR GXL DNA Polymeraseでnested PCR(25 kb → 9 kb、10 kb、10 kbの3断片)。3断片からライブラリ調製し、Miseqで配列解析
Matsumoto, Yuiko, and David Crews. (2017). Genetic Polymorphisms in Aromatase (cyp19a1) Are Not Associated with Gonadal Phenotypes in Red-Eared Slider Turtle Hatchlings Developed at a Pivotal Temperature. Sexual Development, 11(3), 151-160.
クルードサンプルに対応!パワフルな
MightyAmp™シリーズ
通常のPCR酵素では増幅が困難な、クルードなサンプルからでも良好に増幅できるMightyAmp DNA Polymeraseシリーズの使用例をご紹介いたします。
■細胞・臨床検体・糞便での使用例
- DNA二本鎖切断を必要としない遺伝子編集研究において、細胞コロニーからのDNAシークエンス解析用PCR増幅にMightyAmp DNA Polymerase Ver.2を使用
Nakajima et al., Precise and efficient nucleotide substitution near genomic nick via noncanonical homology-directed repair. Genome Res. 2018 Feb; 28(2) - 遺伝性心筋症の治療法研究において、家畜豚のクローン細胞由来の遺伝子変異検出のための細胞からのダイレクトPCR増幅にMightyAmp DNA Polymerase Ver.2を使用
Matsunari et al., Pigs with δ-sarcoglycan deficiency exhibit traits of genetic cardiomyopathy. Lab Invest. 2020; 100(6): 887-899. - 臨床検体中の結核菌検出法の開発に関する研究において、臨床検体より抽出したDNAを鋳型にした結核菌トランスポゾンIS1081のPCR増幅にMightyAmp DNA Polymerase Ver.3を使用
Ren et al., Development and clinical evaluation of a CRISPR/Cas13a-based diagnostic test to detect Mycobacterium tuberculosis in clinical specimens. Front Microbiol. 2023; 14: 1117085. - 主食による腸内細菌叢への影響を調べた研究において、糞便サンプル中の16S rRNA遺伝子配列解析用のライブラリー調製にMightyAmp DNA Polymerase Ver.2を使用
Mano et al., The Effect of White Rice and White Bread as Staple Foods on Gut Microbiota and Host Metabolism. Nutrients. 2018 Sep; 10(9): 1323.
■植物検体・考古学標本での使用例
- リンドウの花の色の濃さを決定づける因子の同定した研究において、リンドウの葉より抽出されたDNAを鋳型にしたmini zinc-fingerタンパクのホモログであるGtMIF1遺伝子のPCR増幅にMightyAmp DNA Polymerase Ver.3を使用
Tasaki et al., Identification of Candidate Genes Responsible for Flower Colour Intensity in Gentiana triflora. Front Plant Sci. 2022 Jun 22:13:906879. -
考古学標本のゼラチンの動物種同定研究において、ゼラチンより抽出精製したDNAを鋳型にした様々な生物種(rabbit, fishなど)のミトコンドリア関連遺伝子(COI、ATP8、ATP6など)のPCR増幅にMightyAmp DNA Polymerase Ver.3を使用
Kuramata et al., Animal species identification utilising DNAs extracted from traditionally manufactured gelatin(Wanikawa). Heritage Science. 2022 10;183.
News
アメリカ国立癌研究所の今清水先生の研究グループが、弊社の高正確性PCR酵素PrimeSTAR Max DNA Polymeraseと高性能逆転写酵素PrimeScript RTaseを用いてRNET-seqという方法を開発し、ゲノムワイドに転写のerrorとpausingを直接検出することにはじめて成功され、論文を発表されました。
先生からは「タカラの高精度の酵素は成功のひとつのカギです」といううれしいコメントをいただいています。
今清水先生の論文は以下のリンクよりご覧いただけます。
Imashimizu, M., Takahashi, H., Oshima, T., McIntosh, C., Bubunenko, M., & Kashlev, M. (2015).
Visualizing translocation dynamics and nascent transcript errors in paused RNA polymerases in vivo.
Genome biology. 16(1), 98.
今清水先生の論文は以下のリンクよりご覧いただけます。
Imashimizu, M., Takahashi, H., Oshima, T., McIntosh, C., Bubunenko, M., & Kashlev, M. (2015).
Visualizing translocation dynamics and nascent transcript errors in paused RNA polymerases in vivo.
Genome biology. 16(1), 98.
その他
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。