【ユーザー様実施例】SMART-Seq® Single Cell Kitを⽤いた⼩細胞塊のRNA-Seqの検討
データご提供:東京大学大学院 新領域創成科学研究科 生命システム観測分野 関先生
■ 実験内容
直径が150-200 μm程度の小さな細胞塊(マウス腎臓と肝臓)を384 plateにソートしLysisした。一部を96 well plateに移し、ナノリッター分注システムdragonfly discovery(SPT Labtech社)で標準プロトコールの1/4スケールでSMART-Seq Single Cell KitによるcDNA合成、およびNextera XT DNA Library Preparation Kit(イルミナ社)でライブラリー調製を行った。NovaSeq 6000(イルミナ社)でシーケンスを行った。■ 実験の流れ
SMART-Seq Single Cell Kit(SSsc) のLysis Mixを調製し、384 plateに20 μl / wellずつ分注する
↓
そこへ直径150-200 μmの小さな細胞塊(マウス腎臓と肝臓)をソートする
↓
72℃ 3 minインキュベーション
↓
384 plate から96 well plateにLysateを3.2 μlずつ移す
↓
dragonfly discoveryでSSscのRT Mixを1.875 μl / wellずつ分注する
↓
42℃ 180 min、70℃ 10 minインキュベーション後、 4℃ ホールド
↓
dragonfly discoveryでSSscのPCR Mixを15 μl / wellずつ分注する
↓
95℃ 1 min、 (95℃ 10 sec、65℃ 30 sec、68℃ 3 min) ×14 cycle、72℃ 10 min、4℃ホールドでPCRを行う
↓
マニュアルでAMPure XP精製を行う(溶出ボリュームは15 μl)
↓
Nextera XT DNA Library Preparation Kitでライブラリーを調製する
↓
NovaSeq 6000のSPフローセルでシーケンスを行う
↓
そこへ直径150-200 μmの小さな細胞塊(マウス腎臓と肝臓)をソートする
↓
72℃ 3 minインキュベーション
↓
384 plate から96 well plateにLysateを3.2 μlずつ移す
↓
dragonfly discoveryでSSscのRT Mixを1.875 μl / wellずつ分注する
↓
42℃ 180 min、70℃ 10 minインキュベーション後、 4℃ ホールド
↓
dragonfly discoveryでSSscのPCR Mixを15 μl / wellずつ分注する
↓
95℃ 1 min、 (95℃ 10 sec、65℃ 30 sec、68℃ 3 min) ×14 cycle、72℃ 10 min、4℃ホールドでPCRを行う
↓
マニュアルでAMPure XP精製を行う(溶出ボリュームは15 μl)
↓
Nextera XT DNA Library Preparation Kitでライブラリーを調製する
↓
NovaSeq 6000のSPフローセルでシーケンスを行う
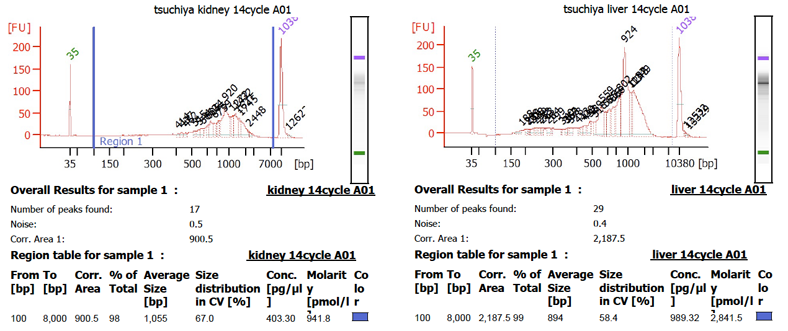
■ 全長cDNAの電気泳動結果
各ウェルにおける増幅されたcDNAを測定したところ、ほとんどのウェルでcDNAの増幅が確認できました。

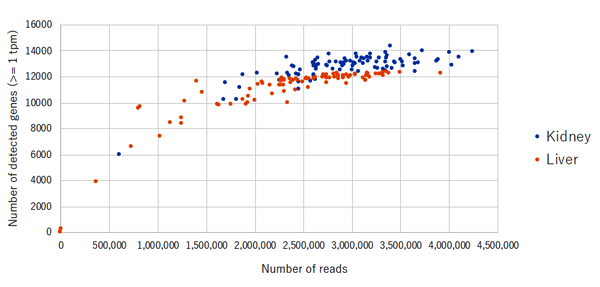
■ シーケンス結果
NovaSeq 6000のSPフローセル1レーンで192サンプルをシーケンスし、発現量集計を行ったところ、250万リード以上読めていたサンプルに関しては11,000-14,000遺伝子が検出されていました。

| 肝臓 | 腎臓 | |
|---|---|---|
| 平均リード数 | 2,349,517 | 2,939,182 |
| 平均検出した遺伝子数 | 10,799 | 12,711 |
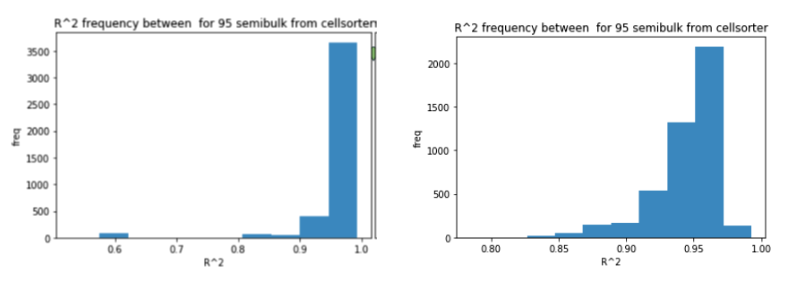
■ 細胞塊の間で比較
肝臓と腎臓の組織間ではR2値が0.5以下であったのに対して(データは示さず)、同じ組織間ではほとんどの組み合わせでは0.9以上でしたので、かなり再現性の高いデータが取れていました。