分子バーコードによりさらに信頼性が向上した超微量mRNA-Seq用ライブラリー調製キット
- Total RNA 10 pg~100 ng、または1~1,000細胞の超微量サンプルからイルミナ社NGS装置用の高品質mRNA-Seq用ライブラリー調製が可能
- 分子バーコード(UMI)の採用により高精度な遺伝子発現/転写産物カウントを実現。従来製品より信頼性が向上
- サンプルロスやハンドリングエラーを最小限に抑制するシンプルな操作ステップ
- Unique Dual Index Kit(別売り)の使用により、インデックスホッピングの影響を軽減
- ユーザーフレンドリーな解析ツール「Cogent NGS」が無料で利用可能
※色文字での表示は、キャンペーン価格およびそのキャンペーン期間です。
※本キットにはIndexが含まれていません。Unique Dual Index Kit(製品コード 634752/634753/634754/634755/634756)を別途ご購入ください。
※本キットにはIndexが含まれていません。Unique Dual Index Kit(製品コード 634752/634753/634754/634755/634756)を別途ご購入ください。
カートにいれる
WEB会員ログインが必要です
製品説明
SMART-Seq mRNA LP (with UMIs)は、total RNA 10 pg~100 ng、または1~1,000細胞の超微量サンプルからイルミナ社NGS装置用の高品質mRNA-Seq用ライブラリーが調製可能なキットです。高品質・高効率なcDNA合成技術SMART法(Switching Mechanism at 5' End of RNA Template)で使用するSMART-Seq用オリゴに分子バーコード(UMI)を搭載することにより、シーケンスエラーやPCR増幅バイアスを抑制し、完全長mRNA-Seqにおける遺伝子発現定量解析の精度が既存製品に比べて向上しています。また、Unique Dual Index Kit(製品コード 634752他、別売り)を用いたマルチプレックスシーケンスにより、インデックスホッピングの影響を軽減することができます。
ライブラリー調製においては、フラグメンテーションやステムループのアダプターライゲーションをシングルチューブで行う当社独自のシングルチューブワークフローにより、ハンズオンタイムの短縮に加え、操作ミスやサンプルロスを抑制します。 また、データ解析には、ユーザーフレンドリーな解析ツール「Cogent NGS」が無料で利用可能です。
Cogent NGSソフトウェアは下記ページからダウンロードしてください。
Cogent NGS Discovery Software(takarabio.com)
Cogent NGS Analysis Pipeline(takarabio.com)
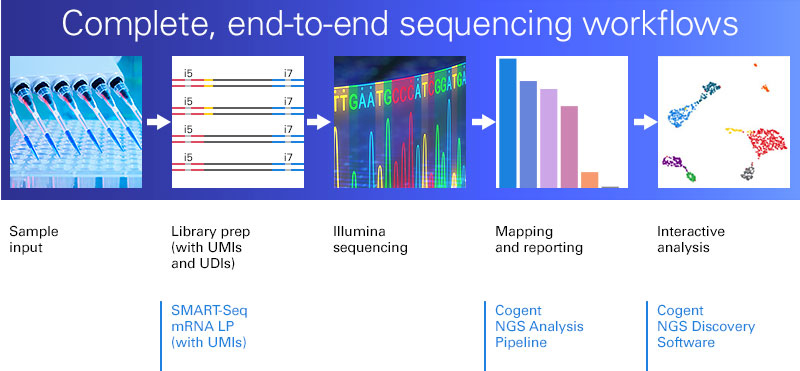
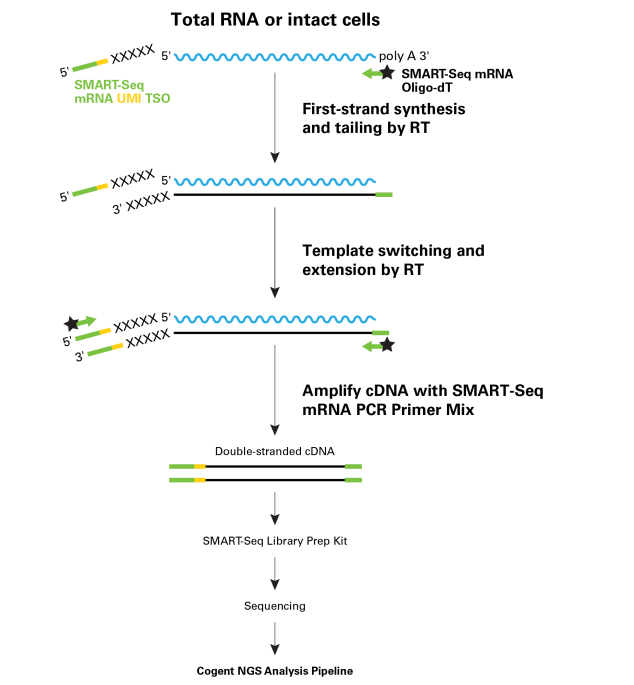
図1. 分子バーコード(UMI)を搭載したSMART法のフロー
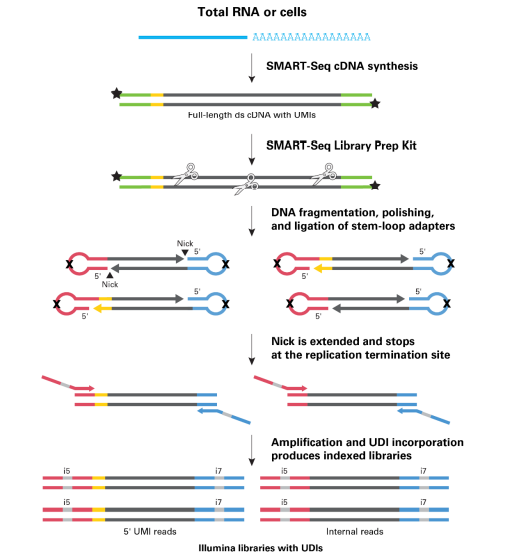
図2. SMART-Seq Library Prep Kitによるライブラリー調製フロー
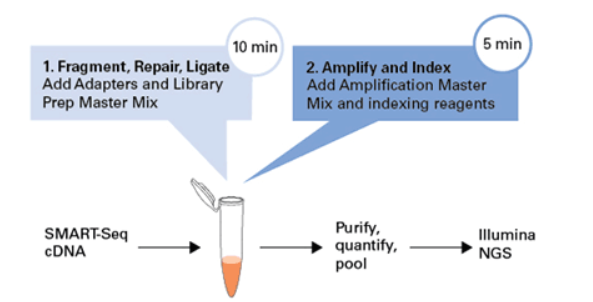
図3. シングルチューブワークフローによるライブラリー調製
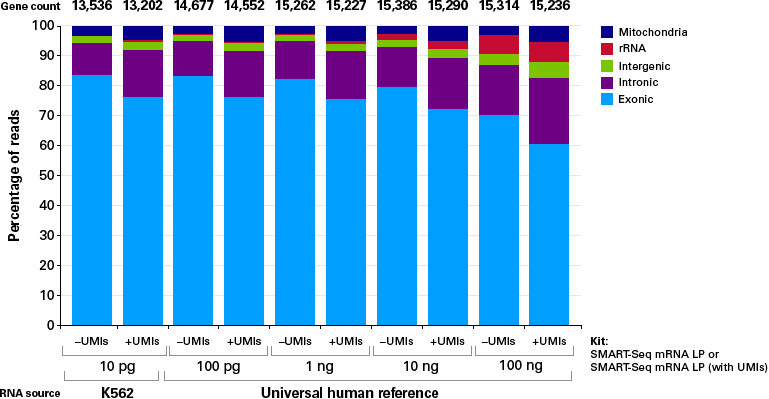
図4. UMIの有無とRNAインプット量の違いによる遺伝子カウント数とリード分布
SMART-Seq mRNA LP(with UMIs)とSMART-Seq mRNA LPを使用し、K562 RNA(10 pg)またはUniversal human reference RNA(UHR)(100 pg、1 ng、10 ng、100 ng)からライブラリーを調製した。2つのライブラリー調製キットから、ほぼ同程度の遺伝子カウントおよびリード分布が得られた。
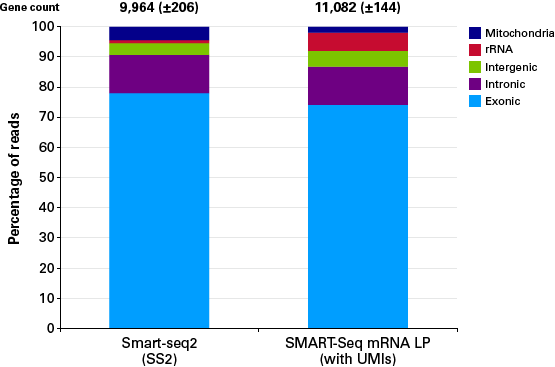
図5. Smart-seq2(SS2)法との比較
SMART-Seq mRNA LP (with UMIs)またはSMART-Seq2(SS2)法により、10 pgのUniversal human reference RNA(UHR)からcDNA合成とライブラリー調製を行った。SS2法では、Nextera XT DNA Library Prep Kit(イルミナ社)を用いてライブラリーを調製した。ライブラリーをプールしてシーケンスを行い、120万リードにダウンサンプリングした後のリード分布は、2つの手法でそれぞれ同等の結果を示し、遺伝子カウントは、SMART-Seq mRNA LP (with UMIs)でより多く得られた。
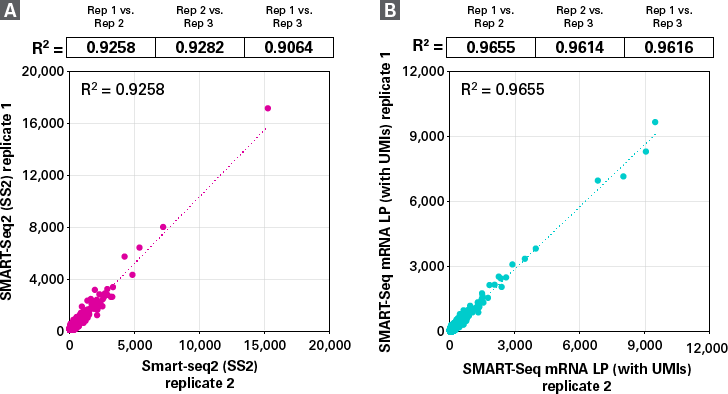
図6. レプリケートペアの回帰分析
リードカウントの絶対数を算出し、ソフトウェアCogent APを用いて、各レプリケートペアについて回帰分析を行った。R2値はSS2法よりもSMART-Seq mRNA LP (with UMIs)の方が一貫して高い値を示した。
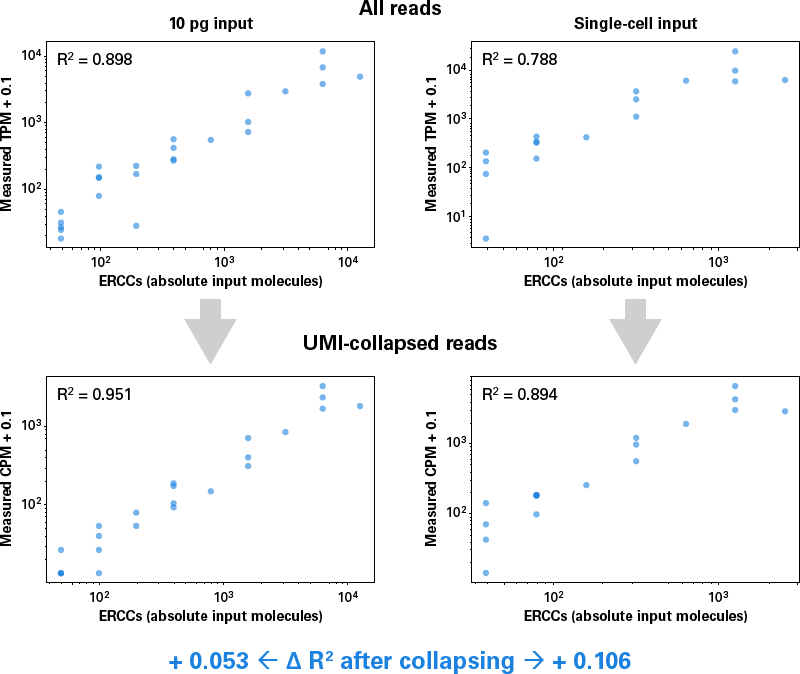
図7. UMIによるRNAカウントの正確性の向上
10 pgのインプットRNAまたはシングルセルから精製したRNAに、ERCC control RNAをスパイクし、SMART-Seq mRNA LP (with UMIs)を用いてライブラリーを調製した。シーケンス後、CogentAPを用いて、ERCC spike-in 転写産物のTPM(Transcripts per million)およびCPM(Count per million)をインプットした分子の絶対数に対しプロットした。UMIを使用した解析結果を組み合わせることで、回帰分析においてR2値が顕著に増加することが示された。
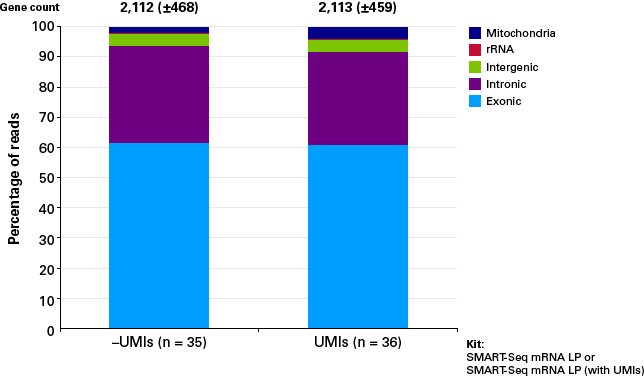
図8. シングルセルワークフローにおけるUMIの影響
末梢血単核細胞(PBMC)のシングルセルをSMART-Seq mRNA LPまたはSMART-Seq mRNA LP (with UMIs)を用いてライブラリー調製した。ライブラリーはプールしてシーケンスを行い、解析用に90万リードにダウンサンプリングした。UMIの有無に関わらず遺伝子カウントのリード分布が同等であることから、UMIを含むことでデータ品質が損なわれることはないということが示された。
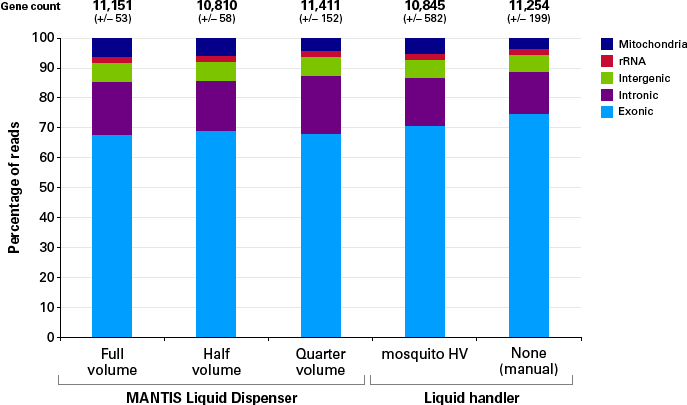
図9. 自動分注装置の影響
SMART-Seq mRNA LP (with UMIs)でのライブラリー調製に、分注装置MANTIS Liquid Dispenserを用いてMouse brain control RNA(10 pg、n=8)を全量、1/2量、1/4量インプットした。ライブラリーはプールしてシーケンスを行い、130万リードにダウンサンプリングした。また、同一のRNAの1/8量をナノリッター分注システムmosquito HV liquid handlerと、ベンチトップで手動でインプットした。これらのライブラリーもプールしてシーケンスを行い、62万リードまでダウンサンプリングした。すべての解析はCogentAPを用いて行った。
Control RNAの分注方法、および分注量に関わらず、同様の遺伝子カウントとリード分布を得られたことから、自動分注装置によるシーケンス結果への影響はないことが示された。
ライブラリー調製においては、フラグメンテーションやステムループのアダプターライゲーションをシングルチューブで行う当社独自のシングルチューブワークフローにより、ハンズオンタイムの短縮に加え、操作ミスやサンプルロスを抑制します。 また、データ解析には、ユーザーフレンドリーな解析ツール「Cogent NGS」が無料で利用可能です。
Cogent NGSソフトウェアは下記ページからダウンロードしてください。
Cogent NGS Discovery Software(takarabio.com)
Cogent NGS Analysis Pipeline(takarabio.com)


図1. 分子バーコード(UMI)を搭載したSMART法のフロー

図2. SMART-Seq Library Prep Kitによるライブラリー調製フロー

図3. シングルチューブワークフローによるライブラリー調製

図4. UMIの有無とRNAインプット量の違いによる遺伝子カウント数とリード分布
SMART-Seq mRNA LP(with UMIs)とSMART-Seq mRNA LPを使用し、K562 RNA(10 pg)またはUniversal human reference RNA(UHR)(100 pg、1 ng、10 ng、100 ng)からライブラリーを調製した。2つのライブラリー調製キットから、ほぼ同程度の遺伝子カウントおよびリード分布が得られた。

図5. Smart-seq2(SS2)法との比較
SMART-Seq mRNA LP (with UMIs)またはSMART-Seq2(SS2)法により、10 pgのUniversal human reference RNA(UHR)からcDNA合成とライブラリー調製を行った。SS2法では、Nextera XT DNA Library Prep Kit(イルミナ社)を用いてライブラリーを調製した。ライブラリーをプールしてシーケンスを行い、120万リードにダウンサンプリングした後のリード分布は、2つの手法でそれぞれ同等の結果を示し、遺伝子カウントは、SMART-Seq mRNA LP (with UMIs)でより多く得られた。

図6. レプリケートペアの回帰分析
リードカウントの絶対数を算出し、ソフトウェアCogent APを用いて、各レプリケートペアについて回帰分析を行った。R2値はSS2法よりもSMART-Seq mRNA LP (with UMIs)の方が一貫して高い値を示した。

図7. UMIによるRNAカウントの正確性の向上
10 pgのインプットRNAまたはシングルセルから精製したRNAに、ERCC control RNAをスパイクし、SMART-Seq mRNA LP (with UMIs)を用いてライブラリーを調製した。シーケンス後、CogentAPを用いて、ERCC spike-in 転写産物のTPM(Transcripts per million)およびCPM(Count per million)をインプットした分子の絶対数に対しプロットした。UMIを使用した解析結果を組み合わせることで、回帰分析においてR2値が顕著に増加することが示された。

図8. シングルセルワークフローにおけるUMIの影響
末梢血単核細胞(PBMC)のシングルセルをSMART-Seq mRNA LPまたはSMART-Seq mRNA LP (with UMIs)を用いてライブラリー調製した。ライブラリーはプールしてシーケンスを行い、解析用に90万リードにダウンサンプリングした。UMIの有無に関わらず遺伝子カウントのリード分布が同等であることから、UMIを含むことでデータ品質が損なわれることはないということが示された。

図9. 自動分注装置の影響
SMART-Seq mRNA LP (with UMIs)でのライブラリー調製に、分注装置MANTIS Liquid Dispenserを用いてMouse brain control RNA(10 pg、n=8)を全量、1/2量、1/4量インプットした。ライブラリーはプールしてシーケンスを行い、130万リードにダウンサンプリングした。また、同一のRNAの1/8量をナノリッター分注システムmosquito HV liquid handlerと、ベンチトップで手動でインプットした。これらのライブラリーもプールしてシーケンスを行い、62万リードまでダウンサンプリングした。すべての解析はCogentAPを用いて行った。
Control RNAの分注方法、および分注量に関わらず、同様の遺伝子カウントとリード分布を得られたことから、自動分注装置によるシーケンス結果への影響はないことが示された。
内容
SMART-Seq mRNA LP (with UMIs)
*本キットの単品販売はありません。
- SMART-Seq mRNA (with UMIs)*
- Box 1(Store at -70℃)
- Box 2(Store at -20℃)
Control Total RNA(1 μg/μl)
SMART-Seq mRNA UMI TSO(50 μM)
SMART-Seq mRNA PCR Primer Mix
5× Ultra Low First Strand Buffer
SMARTScribe Reverse Transcriptase(100 U/μl)
SMART-Seq mRNA Oligo-dT(10 μM)
GTP(20 mM)
RNase Inhibitor(40 U/μl)
Nuclease-Free Water
10× Lysis Buffer
Elution Buffer(10 mM Tris-Cl, pH8.5)
SeqAmp DNA Polymerase
SeqAmp CB PCR Buffer(2×)
- SMART-Seq Library Prep Kit*
10× FE
FE Dilution Buffer
Lib Prep Buffer
Lib Prep Enzyme
Rxn Enhancer
Stem Loop Adapters
Amplification Buffer
PrimeSTAR HS DNA Polymerase(5 U/μl)
Nuclease-Free Water
FE Dilution Buffer
Lib Prep Buffer
Lib Prep Enzyme
Rxn Enhancer
Stem Loop Adapters
Amplification Buffer
PrimeSTAR HS DNA Polymerase(5 U/μl)
Nuclease-Free Water
保存
SMART-Seq mRNA (with UMIs)
SMART-Seq Library Prep Kit:-20℃
(10× Lysis Buffer:融解後は4℃保存、Elution Buffer:融解後は室温保存)
- Control Total RNA:-70℃
- その他のコンポーネント:-20℃
SMART-Seq Library Prep Kit:-20℃
(10× Lysis Buffer:融解後は4℃保存、Elution Buffer:融解後は室温保存)
本製品以外に必要な試薬、機器(主なもの)
- Index Kit
- Unique Dual Index Kit (1-24)(製品コード 634756)
UDI No. U001-U024 - Unique Dual Index Kit (1-96)(製品コード 634752)
UDI No, U001-U096 - Unique Dual Index Kit (97-192)(製品コード 634753)
UDI No, U097-U192 - Unique Dual Index Kit (193-288)(製品コード 634754)
UDI No, U193-U288 - Unique Dual Index Kit (289-384)(製品コード 634755)
UDI No, U289-U384
- Unique Dual Index Kit (1-24)(製品コード 634756)
- PCR増幅およびバリデーション
- First-Strand cDNA 合成専用のPCRサーマルサイクラー
- Double-Strand cDNA 増幅専用のPCRサーマルサイクラー
- Nuclease-free thin-wall PCR tube(0.2 ml;USA Scientific社 Code 1402-4700)または同等のNuclease-free thin-wall PCR tube stripsまたは96-well Plate
- Nuclease-free low adhesion 1.5 ml tube(USA Scientific社 Code 1415-2600)またはDNA LoBind tube(Eppendorf社 Code.022431021)または同等のnucleic acid low-binding tubes
- 96-well Plate用粘着シール Thermo Scientific Adhesive PCR Plate Seals(Thermo Fisher Scientific社 Cat. No. AB0558)または 8 tube strips用Cap strips(Thermo Fisher Scientific社 Cat. No. AB0784/AB0850)
- セルソーティング
- 8-tube strips(Thermo Fisher Scientific社 Cat. No. AB0264)またはセルソーティングに適したPCR tube strips, 96-well plates
- (オプション)BD FACS Pre-Sort Buffer(BD社 Cat. No. 563503)
- チューブ・プレート用クーリングブロック(-20℃で保存してあらかじめ冷やしておく)
- Adhesive PCR Plate Foils(Thermo Fisher Scientific社 Cat. No. AB-0626)
- ドライアイス(細胞の凍結用)
- (オプション)SMART-Seq HT Kit Lysis Components(製品コード 634439)First-Strand cDNA 合成専用のPCRサーマルサイクラー
- SPRI Bead精製
- NucleoMag NGS Clean-up and Size Select(5 ml size:製品コード 744970.5;50 ml size:製品コード 744970.50;500 ml size:製品コード 744970.500)
※NucleoMagの代わりにAMPure XP PCR purification kit(Beckman Coulter社 5 ml size:Cat. No. A63880;60 ml size:Cat. No. A63881)も使用可能 - Magnetic Separation Device 12~24サンプル用
- 80% Ethanol(必ず用時調製すること)
0.2 mlチューブ用マグネットスタンド 0.2 mlチューブまたはストリップ用遠心機24~96サンプル用
96-well フォーマット用:Magnetic Stand 96(Thermo Fisher Scientific社 Cat. No. AM10027)など1.5 ml用(シーケンス用ライブラリーをプールする場合に必要)
Magnetic Stand(製品コード 631964)または NEB Magnetic Separation Rack (NEB社 Cat. No. 1506S)
8 tube strips(Thermo Fisher Scientific社 Cat. No. AB0264)またはPCRラックに固定されNuclease-freeのPCR tube strips、またはセルソーターで動作確認済みの96-well Plate
- NucleoMag NGS Clean-up and Size Select(5 ml size:製品コード 744970.5;50 ml size:製品コード 744970.50;500 ml size:製品コード 744970.500)
- その他
- シングルチャンネルピペット:10 μl、20 μl、200 μlおよび1,000 μl
- 8連チャンネルピペット:20 μl、200 μl
- フィルターピペットチップ:10 μl、20 μl、200 μlおよび1,000 μl
- 1.5 mlチューブ用微量遠心機
- 0.2 mlチューブまたはstrip用微量遠心機
- 96ウェル冷却ラック(IsoFreeze PCR Rack, MIDSCI社 Cat. No. 5640-T4, 96-Well Aluminum Block, Light Labs社 Cat. No. A-7079など)
Technical Notes
超微量RNAインプットからのライブラリー作製について―Nextera XTキットとの性能比較(英語)
RNA-seqを成功させるためのコツ(英語)
SSv4とSMARTer Target RNA Capture for Illuminaを組み合わせた完全長cDNAの獲得(英語)
SMART-Seq® mRNA LPキットによるSNP、アイソフォーム、および融合遺伝子の検出(英語)
2種類のscRNA-seqアプローチを組み合わせた脳の進化と発達メカニズム解明(英語)
遺伝子アイソフォーム発現パターン空間解析(英語)
シングルセルとバルクmRNA-seqの実施による肝線維症メカニズムの解明(英語)
わずか1~20個の細胞からの組織の高解像度プロファイリング(英語)
RNA-seqのヒント(英語)
がんアプリケーションにおけるシングルセル解析(英語)
この製品を見た人は、
こんな製品も見ています
-

SMART-Seq® mRNA / SMART-Seq® mRNA LP
イルミナ社NGS装置用の超微量mRNA-Seq用ライブラリー調製キット
-
次世代シーケンス(NGS)関連試薬選択ガイド RNAシーケンス
-

SMART-Seq® Human BCR (with UMIs)
ヒトBCR H鎖/L鎖の可変領域の完全長シーケンス
-

Unique Dual Index Kits
イルミナ社NGSライブラリー調製キット用インデックスキット
-

SMART-Seq® mRNA HT/SMART-Seq® mRNA HT LP
低価格・簡便操作の全長mRNA解析用ライブラリー調製キット(シングルセル or 超微量RNA)
-
SMART-Seq® mRNA Single Cell / SMART-Seq® mRNA Single Cell LP
シングルセル解析に最適化されたイルミナ社NGS装置用mRNA-Seq解析
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。

