UMI搭載!マウスTCRα/β V(D)J可変領域の完全長シーケンスが可能
- イルミナ社次世代シーケンサー用マウスT細胞受容体(TCR)のレパトア解析ライブラリー調製キット
- 脾臓/T細胞total RNA 1 ng~、全血/骨髄/胸腺total RNA 10 ng~、T細胞100個から調製可能
- 分子バーコード(UMI)やUnique Dual Indexの利用で、より正確なNGS解析が可能
- CDR3領域のみでもシーケンスでき、シーケンスコストの削減も可能
※色文字での表示は、キャンペーン価格およびそのキャンペーン期間です。
※ 本キットにはIndexが含まれていません。Unique Dual Index Kit(製品コード634752/634753/634754/634755/634756)を別途ご購入ください。
※ シーケンスを行う際には、サンプルにControl DNA(PhiX)を加えて解析してください。
※ 本キットにはIndexが含まれていません。Unique Dual Index Kit(製品コード634752/634753/634754/634755/634756)を別途ご購入ください。
※ シーケンスを行う際には、サンプルにControl DNA(PhiX)を加えて解析してください。
カートにいれる
WEB会員ログインが必要です
製品説明
SMART-Seq Mouse TCR (with UMIs)は、イルミナ社次世代シーケンサーを用いたマウスT細胞受容体(TCR)のレパトア解析用ライブラリー調製キットです。本製品はSMARTテクノロジー(Switching Mechanism at 5’ End of RNA Template)と5’ RACE法の応用により、マウスTRA/TRB遺伝子の完全長V(D)J可変領域を得ることが可能です。
本製品は下記表に示す様々なRNAサンプル(RIN>7)から高品質なライブラリーを調製することができ、TCR α鎖とβ鎖両方の多様性情報を得られます。また本製品は分子バーコード(Unique Molecular Identifier : UMI)を付加することにより、PCR duplicatesやシーケンスエラーを取り除き、より正確で信頼性の高いNGS解析が可能です。さらに、Unique Dual Index Kit(別売り)と組み合わせることでインデックスホッピングによる他サンプルライブラリー由来のリードコンタミネーションを防止でき、最大384サンプルのマルチプレックス解析に対応可能です。
本製品は下記表に示す様々なRNAサンプル(RIN>7)から高品質なライブラリーを調製することができ、TCR α鎖とβ鎖両方の多様性情報を得られます。また本製品は分子バーコード(Unique Molecular Identifier : UMI)を付加することにより、PCR duplicatesやシーケンスエラーを取り除き、より正確で信頼性の高いNGS解析が可能です。さらに、Unique Dual Index Kit(別売り)と組み合わせることでインデックスホッピングによる他サンプルライブラリー由来のリードコンタミネーションを防止でき、最大384サンプルのマルチプレックス解析に対応可能です。
| サンプルタイプ | インプット量 |
| マウス脾臓、あるいはT細胞由来のtotal RNA | 1 ng~1 μg |
| マウス全血、骨髄、胸腺由来のtotal RNA | 10 ng~1 μg |
| 精製済みマウスT細胞 | 100~10,000細胞 |
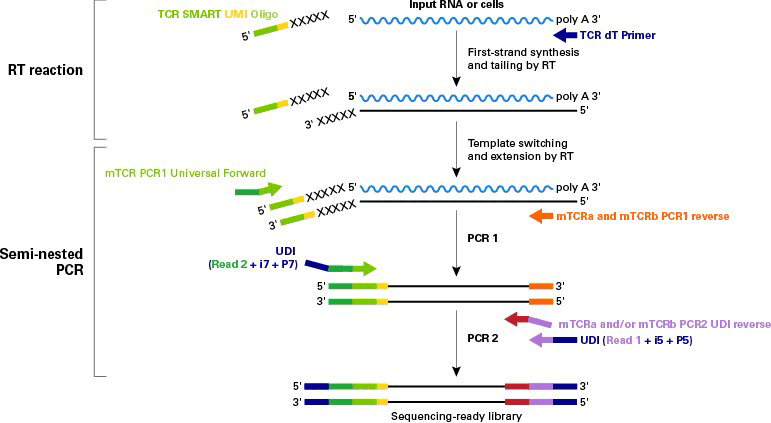
図1. SMART-Seq Mouse TCR (with UMIs)を用いたライブラリー調製の概要
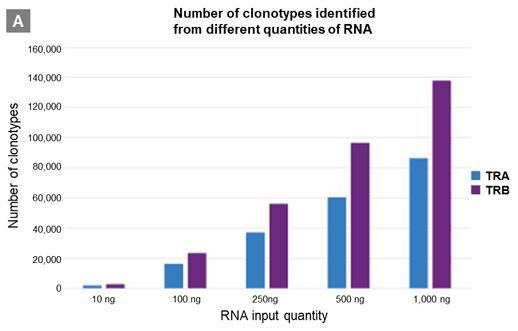
パネルA:マウス脾臓RNAのインプット量を変えてTCRα鎖、β鎖からそれぞれライブラリーを調製した。シーケンスはイルミナ社NextSeqを用い2×150 bpで行い、シーケンス結果は~1.5×107リードまでダウンサンプリングした。
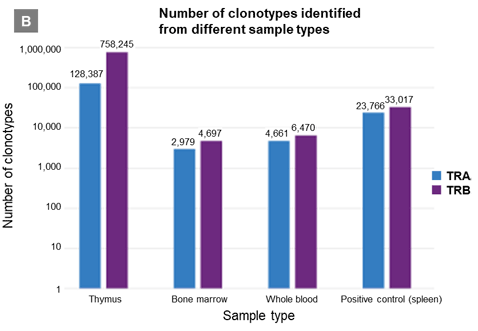
パネルB:様々なサンプルタイプから200 ngのRNAを単離しライブラリーを調製した。シーケンスはパネルA同様NextSeq、2×150 bpで行い、シーケンス結果は2.0×107リードまでダウンサンプリングした。
パネルA、Bそれぞれの棒グラフはRNAのインプット量やサンプルタイプにおけるクロノタイプカウント数を表しており、広範なインプット量とサンプルタイプに対応していることが示された。
TRA=TCR α鎖、TRB=TCR β鎖
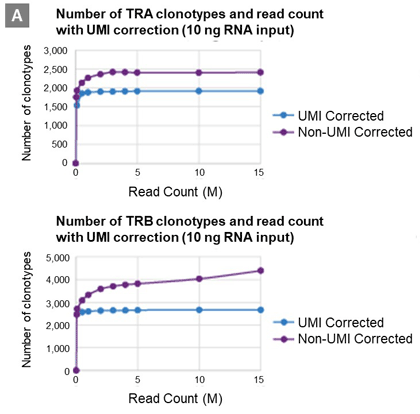
パネルA:10 ngマウス脾臓RNAからTCR α鎖とβ鎖のライブラリーを調製した。異なるシーケンスデプスでのTRAとTRBのクロノタイプカウントについて、UMIで補正した結果を青で、補正をしていない結果を紫で示した。
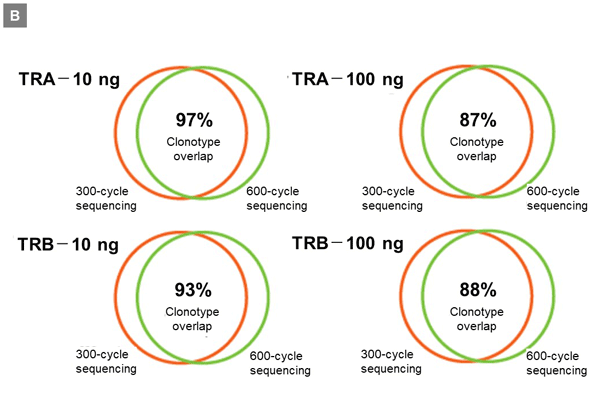
パネルB:TRA、TRBそれぞれのライブラリーを異なるサイクルでシーケンスした際のクロノタイプの重複をベン図で示したところ、TRAのライブラリーでは87%以上、TRBのライブラリーでは88%以上のクロノタイプが重複していた。
これらのことからUMIやUDIにより正確性と再現性の高い結果が得られることが示された。
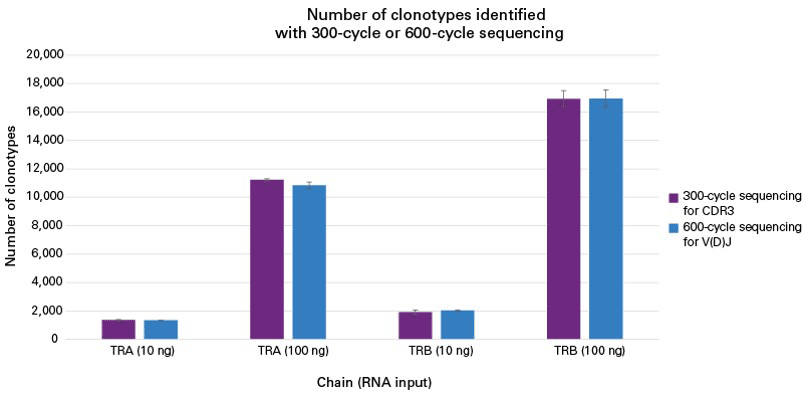
図4. シーケンスリード長の柔軟性評価
本製品がV(D)J領域の完全長シーケンスとCDR3領域のみのシーケンスに対応可能かを評価した。
10 ng、100 ngのマウス脾臓RNAからライブラリーを調製し、イルミナ社のMiSeqを用いてCDR3領域のシーケンスを2×150 bp(300サイクル)で、V(D)J領域を2×300 bp(600サイクル)で行い、1×106リードまでダウンサンプリングした。
TRAとTRBのクロノタイプカウントを棒グラフで示しており、V(D)J領域の完全長シーケンスとCDR3領域のみのシーケンスで同程度のクロノタイプカウントを得られたことから、どのイルミナ社シーケンスフォーマットでも完全長V(D)J領域、あるいはCDR3領域のみのシーケンスができることが示された。
内容
SMART-Seq Mouse TCR (with UMIs)(製品コード 634815/634816/634814)
Box1
Box2
Box1
- Control RNA (1 μg/μl)
- TCR SMART UMI Oligo (48 μM)
Box2
- 10×Lysis Buffer
- 5×Ultra Low First-Strand Buffer
- TCR dT Primer (12 μM)
- SMARTScribe Reverse Transcriptase (100 U/μl)
- RNase Inhibitor (40 U/μl)
- Elution Buffer(10 mM)
- Nuclease-Free Water
- PrimeSTAR GXL Premix (2×)
- mTCR PCR1 Universal Forward (12 μM)
- mTCRa PCR1 Reverse (12 μM)
- mTCRb PCR1 Reverse (12 μM)
- mTCRa PCR2 UDI Reverse (12 μM)
- mTCRb PCR2 UDI Reverse (12 μM)
保存
Box1:-70℃
Box2:-20℃
(10×Lysis Buffer:融解後は4℃保存、Elution Buffer:融解後は室温保存)
Box2:-20℃
(10×Lysis Buffer:融解後は4℃保存、Elution Buffer:融解後は室温保存)
本製品以外に必要な試薬、機器(主なもの)
下記の製品は本キット中に含まれません。下記の製品はユーザーマニュアルに記載のProtocolで使用できることが確認されています。
Index Kit
PCR増幅およびバリデーション
Beads精製
その他
Index Kit
- Unique Dual Index Kit (1-24)(製品コード 634756)
UDI No. U001-U024 - Unique Dual Index Kit (1-96)(製品コード 634752)
UDI No, U001-U096 - Unique Dual Index Kit (97-192)(製品コード 634753)
UDI No, U097-U192 - Unique Dual Index Kit (193-288)(製品コード 634754)
UDI No, U193-U288 - Unique Dual Index Kit (289-384)(製品コード 634755)
UDI No, U289-U384
PCR増幅およびバリデーション
- First-strand cDNA合成専用のPCRサーマルサイクラー
- ライブラリー増幅専用のPCRサーマルサイクラー
- Agilent 2100 Bioanalyzer:Agilent High Sensitivity DNA Kit(Agilent Technologies, Cat. No. 5067-4626)、Agilent DNA 1000 Kit(Agilent Technologies, Cat. No. 5067-1504)、あるいはThe Agilent TapeStation (HSD1000/HSD5000) 、Agilent Fragment Analyzerなど
- Qubit dsDNA HS Kit(Thermo Fisher Scientific社 Cat. No. Q32851)
- Nuclease-free thin-wall PCR tube(0.2 ml;USA Scientific社 Cat. No. 1402-4700)
- Nuclease-free low-adhesion 1.5 ml tubes(USA Scientific社 Cat No. 1415-2600)または、DNA LoBind tubes(Eppendorf社 Cat. No. 022431021)
Beads精製
- NucleoMag NGS Clean-up and Size Select(5 ml size:製品コード 744970.5;50 ml size:製品コード 744970.50;500 ml size:製品コード 744970.500)
※NucleoMagの代わりにAMPure XP PCR purification kit(Beckman Coulter社 5 ml size:Cat. No. A63880;60 ml size:Cat. No. A63881)も使用可能 - Magnetic Separation Device
0.2 mlチューブ用マグネットスタンド
96-well plate:Magnetic Stand 96(Thermo Fisher Scientific社 Cat. No. AM10027) - 100%エタノール(molecular biology grade)
- 96-well plate用低速遠心機
その他
- シングルチャンネルピペット:10 μl、20 μlおよび200 μl
- 8連チャンネルピペット:20 μl、200 μl
- フィルターピペットチップ:2 μl、20 μlおよび200 μl
- 1.5 mlチューブ用微量遠心機
- 0.2 mlチューブまたはstrip用微量遠心機
この製品を見た人は、
こんな製品も見ています
-

SMARTer® Human scTCR a/b Profiling Kit
シングルセルからのヒトTCRレパトア解析用キット
-
TCR/BCRレパトア解析
定量性に優れた低バイアスなレパトア解析
-

SMART-Seq® Human BCR (with UMIs)
ヒトBCR H鎖/L鎖の可変領域の完全長シーケンス
-

Unique Dual Index Kits
イルミナ社NGSライブラリー調製キット用インデックスキット
-

SMART-Seq® Mouse BCR (with UMIs)
UMI搭載!マウスBCR H鎖/L鎖 V(D)J可変領域の完全長シーケンスが可能
-

SMART-Seq® Human TCR (with UMIs)
UMI搭載!ヒトTCRα/β V(D)J可変領域の完全長シーケンスが可能
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。
