- CRISPR/Casのノックアウトを利用した関連遺伝子特定のためのスクリーニングシステム
- Cas9とsgRNAの導入にレンチウイルスを使用しており幅広い細胞で効率の良い導入が可能
- プール型なので簡単に網羅的な解析が可能
- 約19,000種類の遺伝子に対し、オフターゲットを考慮した4種類のsgRNA(約76,000種類)が設計済
- ウイルス作製に必要な試薬が一式揃ったオールインワンタイプ。しかも簡単!
WEB会員ログインが必要です
製品説明
Guide-it CRISPR Genome-Wide sgRNA Library NGS Analysis Kit(製品コード 632647)は、スクリーニングを行った細胞、もしくはコントロール細胞(非スクリーニング細胞)からゲノムDNAを抽出してNGS解析用のライブラリーを調製するためのキットで、ゲノムDNA精製キット、イルミナ社NGS装置用ライブラリー調製プライマー、PCR酵素などが入っている。
Guide-it CRISPR Genome-Wide Library PCR Kit(製品コード 632651)は、NGS装置用ライブラリー調製プライマーやPCR酵素などが入っており、1キットでPCR 20回分の試薬が含まれる。尚、Guide-it CRISPR Genome-Wide sgRNA Library NGS Analysis Kitは、Guide-it CRISPR Genome-Wide Library PCR KitとゲノムDNA精製キット(NucleoBond CB 500)とPCRクリーンアップ用キット(NucleoSpin Gel and PCR Clean-up)のセット品である。
sgRNA配列と対応する遺伝子情報をまとめたエクセルベースのデータは、下記サイトにアクセスしダウンロードできる。
https://go.takarabio.com/US-GFE-LP-sgRNA-Library_data-download_LP-SignUp.html
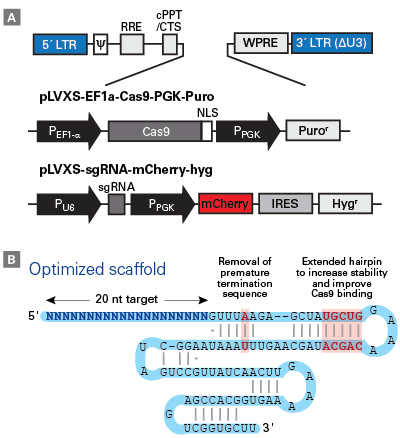
- Panel A. pLVXS-EF1a-Cas9-PGK-PuroおよびpLVXS-sgRNA-mCherry-hyg vectorのマップと骨格
- これらのベクターはSIN(self inactivating)型である。pLVXS-sgRNA-mCherry-hyg Vectorは、IRESバイシストロニック発現カセットから発現されたmCherryマーカーおよびハイグロマイシンマーカーの両方を含む。
- Panel B. 最適化されたScaffold配列
- sgRNAは、ヒトU6プロモーターにより発現される。最適化されたScaffold配列により、Cas9への高い相互作用と編集効率を示す。
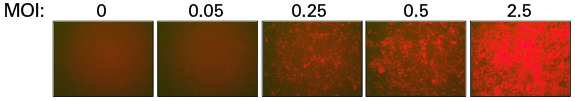
sgRNAライブラリー発現レンチウイルスを、マニュアルに従って作製した。 レンチウイルスを48時間後に回収し、様々なMOIでCas9発現A375細胞に導入した。 48時間後に蛍光顕微鏡およびFACSにより導入効率を測定した。
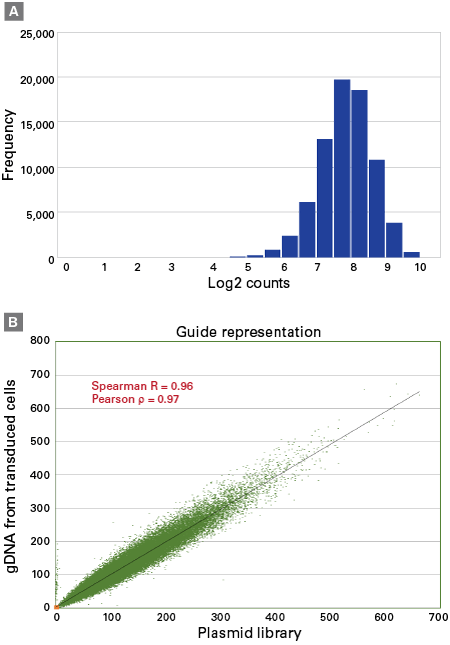
- Panel A. sgRNAのライブラリーのガイド配列の発現
- Brunelloに基づくsgRNA libraryをpLVXS-sgRNA-mCherry-hyg Vectorにクローニングし、プラスミドを増幅した。プラスミドDNAライブラリー内のsgRNAの配列をNGSにより確認した。ライブラリーに含まれるすべてのsgRNAの90%以上の配列は、10倍の分布範囲内に含まれた。
- Panel B. プラスミドライブラリーと形質導入細胞におけるガイド配列の比較
- ハイグロマイシンで選択されたCas9/sgRNA発現A375細胞からゲノムDNAを抽出し、PCR増幅した。PCR産物のシーケンスを行い、プラスミドDNAライブラリーに対応するsgRNAのリードのカウントを決定した。その結果、SpearmanおよびPearsonの強い相関が観察され、このシステムが、形質導入および選択された細胞集団においてsgRNA配列を保持していることが確認された。
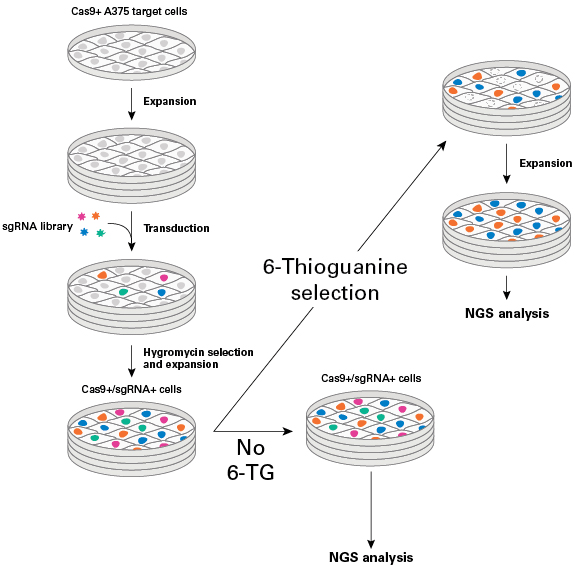
Cas9発現A375細胞にsgRNAライブラリーを導入後、ハイグロマイシンにより選択し、増殖後、Cas9/sgRNA発現細胞を2つの集団に分けた。
Cas9+/sgRNA+細胞集団の一方に6-TGを添加してスクリーニングを行った。もう一方の細胞集団はコントロール(非スクリーニング細胞)として6-TGを添加せず培養した。両方の細胞集団を培養した後、Guide-it CRISPR Genome-Wide sgRNA Library NGS Analysis Kitを使用して、ゲノムDNAの精製とライブラリー調製を行い、イルミナ社MiSeqを使用して解析した。
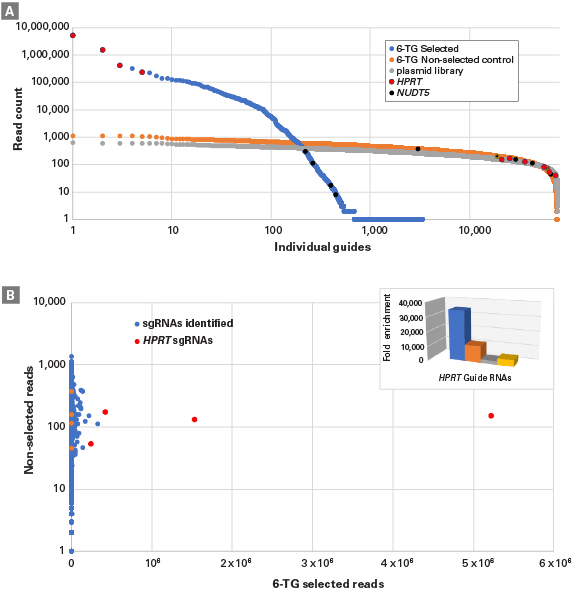
Panel A. 6-TGでスクリーニングを行った細胞と行っていない細胞に対して、sgRNAの発現比較を行った。スクリーニングを行った細胞(青い点)では、6-TGの代謝に関わっているHPRT遺伝子をターゲットとしたsgRNA由来のリード数の優位な増加が見られ(赤い点)、今回の表現型の変化に関わっている候補遺伝子として示された。一方、非スクリーニング細胞(オレンジの点)では、各sgRNA由来のリード数はほぼ均一であった。
Panel B. スクリーニングを行った細胞について、HPRT遺伝子をターゲットとする4つのsgRNA由来のリード数が優位に増加していることが観察された(赤い点)。非スクリーニング細胞では、各sgRNA由来のリード数に大きな差はなかった(青い点)。
内容
- Guide-it CRISPR Genome-Wide sgRNA Library System(製品コード 632646)
-
Guide-it Cas9 Lentiviral Transfection Mix5 vial
Guide-it Lentiviral sgRNA Library Transfection Mix5 vial×2
pLVXS-sgRNA-mCherry-hyg Vector (0.5 μg/μl)10μl
Lenti-X 293T Cell Line1 ml
Lenti-X GoStix Plus- Chase Buffer3 ml
- Lenti-X GoStix Plus3 testes
- p24 Control5 tests
- Guide-it CRISPR Genome-Wide Library PCR Kit(製品コード 632651)
-
LVXS P5 primer pool with index 1 (10 μM)100 μl
LVXS P7 primer with index 2 (10 μM)100 μl
LVXS P7 primer with index 3 (10 μM)100 μl
LVXS P7 primer with index 4 (10 μM)100 μl
LVXS P7 primer with index 5 (10 μM)100 μl
TaKaRa Ex Taq (5 U/μl)30 μl
10X Ex Taq Buffer (Mg2+ plus)500 μl
dNTP Mixture (2.5 mM each)400 μl
- Guide-it CRISPR Genome-Wide sgRNA Library NGS Analysis Kit(製品コード 632647)
-
・Guide-it CRISPR Genome-Wide Library PCR Kit(製品コード 632651)
・NucleoBond CB 500(製品コード 740509)
・NucleoSpin Gel and PCR Clean-up (製品コード 740609.10)
保存
- Guide-it CRISPR Genome-Wide sgRNA Library System
-
- Guide-it Cas9 and sgRNA library transfection mix:-20℃
付属の乾燥剤を含むポーチ内で保存する。使用後も、未使用のtransfection mixはポーチに戻して保存する。 - pLVXS-sgRNA-mCherry-hyg Vector:-20℃
- Lenti-X 293T Cell Line:液体窒素
到着後、ただちに液体窒素中で保存する。液体窒素保存ができない場合は、ただちに細胞を融解し、培養を行う。 - Lenti-X GoStix Plus:室温
- Guide-it Cas9 and sgRNA library transfection mix:-20℃
- Guide-it CRISPR Genome-Wide Library PCR Kit:-20℃
- NucleoBond CB 500、NucleoSpin Gel and PCR Clean-up:室温
この製品を見た人は、
こんな製品も見ています
-

Guide-it™ CRISPR/Cas9 Gesicle Production System
エキソソーム様小胞を利用してCas9タンパク質/sgRNA複合体を細胞に直接導入
-
Guide-it™ Genotype Confirmation Kit
ゲノム編集後のクローンの簡易ジェノタイピング
-
Guide-it™ sgRNA In Vitro Transcription and Screening System
in vitro transcriptionによるsgRNAの調製およびsgRNAの有効性の確認
-
ゲノム編集ガイド
ゲノム編集を強力に支えるサポートツール
-

植物に使えるゲノム編集関連製品
-
CRISPR/Cas9 プラスミド システム
プラスミドDNA用いるゲノム編集システム
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。