低価格・簡便操作の全長mRNA解析用ライブラリー調製キット(シングルセル or 超微量RNA)
- 高感度 - シングルセルまたは10 pg total RNAからライブラリー調製が可能(インプット量:1~100細胞または10 pg~1 ng total RNA)
- 高品質のRNA-Seqデータ - 完全長の遺伝子情報を保持し、rRNAのリード割合が低く、GC richのトランスクリプトも効率よく増幅
- シングルチューブワークフロー -サンプルロスやハンドリングエラーを最小限に抑えた簡便なcDNA合成とライブラリー調製
- 信頼性の高いマルチプレックス解析 - Unique Dual Index Kit(別売り)の利用によりインデックスホッピングの影響を軽減
- 高いライブラリー収量 - NovaSeqなどによるハイスループットシーケンスに必要な量を簡単に確保
- 自動化対応のワークフロー - より多くのサンプルを短時間で処理可能
注: SMART-Seq mRNA HTおよび SMART-Seq mRNA HT LPは、SMART-Seq HT Kitおよび SMART-Seq HT PLUS Kit(終売)のリニューアル製品です。基本性能、プロトコールに変更はありませんが、インデックスが別売りとなっていますので、必ず別途ご購入ください。(Unique Dual Index Kit参照)
新製品と既存製品の対応表はこちら(TB USA社のサイト)
新製品と既存製品の対応表はこちら(TB USA社のサイト)
製品説明
SMART-Seq mRNA HT(SSmRNA HT)は、10 pg~1 ng total RNAまたは1~100細胞から、Oligo(dT)プライマーを用いて高品質な全長cDNAを調製することができ、自動化装置にも対応したキットです。
逆転写とPCR増幅をワンステップで行うことができ、ハンズオンタイムが短く簡単なプロトコールで実験が可能です。FCMの下流実験にも使用できるようプロトコールが最適化されています。
本キットのcDNA合成ステップの核となるSMART(Switching Mechanism at 5' End of RNA Template)テクノロジーは、完全長の遺伝子カバレッジを得ることができ、スプライスジャンクションや細胞内の選択的スプライシングの解析を含む、シングルセルにおける遺伝子発現研究の強力なツールです。
本キットはOligo(dT)プライミングにより、高品質total RNA(RIN>8)またはインタクトな細胞からmRNAを特異的に増幅します。また、LNA(Locked Nucleic Acid)技術を取り入れることにより、テンプレートスイッチング効率を向上させ、他の手法よりも多くの遺伝子検出が可能になりました。
SMART-Seq mRNA HT LP(SSmRNA HT LP)は、cDNA合成のためのSSmRNA HTと、酵素による断片化およびステムループアダプターを組み込んだライブラリー調製キットが含まれており、イルミナ社次世代シーケンサー対応の高品質なライブラリー調製が可能です。またUnique Dual Index Kit(別売り)と組み合わせることで、最大384サンプルのマルチプレックス解析に対応できます。断片化とアダプター付加の2ステップワークフローは、途中で精製工程が不要なため1チューブで完結でき、コンタミやサンプルロスを軽減し、約2時間でステップが完了します。これにより高収量なライブラリーが得られ、ハイスループットシーケンサーへの対応や再現性の高い解析が可能となります。
無料のバイオインフォマティクス解析ツールCogent NGSもご用意しており、サンプルからのcDNA合成、ライブラリー調製、データ解析までend-to-endのソリューションを提供いたします。
Cogent NGSソフトウェアは下記ページからダウンロードしてください。
Cogent NGS Analysis Pipeline(TB USA社)
Cogent NGS Discovery Software(TB USA社)
逆転写とPCR増幅をワンステップで行うことができ、ハンズオンタイムが短く簡単なプロトコールで実験が可能です。FCMの下流実験にも使用できるようプロトコールが最適化されています。
本キットのcDNA合成ステップの核となるSMART(Switching Mechanism at 5' End of RNA Template)テクノロジーは、完全長の遺伝子カバレッジを得ることができ、スプライスジャンクションや細胞内の選択的スプライシングの解析を含む、シングルセルにおける遺伝子発現研究の強力なツールです。
本キットはOligo(dT)プライミングにより、高品質total RNA(RIN>8)またはインタクトな細胞からmRNAを特異的に増幅します。また、LNA(Locked Nucleic Acid)技術を取り入れることにより、テンプレートスイッチング効率を向上させ、他の手法よりも多くの遺伝子検出が可能になりました。
SMART-Seq mRNA HT LP(SSmRNA HT LP)は、cDNA合成のためのSSmRNA HTと、酵素による断片化およびステムループアダプターを組み込んだライブラリー調製キットが含まれており、イルミナ社次世代シーケンサー対応の高品質なライブラリー調製が可能です。またUnique Dual Index Kit(別売り)と組み合わせることで、最大384サンプルのマルチプレックス解析に対応できます。断片化とアダプター付加の2ステップワークフローは、途中で精製工程が不要なため1チューブで完結でき、コンタミやサンプルロスを軽減し、約2時間でステップが完了します。これにより高収量なライブラリーが得られ、ハイスループットシーケンサーへの対応や再現性の高い解析が可能となります。
無料のバイオインフォマティクス解析ツールCogent NGSもご用意しており、サンプルからのcDNA合成、ライブラリー調製、データ解析までend-to-endのソリューションを提供いたします。
Cogent NGSソフトウェアは下記ページからダウンロードしてください。
Cogent NGS Analysis Pipeline(TB USA社)
Cogent NGS Discovery Software(TB USA社)
※以下のデータは、SSmRNA HT / HT LPと同じケミストリー・性能のSMART-Seq HT Kit(SS-HT)(終売)とSMART-Seq HT PLUSキット(SS-HT PLUS)(終売)で得たものを示しています。
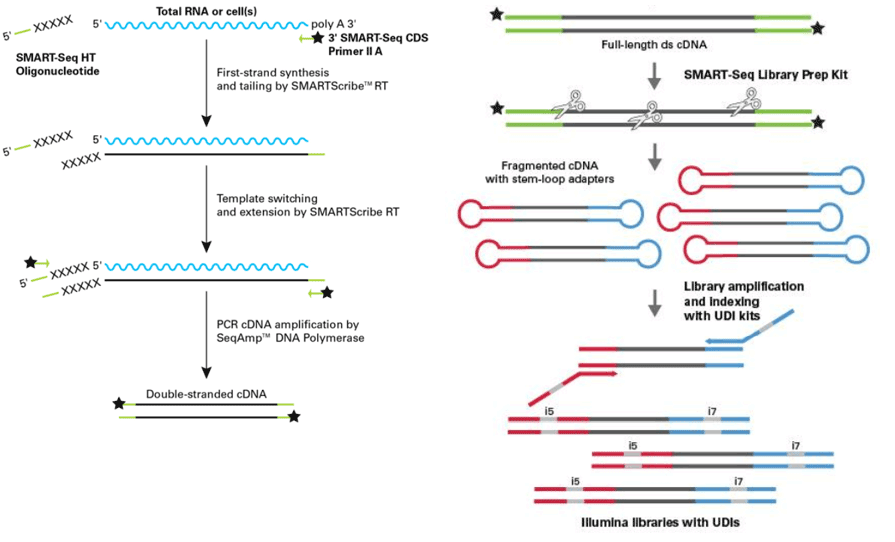
図1. cDNA合成とライブラリー調製のワークフロー
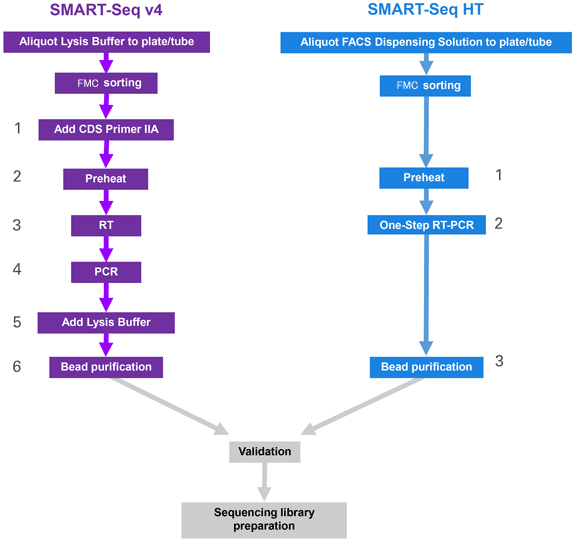
図2. SMART-Seq v4 KitとSMART-Seq HT Kit のワークフローの比較
SMART-Seq HT(右)は、SMART-Seq v4のプロトコール(左)を改変することにより、簡便でハイスループットなワークフローで実験が可能。セルソーターによりシングルセルを取得した後、SMART-Seq v4 Kitの場合、6つのハンズオンステップが含まれるが、SMART-Seq HT Kitの場合は、3つのハンズオンステップのみで行うことができる。SMART-Seq HTワークフローの重要なステップは、効率的な逆転写およびPCR cDNA増幅のために最適化されたOne-Step RT-PCR Bufferを使用してOne-Step RT-PCRを行うことである。One-Step RT-PCR BufferはLysis Bufferを添加する必要がなく、AMPureビーズ精製が可能である。SMART-Seq HT Kitは、従来のSMART-Seq v4 Kitと同様に、シーケンス用のライブラリー調製(Nextera XT)の前に、調製したcDNAの検証(定量、サイズ分布)が必要である。
※SMART-Seq v4は後継品SMART-Seq mRNAシリーズと同じプロトコール・性能を示します。
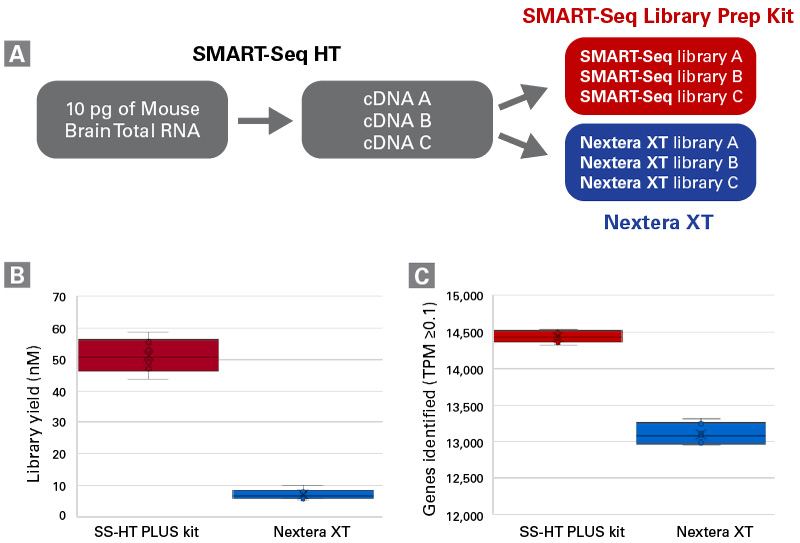
図3. SMART-Seq HT KitとNextera XTとの比較
パネルA:SMART-Seq HT Kitを使用して10 pgのマウス脳total RNAからtriplicateでcDNAを合成した。次に、SMART-Seq HT PLUS Kit(終売)に含まれるSMART-Seq Library Prep KitまたはNextera XT Kit(イルミナ社)を使用して、イルミナ社シーケンサー用のライブラリーを調製した。ライブラリーを4M paired-endリードにノーマライズし、NextSeq 500でシーケンスを行った。
パネルB:SMART-Seq HT PLUS Kit(終売)はNextera XTより高収量のライブラリーが得られた。
パネルC:SMART-Seq HT PLUS Kit(終売)は、Nextera XTと比較して10%多くの遺伝子が同定できた。
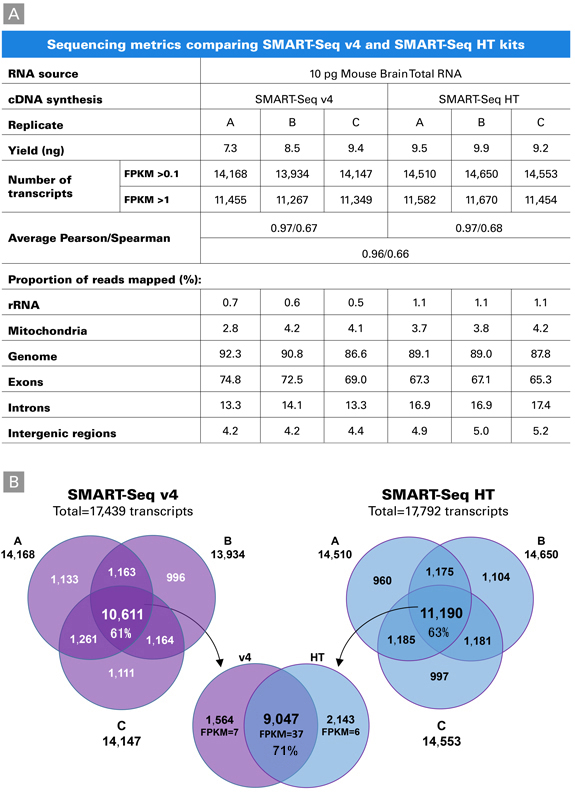
図4.SMART-Seq v4 KitおよびSMART-Seq HT Kitで検出されたトランスクリプトのオーバーラップ
パネルA:マウス脳 total RNA10 pgからtriplicateでライブラリーを調製し、各キットの得られた収量やトランスクリプト数、マッピングされたリードの割合を表に示した。
パネルB:パネルAの数値をもとに、各キットのreplicate間で検出されたトランスクリプト(fragment per kilobase of exon per million reads mapped:FPKM>0.1)のオーバーラップ数について評価した結果、replicate間のトランスクリプトは両キットとも類似性が高いことが示された(SMART-Seq v4:61%、SMART-Seq HT:63%)。また、各キットの3つのreplicateで共通して検出されたトランスクリプトを互いに比較した結果、71%のオーバーラップ率を示した。オーバーラップしているトランスクリプトの発現量は平均37 FPKMであったのに対し、それぞれのキットでのみ検出されたトランスクリプトの発現量は平均6~7 FPKMと少なかったことから、低レベルで発現しているトランスクリプトは検出されにくいことが分かる。
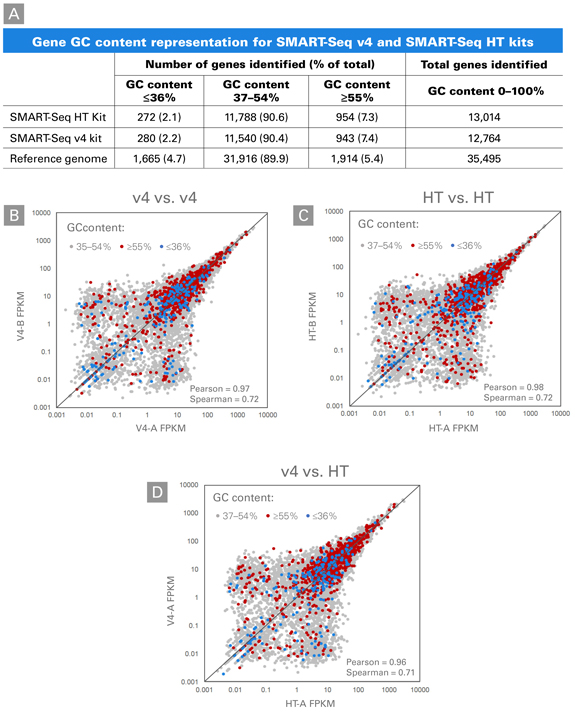
図5. SMART-Seq v4 KitとSMART-Seq HT Kitを用いたGC含量の違いによる遺伝子発現レベルの比較
図3においてマウス脳のtotal RNA 10 pgから調製したライブラリーを、GC含有量についてさらに分析した(パネルA)。遺伝子をGC含量(≦36%、37~54%、≧55%)で分類し、スキャッタープロットを使用して各分類内の遺伝子の発現レベル(FPKM)の再現性を視覚化した。SMART-Seq v4(パネルB)とSMART-Seq HT Kit(パネルC)どちらを使用した場合も、それぞれのreplicate間において、遺伝子発現レベルに非常に高い再現性が見られた。またSMART-Seq v4 KitとSMART-Seq HT Kit間においても、高GC含量(●)または低GC含量(●)の遺伝子は同様の発現レベルを示した(パネルD)。このことからSMART-Seq HT Kitで採用されているOne-step RT-PCR反応は、逆転写とPCRを2ステップで行うSMART-Seq v4と比較しても、低GC含量および高GC含量遺伝子の増幅に影響がないことが示された。
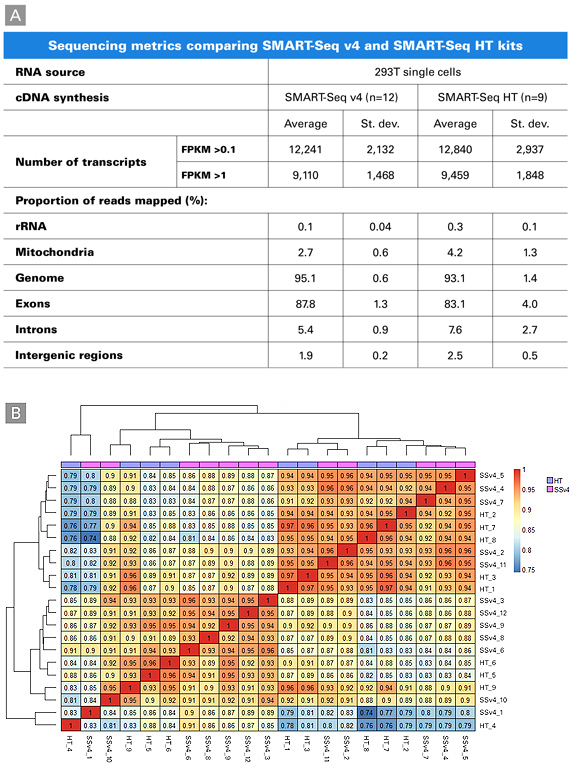
図6. SMART-Seq v4 KitおよびSMART-Seq HT Kitを用いた、遺伝子発現データの再現性比較
SMART-Seq v4 Kit(SSv4_1~SSv4_12)およびSMART-Seq HT Kit (HT_1 to HT_9)を用いて、293T細胞の21個のシングルセルからライブラリーを調製し、各細胞から得られた遺伝子発現の再現性を評価した。
0.74から0.97のピアソン相関係数から階層的クラスタリングヒートマップを作成し、21個の細胞間のユークリッド距離を示した。
同一のキットで調製された細胞間で最も高い相関が見られるが、2つのキット間の相関関係も依然として非常に高く、ライブラリー調製方法によるクラスター化はしていないことが分かる。
これらのデータより、簡便ワークフローを採用したSMART-Seq HTはSMART-Seq v4と比較して、遺伝子発現解析において大きな偏りがないことが示された。
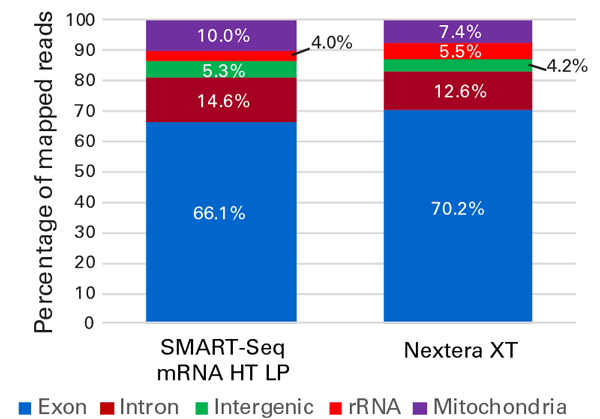
図7. SSmRNA HTとNextera XTから調製したライブラリーのリード分布の比較
SSmRNA HT LPを用いてマウス脳のtotal RNA 10 pgからtriplicateでcDNA合成を行った。ライブラリーはSMART-Seq Library Prep(SSmRNA HT LPのコンポーネント)とイルミナ社のNextera XTを用いて、cDNA 1 ngあるいは125 pgから調製し、NextSeq 500でシーケンスした。シーケンス結果は4M paired-endリードにノーマライズし、CLC Genomic Workbenchで解析を行った。
2キットからそれぞれ調製されたライブラリーからは、類似したリード分布が得られた。
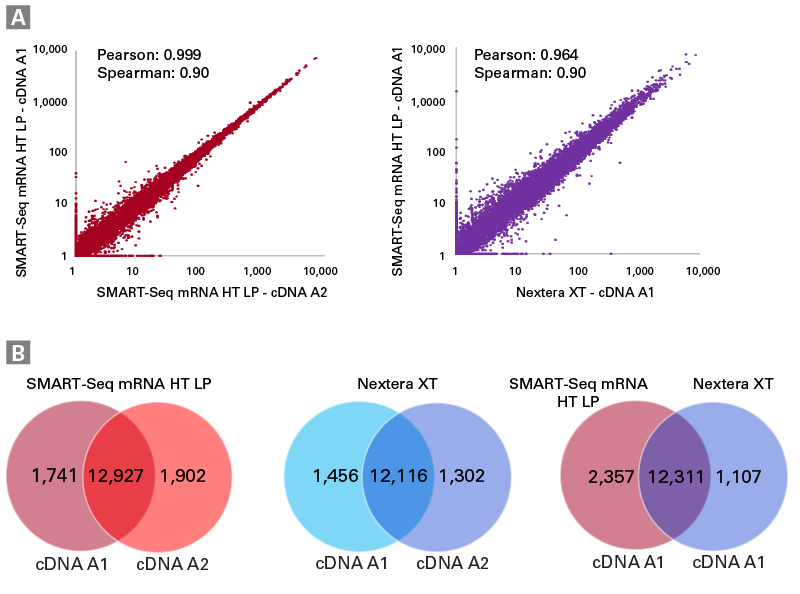
図8. SSmRNA HT LPとNextera XTの遺伝子同定性能の比較
cDNA合成はSSmRNA HTで、ライブラリー調製はSMART-Seq Library Prep(SSmRNA HT LPのコンポーネント)とイルミナ社のNextera XTで調製し、比較した。ライブラリーはそれぞれduplicate(上図A1, A2)で調製した。
パネルA:SSmRNA HTとNextera XTで調製したライブラリーの全遺伝子のTPM(transcript per million)値を対数目盛でプロットし、相関係数を算出した。
パネルB:同じキットで調製されたライブラリー同士、またそれぞれのキットで調製されたライブラリーから同定された遺伝子をベン図で示した。どちらのキットで調製されたライブラリーも同様のTPMを示し、両キット間では12,311個の同じ遺伝子が同定された。
内容
- SMART-Seq mRNA HT(製品コード 634795/634796/634791)
-
Box 1(Store at -70℃)
Control Total RNA(1 µg/µl) (マウス脳由来)Box 2(Store at -20℃)
SMART-Seq HT Oligonucleotide
3ʹ SMART-Seq CDS Primer II A
RNase Inhibitor(40 U/µl)
SMARTScribe Reverse Transcriptase(100 U/µl)
One-Step Buffer
SeqAmp DNA Polymerase
10X Lysis Buffer
Elution Buffer(10 mM Tris-Cl, pH 8.5)
Nuclease-Free Water - SMART-Seq mRNA HT LP(製品コード 634792/634793/634794)
-
SMART-Seq mRNA HT(製品コード 634795/634796/634791)
SMART-Seq Library Prep Kit*
10X FE
FE Dilution Buffer
Lib Prep Buffer
Lib Prep Enzyme
Rxn Enhancer
Stem Loop Adapters
Amplification Buffer
PrimeSTAR HS DNA Polymerase(5 U/µl)
Nuclease-Free Water
*Library Prep Kitの単品販売はありません。
保存
SMART-Seq mRNA HT
SMART-Seq Library Prep Kit:-20℃
Control Total RNA:-70℃
その他のコンポーネント:-20℃
(10X Lysis Buffer:融解後は4℃保存、Elution Buffer:融解後は室温保存)
その他のコンポーネント:-20℃
(10X Lysis Buffer:融解後は4℃保存、Elution Buffer:融解後は室温保存)
SMART-Seq Library Prep Kit:-20℃
本製品以外に必要な試薬、機器(主なもの)
下記の製品は本キット中に含まれません。下記の製品はユーザーマニュアルに記載のProtocolで使用できることが確認されています。
Index Kit
セルソーティング
One-Step RT PCR増幅およびバリデーション
SPRI Beads精製
ライブラリー調製
その他
Index Kit
- Unique Dual Index Kit (1-24)(製品コード 634756)
UDI No. U001-U024 - Unique Dual Index Kit (1-96)(製品コード 634752)
UDI No, U001-U096 - Unique Dual Index Kit (97-192)(製品コード 634753)
UDI No, U097-U192 - Unique Dual Index Kit (193-288)(製品コード 634754)
UDI No, U193-U288 - Unique Dual Index Kit (289-384)(製品コード 634755)
UDI No, U289-U384
セルソーティング
- 8-tube strips(Thermo Fisher Scientific社 Cat. No. AB0264)またはセルソーティングに適したPCR tube strips, 96 well plates
- (オプション)BD FACS Pre-Sort Buffer(BD社 Cat. No. 563503)
- Low-speedの96 well plate/strip用卓上遠心機
- Adhesive PCR Plate Foils(Thermo Fisher Scientific社 Cat. No. AB-0626)またはFlat PCR Caps, strips of 8(Thermo Fisher Scientific社 Cat. No. AB0784/AB0850)
- ドライアイス(細胞の凍結用)
- (オプション)SMART-Seq HT Kit Lysis Components(製品コード 634439)または10× Lysis Buffer(製品コード 635013)
One-Step RT PCR増幅およびバリデーション
- One-step cDNA合成専用のPCRサーマルサイクラー
- ライブラリー増幅専用のPCRサーマルサイクラー
- 96 well Plate用粘着シール:Thermo Scientific Adhesive PCR Plate Seals(Thermo Fisher Scientific社 Cat. No. AB0558)または 8 tube strips用Cap strips(Thermo Fisher Scientific社 Cat. No. AB0784/AB0850)
- 精製済みのRNAからcDNA合成をする場合
- Nuclease-free, PCR grade, thin-wall PCR strips(0.2 ml PCR 8 tube strip; Thermo Fisher Scientific社 Cat. No. AB0264)
- Nuclease-free low-adhesion 1.5 ml tubes(USA Scientific社 Cat No. 1415-2600)または、DNA LoBind tubes(Eppendorf社 Cat. No. 022431021)
SPRI Beads精製
- NucleoMag NGS Clean-up and Size Select(5 ml size:製品コード 744970.5;50 ml size:製品コード 744970.50;500 ml size:製品コード 744970.500) ※NucleoMagの代わりにAMPure XP PCR purification kit(Beckman Coulter社 5 ml size:Cat. No. A63880;60 ml size:Cat. No. A63881)も使用可能
- Magnetic Separation Device
12~24サンプル用0.2 mlチューブ用マグネットスタンド24~96サンプル用
0.2 mlチューブまたはストリップ用遠心機
96 well フォーマット用:Magnetic Stand 96(Thermo Fisher Scientific社 Cat. No. AM10027)など1.5 ml用(シーケンス用ライブラリーをプールする場合に必要)Magnetic Stand(製品コード 631964) - 8 tube strips(Thermo Fisher Scientific社 Cat. No. AB0264)またはPCRラックに固定されNuclease-freeのPCR tube strips、またはセルソーターで動作確認済みの96 well Plate
- 80% Ethanol(必ず用時調製すること)
ライブラリー調製
- Nuclease-free, PCR grade, thin-wall PCR strips(0.2 ml PCR 8 tube strip; Thermo Fisher Scientific社 Cat. No. AB0264)
- Nuclease-free low-adhesion 1.5 ml tubes(USA Scientific社 Cat No. 1415-2600)または、DNA LoBind tubes(Eppendorf社 Cat. No. 022431021)
- Benchtop cooler, VWR CryoCoolers(VWR社 Cat. No. 414004-286)など
その他
- シングルチャンネルピペット:10 μl、20 μlおよび200 μl
- 8連チャンネルピペット:20 μl、200 μl
- フィルターピペットチップ:10 μl、20 μlおよび200 μl
- 1.5 mlチューブ用微量遠心機
- 0.2 mlチューブまたはstrip用微量遠心機
- 96 well冷却ラック:IsoFreeze PCR Rack(MIDSCI社 Cat. No. 5640-T4)または96-Well Aluminum Block(Light Labs社 Cat. No. A-7079)など
この製品を見た人は、
こんな製品も見ています
-

SMART-Seq® HT Kit
低価格・簡便操作の全長mRNA解析(シングルセル or 超微量)
-

Unique Dual Index Kits
イルミナ社NGSライブラリー調製キット用インデックスキット
-

SMART-Seq® mRNA / SMART-Seq® mRNA LP
イルミナ社NGS装置用の超微量mRNA-Seq用ライブラリー調製キット
-
SMART-Seq® mRNA Single Cell / SMART-Seq® mRNA Single Cell LP
シングルセル解析に最適化されたイルミナ社NGS装置用mRNA-Seq解析
-
次世代シーケンス(NGS)関連試薬選択ガイド RNAシーケンス
-

SMART-Seq® mRNA LP (with UMIs)
分子バーコードによりさらに信頼性が向上した超微量mRNA-Seq用ライブラリー調製キット
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。