- 1~1,000個の細胞ダイレクトまたは微量(10 pg~10 ng)のtotal RNAから7時間でRNA-Seq用ライブラリーを調製
- シングルセルから方向性情報を持った全長total RNAシーケンスが可能
- シングルセルまたはtotal RNAからコーディング/ノンコーディングのトランスクリプトを再現性高く正確に検出
- 分解を受けたRNAからもライブラリー調製可能
- 対象生物種はヒト、マウス、ラット
製品説明
本キットはランダムプライマーによるcDNA合成を行うため、すべてのトランスクリプトをキャプチャーし、分解の進んだ低品質RNAでも使用可能である。高品質RNAを用いてオリゴdTプライマーによるcDNA合成を行いたい場合はSMART-Seq v4 Ultra Low Input RNA Kit for Sequencingをお勧めする。
また、本キットにはインデックスプライマーが含まれているが、インデックスホッピング低減のためのユニークデュアルインデックスキット(別売)も使用可能である。
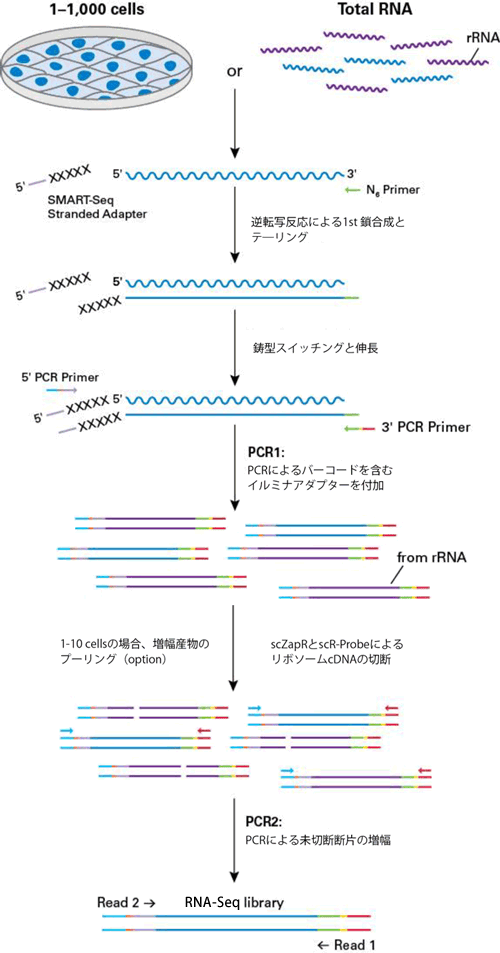

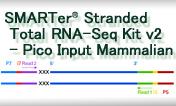
PCR1で付加されたアダプターは、イルミナ社次世代シーケンサーのフローセルへの結合部位(P7、P5)およびIndex(Index 1 [i5]、Index 2 [i7])配列を含んでおり、シーケンスプライマーRead 2およびRead 1を認識する領域も含んでいる。
Read 1は元のRNAのアンチセンスの配列が得られ、Read 2では元のRNAのセンス鎖の配列が得られる。Read 2を用いた場合の最初の3塩基 (XXX)はSMART-Seq Stranded Adapter由来の配列になるので、paired-end sequencingを行った場合、この3塩基はマッピング前にトリミングする必要がある。
| Sequencing alignment metrics for 10 pg–10 ng total RNA | |||||
|---|---|---|---|---|---|
| RNA source | Human brain total RNA | ||||
| Input amount (ng) | 10 | 1 | 0.25 | 0.05 | 0.01 |
| Number of reads (paired-end) | 2,500,000 | 2,500,000 | 2,500,000 | 2,500,000 | 1,000,000 |
| Number of transcripts > 1 FPKM | 15,128 | 15,097 | 15,066 | 14,394 | 13,151 |
| Number of transcripts > 0.1 FPKM | 23,864 | 23,631 | 23,274 | 21,335 | 16,700 |
| Pearson/Spearman correlations | 0.99/0.87 | 0.99/0.85 | 0.99/0.82 | 0.97/0.68 | 0.92/0.46 |
| Correct strand per biological annotation (%) | 97.7 | 97.8 | 97.6 | 97.5 | 97.1 |
| Proportion of reads (%): | |||||
| Exonic | 37.1 | 36.5 | 41.5 | 39.7 | 34.1 |
| Intronic | 36.1 | 35.6 | 36.7 | 35.4 | 30.6 |
| Intergenic | 8.6 | 8.5 | 8.8 | 8.7 | 7.4 |
| rRNA | 9.7 | 9.6 | 3.6 | 4.1 | 6.7 |
| Mitochondrial | 5.2 | 6.3 | 6.4 | 6.4 | 5.9 |
| Overall mapping (%) | 96.8 | 96.4 | 97 | 94.4 | 84.7 |
| Duplicate rate (%) | 13.3 | 20.2 | 35.2 | 59.0 | 62.4 |
ヒト脳の total RNA(10 pg~10 ng)から本キットと用いて、RNA-seqライブラリーを作製し、NextSeq 500でシーケンスを行った。
本表のデータは、三連の実験の平均を示し、わずか10 pgのRNA input量の場合でも非常に高い再現性(Pearson/Spearman)を示した。
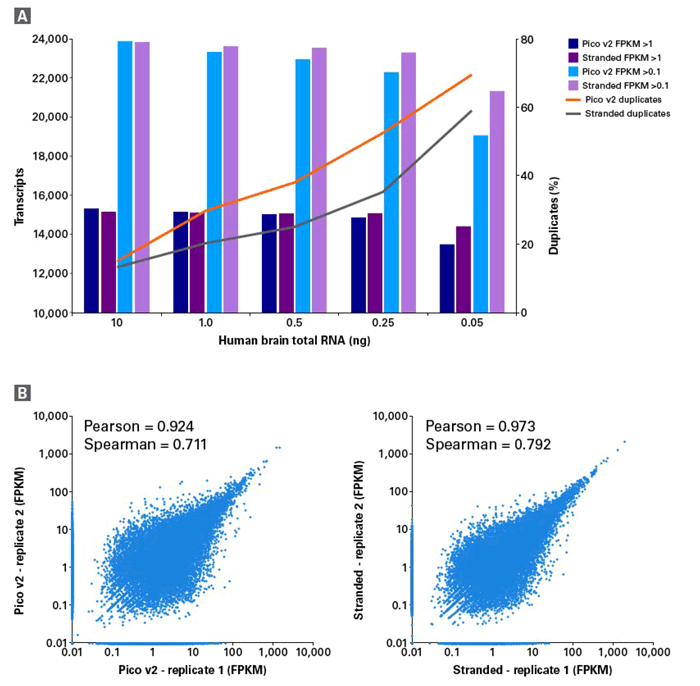
Panel A:ヒト脳のtotal RNA(50 pg~10 ng)を用いてSMART-Seq Stranded KitおよびSMARTer Stranded Total RNA-Seq Kit v2 - Pico Input Mammalian(Pico v2)を用いてRNA-seqライブラリーを三連で作製した。取得したデータは250万リード(paired-end)で解析を行った。
Pico v2 KitのインプットRNAの範囲(250 pg~10 ng)では、両キットでほぼ同等の性能を示したが、1 ng以下では、SMART-Seq Stranded Kitのほうがより多くのトランスクリプトを検出した。
Panel B:50 pgの total RNAから各々のキットで作製されたライブラリーを用いて発現解析を行い、再現性を比較した。
SMART-Seq Stranded Kit(Pearson=0.97)は50 pg total RNAにおいてもPico v2(Pearson=0.92)よりも高い再現性を示した。
| Sequencing alignment metrics for A375 total RNA and cells | |||||||
|---|---|---|---|---|---|---|---|
| Input | Total RNA | 1,000 cells | 500 cells | 100 cells | 10 cells | 5 cells | 1 cell |
| Number of reads (pairs) | 6,000,000 | 6,000,000 | 6,000,000 | 6,000,000 | 6,000,000 | 6,000,000 | 5,873,974 |
| Number of transcripts >1 FPKM |
13,260 | 13,294 | 13,583 | 13,520 | 12,726 | 12,602 | 11,540 |
| Number of transcripts >0.1 FPKM |
21,334 | 21,113 | 21,365 | 21,145 | 20,550 | 18,888 | 15,815 |
| Proportion of reads(%): | |||||||
| Exonic | 34.7 | 36.4 | 39.2 | 42.7 | 36.7 | 36.2 | 37.3 |
| Intronic | 29.6 | 29.3 | 27.7 | 28.3 | 34 | 30.4 | 21.1 |
| Intergenic | 14.2 | 13.4 | 12.2 | 12.9 | 16.7 | 16.8 | 10.1 |
| rRNA | 7.0 | 11.4 | 11.5 | 6.3 | 3.6 | 4.9 | 7.1 |
| Mitochondrial | 4.1 | 3.5 | 3.7 | 4.9 | 3.8 | 4.4 | 4.6 |
| Overall maping (%) | 89.6 | 93.9 | 94.3 | 95.1 | 94.9 | 92.7 | 80.2 |
| Duplicate rate(%) | 37.3 | 45.2 | 40.3 | 46.1 | 52.5 | 72.2 | 78.5 |
| lncRNA mapping: | |||||||
| Number of mapped reads(%) | 7.2 | 10.4 | 10.8 | 9.4 | 8.7 | 8.6 | 7.3 |
| lncRNA transcripts detected | 5,395 | 4,687 | 4,565 | 5,439 | 5,440 | 4,983 | 2,802 |
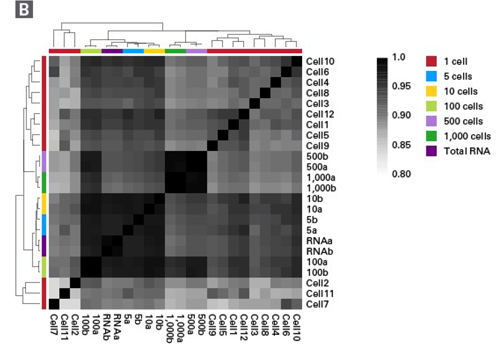
セルソーティングによって単離されたA375細胞から、SMART-Seq Stranded Kitを用いてライブラリーを作製した。インプットした細胞数は1細胞から1,000細胞で、5~1,000細胞は二連、1細胞は12連で行った。 比較のために1,000細胞から精製したtotal RNAを用いて二連でライブラリーを作製した。
Panel A: 1~1,000細胞から調製したライブラリーの評価
Panel B: Panel Aに示された全てのサンプル間のユークリッド距離を表示した階層的クラスタリング解析。Pearson相関係数は0.85から0.99を示した。シングルセルはCell1~Cell12と表示し、他のインプット量の細胞のレプリケートはa~bと表示している。
内容
- Box 1
-
SMART scTSO Mix
scR-Probes
Control Total RNA (1 μg/μl)
- Box 2
-
scZapR v2
SMART scN6
scRT Buffer
SMARTScribe RT (100 U/μl)
RNase Inhibitor (40 U/μl)
ZapR Buffer (10X)
Tris Buffer (5 mM)
PCR2 Primers v2
Nuclease-Free Water
SeqAmp DNA Polymerase
SeqAmp CB PCR Buffer (2X):
10X Lysis Buffer
Indexing Primer Set:
- 12回用(製品コード 634442)
-
Indexing Primer Set HT for Illumina v2-12
3' PCR Primer HT Index 2 (3' 2;12.5 μM)
5' PCR Primer HT Index 1~12 (5' 1~12; 12.5 μM) - 48回用(製品コード 634443)
-
Indexing Primer Set HT for Illumina v2- 48
3' PCR Primer HT Index 1~4 (3' 1~4; 12.5 μM)
5' PCR Primer HT Index 1~12 (5' 1~12; 12.5 μM) - 96回用(製品コード 634444)
-
Indexing Primer Set HT for Illumina v2-96
3' PCR Primer HT Index 1~8 (3' 1~8; 12.5 μM)
5' PCR Primer HT Index 1~12 (5' 1~12; 12.5 μM)
保存
Package 2:-20℃
Indexing Primer Set:-20℃
本製品以外に必要な試薬、機器(主なもの)
- シングルチャンネルピペット: 10 μl、20 μlおよび200 μl
- 8連チャンネルピペット: 20 μl、200 μl
- Pre-PCR amplification用のPCRサーマルサイクラー
- PCR amplification用のPCRサーマルサイクラー
(Final RNA-Seq Library Amplificationのステップを100μlの反応液でPCRを行うため、100 μlの反応液量に対応したPCRサーマルサイクラーを使用する。50 μlの反応液量に対応したサーマルサイクラーの場合は、2本のチューブにわけてPCRを行う。) - フィルターピペットチップ: 10 μl、20 μlおよび200 μl
- 0.2 ml tube minicentrifuge
- 96-well PCR chiller rack: IsoFreeze PCR Rack (MIDSCI Cat. No. 5640-T4) or 96 Well Aluminum Block (Light Labs Cat. No. A-7079)
- セルソーティング
-
- 96-well polycarbonate PCR plates (USA Scientific社 Cat.No . 2796-3330) or 8-tube strips (Thermo Fisher Scientific社 Cat No. AB-0264) inserted into a PCR rack
- BD FACS Pre-Sort Buffer (BD Cat No. 563503)
- Dulbecco’s phosphate-buffered saline (DPBS without Ca2+ and Mg2+;) (Sigma-Aldrich 社 Cat. No. D8537)
- Adhesive PCR Plate Foils (Thermo Fisher Scientific社 Cat No. AB-0626) or 8-cap strips (Thermo Fisher Scientific社 Cat No. AB-0784)
- ドライアイス
- PCR増幅およびバリデーション
-
- Agilent High Sensitivity DNA Kit (Agilent Cat No.5067-4626)
- Qubit dsDNA HS kit (Thermo Fisher Scientific Cat. No. Q32851)
- Nuclease-free thin-wall PCR tube(0.2 ml;USA Scientific社 Code 1402-4700)
- Nuclease-free nonsticky 1.5 ml tubes (USA Scientific社 Code 1415-2600)
- SPRI Bead精製
-
- Agencourt AMPure XP PCR Purification Kit(5 ml; Beckman Coulter社; 5 ml Code. A63880、60 ml Code. A63881)
- 0.2 mlチューブ用マグネットスタンド
- 80% Ethanol(用時調製)
Application
【ユーザー様実施例】レーザーマイクロダイセクションで採取した細胞由来の微量分解RNAのリアルタイムPCRによる遺伝子発現解析
セルソーターを用いたシングルセルRNA-Seq実験を成功させるための5つのTips
シングルセルRNA-seq実験を成功させるための5つのTips
Technical Notes
High-quality-stranded-RNA-seq-libraries-from-single-cells-using-the-SMART-Seq-Stranded-Kit
RNA-seqを成功させるためのコツ(英語)
SMART-Seq® Stranded Kitによる卵巣がんの解析(英語)
RNA-seqのヒント(英語)
コントロールサンプルの準備(英語)
Total RNA-seqの概要(英語)
がんアプリケーションにおけるシングルセル解析(英語)
この製品を見た人は、
こんな製品も見ています
-

SMART-Seq® HT Kit
低価格・簡便操作の全長mRNA解析(シングルセル or 超微量)
-

SMART-Seq® Total RNA Single Cell Library Prep with ZapR® Depletion
シングルセルまたはtotal RNA 10 pgからの方向性情報を持ったtotal RNA-Seq解析
-

SMART-Seq® mRNA HT/SMART-Seq® mRNA HT LP
低価格・簡便操作の全長mRNA解析用ライブラリー調製キット(シングルセル or 超微量RNA)
-
SMARTer® Stranded Total RNA-Seq Kit v2 - Pico Input Mammalian
微量FFPEサンプルからもRNA-Seqが可能
-

SMART-Seq® v4 Ultra® Low Input RNA Kit for Sequencing
Illumina社NGSプラットフォームおよびIon TorrentのためのcDNA調製キット(超微量)
-

SMART-Seq® mRNA / SMART-Seq® mRNA LP
イルミナ社NGS装置用の超微量mRNA-Seq用ライブラリー調製キット
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。

