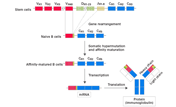
製品説明
SMARTer Human BCR IgG IgM H/K/L Profiling Kitは、5'RACE法とNGSテクノロジーを組み合わせることにより、RNAサンプルから、ヒトBCRプロファイリングの高感度で正確かつ最適化されたアプローチを提供する。5'RACE法の採用によりバイアスを減らし、BCRのH鎖またはL鎖の定常領域からのプライミングを可能にする。本キットは、遺伝子特異的増幅におけるこれらの利点を組み合わせて、BCR転写産物のV(D)J可変領域全体をキャプチャーし、高感度で再現性の高いB細胞レパトア解析を可能にする。
本キットは、末梢血単核細胞(PBMC)から10 ng~1 μgのtotal RNAまたはB細胞から1~100 ngのtotal RNAから高品質のイルミナ社次世代シーケンサー用ライブラリーを調製し、ヒト免疫グロブリンIgGおよびIgMのH鎖およびL鎖(κ鎖、λ鎖)の両方のデータが得られる。このプロファイリングキットには、分子バーコード(UMI)が含まれており、PCRエラー、duplicateやシーケンスエラーに由来するリードを除去できるため、より正確で信頼性の高い結果が得られる。
本キットは、末梢血単核細胞(PBMC)から10 ng~1 μgのtotal RNAまたはB細胞から1~100 ngのtotal RNAから高品質のイルミナ社次世代シーケンサー用ライブラリーを調製し、ヒト免疫グロブリンIgGおよびIgMのH鎖およびL鎖(κ鎖、λ鎖)の両方のデータが得られる。このプロファイリングキットには、分子バーコード(UMI)が含まれており、PCRエラー、duplicateやシーケンスエラーに由来するリードを除去できるため、より正確で信頼性の高い結果が得られる。
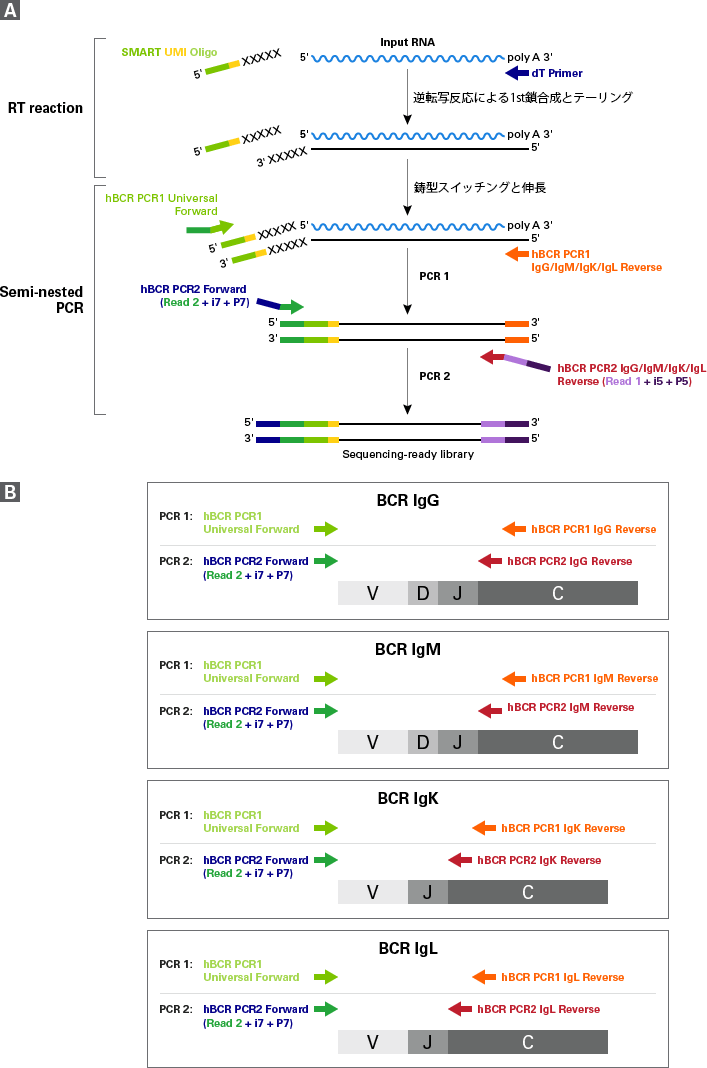
Panel B BCR IgGの可変領域のcDNA増幅のための2段階PCR
Panel A. SMARTScribe Reverse Transcriptase による1st strand cDNA合成を行い、mRNAの5'末端に達するとテンプレートに依存しないヌクレオチドが付加される。
SMART UMI Oligoは、この付加されたヌクレオチドにアニーリングし、逆転写反応によってヌクレオチドの追加配列をfirst stranded cDNAに組み込むためのテンプレートとして機能する。(これがtemplate switching ステップに相当する)。次に、1 st strand cDNAを用いて2段階の遺伝子特異的PCR増幅を行う。(Panel Bの詳細を参照)。nested PCRの2 nd PCRの産物は、大部分のリードがB細胞受容体転写物にマッピングされる。
SMART UMI Oligoは、この付加されたヌクレオチドにアニーリングし、逆転写反応によってヌクレオチドの追加配列をfirst stranded cDNAに組み込むためのテンプレートとして機能する。(これがtemplate switching ステップに相当する)。次に、1 st strand cDNAを用いて2段階の遺伝子特異的PCR増幅を行う。(Panel Bの詳細を参照)。nested PCRの2 nd PCRの産物は、大部分のリードがB細胞受容体転写物にマッピングされる。
Panel B. 1st PCRは1st strand cDNAをテンプレートにして、イルミナの Read 2 Primer配列と相補的なフォワードプライマー(PCR1 Universal Forward primer)とBCRの H鎖またはL鎖の定常領域(非可変領域)に相補的なリバースプライマー(PCR1 IgG/IgM/IgK/IgL Reverse primer)で行う。
Read 2 Primer配列と定常領域から増幅することにより、1st PCRではBCRのH鎖またはL鎖のcDNAの可変領域全体と大部分の定常領域を特異的に増幅する。2nd PCRは 1st PCRの産物をテンプレートにして、semi-nested プライマー(PCR2 IgG/IgM/IgK/IgL Reverse primer)を使用してBCRのH鎖またはL鎖のcDNAの可変領域全体と定常領域の部分を増幅する。
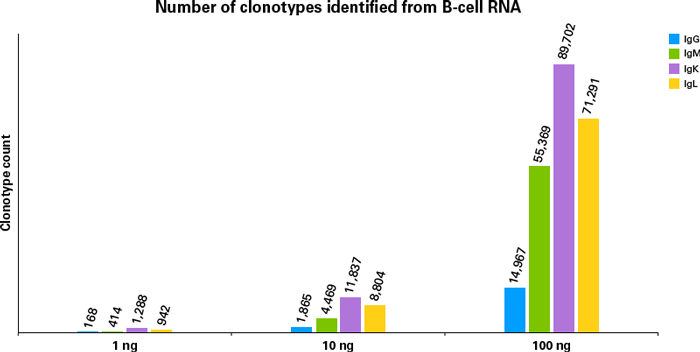
図2.1~100 ngのB細胞RNAから同定されたクロノタイプカウント
1 ng、10 ng、および100 ngのヒトCD19+B細胞由来total RNAから本製品を用いてBCRプロファイリングIgG、IgM、IgK、およびIgLライブラリーを作製した。1 ng、10 ng、100 ngのRNAインプットのライブラリーに対して、各々100,000リード、400,000リード、5,000,000リードにダウンサンプリングして解析した。インプットRNA量が増えるとクロノタイプカウントが増加し、RNA量が少なく複雑性の高いサンプルでも安定したデータが取得された。
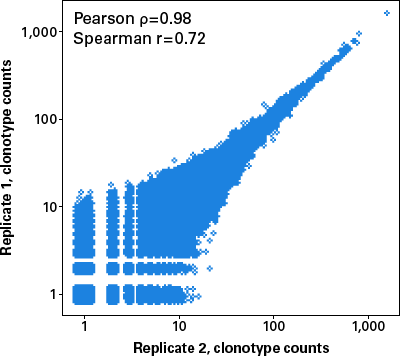
図3.1 μgのPBMC RNAから作製されたライブラリー間の再現性を示すスキャッタープロット
健常人ドナーからのPBMC由来のRNA 1μgからIgG、IgM、IgK、およびIgLのライブラリーを2連で作製した。ライブラリーは13Mリードにダウンサンプリングして解析された。プロットには、複製した4つのライブラリー(IgG、IgM、IgK、およびIgL)がすべて含まれている。1 μgのPBMC RNAを用いて行った場合、ピアソン相関係数は0.98、スピアマン相関係数は0.72になり高い再現性を示した。
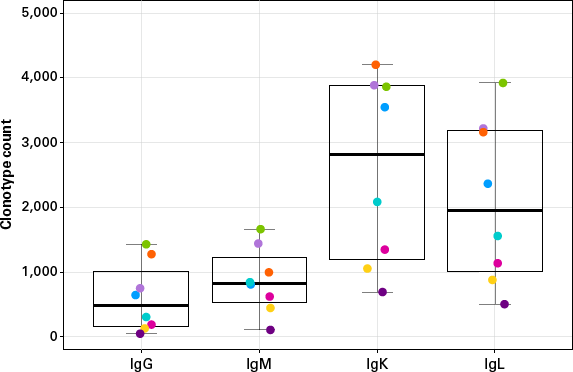
図4.さまざまなドナーから取得したPBMC RNAのクロノタイプカウント
8人のドナー(異なる色で表示)からのPBMC RNAの各10 ngからSMARTer Human BCR IgG IgM H/K/L Profiling Kitを使用してIgG、IgM、IgK、およびIgLクロノタイプを決定した。解析はすべて100,000リードにノーマライズされた。クロノタイプカウントはサンプルによって異なるが、本キットを使用すれば様々なクロノタイプが検出された。
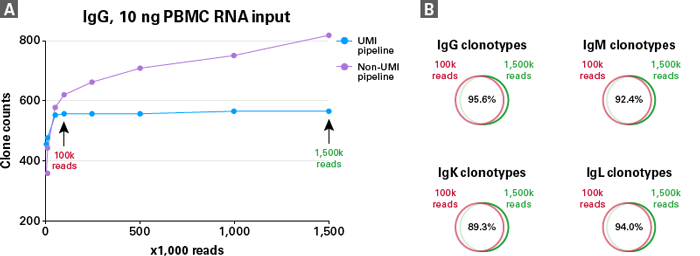
図5.10 ng PBMC RNAライブラリーのシーケンス飽和の評価
SMARTer Human BCR IgG IgM H/K/L Profiling Kitを使用して単一ドナーからのPBMC RNA10 ngからBCR Profilingライブラリーを調製した。
Read 2 Primer配列と定常領域から増幅することにより、1st PCRではBCRのH鎖またはL鎖のcDNAの可変領域全体と大部分の定常領域を特異的に増幅する。2nd PCRは 1st PCRの産物をテンプレートにして、semi-nested プライマー(PCR2 IgG/IgM/IgK/IgL Reverse primer)を使用してBCRのH鎖またはL鎖のcDNAの可変領域全体と定常領域の部分を増幅する。




Panel A. 異なるシーケンスdepthでのIgGクロノタイプカウントを、UMIベースのエラー補正あり(青線)となし(紫線)で示した。 IgGおよびその他の鎖(IgM、IgK、およびIgLは未掲載)をライブラリーごとに150万リードで配列決定し、100万、500K、200K、100K、50K、10K、および5Kリードにダウンサンプリングし、さまざまなシーケンスdepthで解析を行った。UMIを用いることで、正確性の高いクロノタイプを検出した。
Panel B. 100K(低depth)および1,500K(高depth)のライブラリー間で重複したクロノタイプを示した。
内容
- Package 1:
- Control Total RNA (1 μg/μl)
SMART UMI Oligo - Package 2:
- BCR Enhancer
First-Strand Buffer
dT Buffer
SMARTScribe Reverse Transcriptase (100 U/μl)
RNase Inhibitor (40 U/μl)
Elution Buffer (10 mM)
Nuclease-Free Water
PrimeSTAR GXL SP DNA Polymerase (1.25 U/μl)
5X PrimeSTAR GXL Buffer
dNTP Mixture (2.5 mM each)
hBCR PCR1 Universal Forward
hBCR PCR1 IgG Reverse
hBCR PCR1 IgM Reverse
hBCR PCR1 IgK Reverse
hBCR PCR1 IgL Reverse
- Package 3:
- 12回用(製品コード 634466)
hBCR PCR2 IgG Reverse 1
hBCR PCR2 IgM Reverse 1
hBCR PCR2 IgK Reverse 1
hBCR PCR2 IgL Reverse 1
hBCR PCR2 Forward 1-12
48回用(製品コード 634467)
hBCR PCR2 IgG Reverse 1-4
hBCR PCR2 IgM Reverse 1-4
hBCR PCR2 IgK Reverse 1-4
hBCR PCR2 IgL Reverse 1-4
hBCR PCR2 Forward 1-12
保存
Package 1:-70℃
Package 2,package 3:-20℃
BCR Enhancer, Nuclease-Free Water:-20℃保存。一度融解した後は4℃保存
Elution Buffer: -20℃保存。一度融解した後は室温保存。
その他:-20℃
Package 2,package 3:-20℃
BCR Enhancer, Nuclease-Free Water:-20℃保存。一度融解した後は4℃保存
Elution Buffer: -20℃保存。一度融解した後は室温保存。
その他:-20℃
本製品以外に必要な試薬、機器(主なもの)
下記の製品は本キット中に含まれません。下記の製品はユーザーマニュアルに記載のProtocolで使用できることが確認されています。他製品では代替はできないため、ご注意ください。
PCR増幅およびバリデーション
SPRI Bead精製
- シングルチャンネルピペット: 10 μl、20 μlおよび200 μl
- 8連チャンネルピペット: 20 μl、200 μl
- フィルターピペットチップ: 10 μl、20 μlおよび200 μl
- 微量遠心機 (1.5 ml チューブ用、0.2 mlチューブまたはストリップ用)
PCR増幅およびバリデーション
- 1st strand cDNA合成用のPCRサーマルサイクラー
- PCR amplification用のPCRサーマルサイクラー
- High Sensitivity NGS Fragment Analysis Kit (Advanced Analytical, Cat. No. DNF-474)
- Agilent 2100 Bioanalyzer: DNA 1000 Kit (Agilent, Cat. No. 5067-1504)
- Nuclease-free thin-wall PCR tube(0.2 ml;USA Scientific社 Code 1402-4700)
- Nuclease-free-low adhesion1.5 ml tubes (USA Scientific社 Code 1415-2600) またはLoBond tubes (Eppendorf, No.022431021)
SPRI Bead精製
- Agencourt AMPure XP PCR Purification Kit(5 ml; Beckman Coulter社; 5 ml, Code. A63880、60 ml, Code. A63881)
- 0.2 mlチューブ用マグネットスタンド
- 100% Ethanol
この製品を見た人は、
こんな製品も見ています
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。