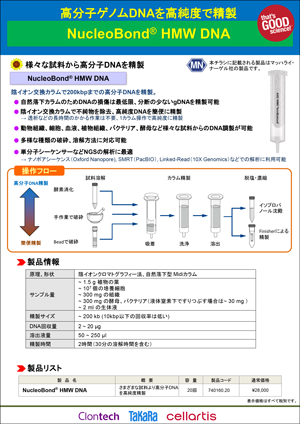
製品説明
NucleoBond HMW DNAは、グラム陽性菌、グラム陰性菌、酵母、植物、昆虫、動物組織、培養細胞、血液など、様々なサンプルから高分子ゲノムDNAを調製できるキットである。陰イオン交換によって不純物が除去されるため、サンプルの種類や溶解方法に関わらず、高純度なDNAを得ることができる。また自然落下カラムを用いているため、DNAの箭断が最小限に抑えられ、高分子のDNAを抽出することができる。本製品はMidiスケールのカラムを用いており、1回の精製に1.5 gまでの植物サンプル、300 mgまでの動物組織などのサンプルが使用可能である。本製品で精製したDNAは、SMRT sequencing(Pacific Biosciences社)やNanopore Sequencing(Oxford Nanopore Technologies社)などのロングリードシーケンスに最適である。また、Linked-Read sequencing(10x Genomics社)などのロングレンジのショートリードシーケンスにも適している。
| 原理 | 陰イオン交換クロマトグラフィー | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 形状 | 自然落下型カラム | ||||||||||||
| サンプル量 |
| ||||||||||||
| DNAサイズ | 最大200 kb(10 kb以下のDNAの回収率は低い) | ||||||||||||
| 回収量 | ~100 μg(サンプルの量とタイプに依存) | ||||||||||||
| 溶出液量 | 50~250 μl | ||||||||||||
| 精製時間 | 2時間(サンプル溶解時間30分を含む) |
内容
・Lysis Buffer H1
・Binding Buffer H2
・Wash Buffer H3
・Wash Buffer H4
・Elution Buffer H5
・Resuspension Buffer HE*
・Liquid Proteinase K
・Liquid RNase A
・NucleoBond HMW Columns + Column Filter
・Plastic Washer
* 組成:5 mM Tris-HCl, pH8.5
・Binding Buffer H2
・Wash Buffer H3
・Wash Buffer H4
・Elution Buffer H5
・Resuspension Buffer HE*
・Liquid Proteinase K
・Liquid RNase A
・NucleoBond HMW Columns + Column Filter
・Plastic Washer
* 組成:5 mM Tris-HCl, pH8.5
保存
室温
本製品以外に必要な試薬、機器(主なもの)
・イソプロパノール
・70%エタノール
・50 ml遠心チューブ
・インキュベーター(50℃設定が可能なもの)
・高速遠心機(50 mlチューブ用)
・サンプル破砕用器具、または酵素(オプション)
細胞溶解酵素(lyticase, zymolyase, lysozyme)
液体窒素、乳鉢、乳棒
MN Bead Tubes Type A(製品コード 740786.50)など
・70%エタノール
・50 ml遠心チューブ
・インキュベーター(50℃設定が可能なもの)
・高速遠心機(50 mlチューブ用)
・サンプル破砕用器具、または酵素(オプション)
細胞溶解酵素(lyticase, zymolyase, lysozyme)
液体窒素、乳鉢、乳棒
MN Bead Tubes Type A(製品コード 740786.50)など
操作手順
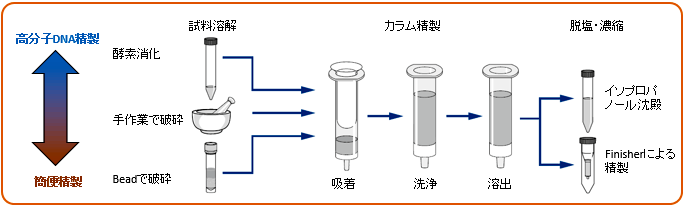
サンプルの種類に応じて適切な破砕溶解方法で調製したライセートをNucleoBond HMW Columnにロードする。カラムにセットされた特殊なろ過フィルターによってライセートが清澄化され、同時にDNAがカラムに吸着される。ろ過フィルターの除去に続く洗浄ステップで、DNAのせん断化を最小限に抑えながら、阻害物質や夾雑物質が除去される。カラムから溶出バッファーでDNAを溶出し、イソプロパノール沈殿によって脱塩濃縮を行いDNAを回収する。イソプロパノール沈殿の代わりにNucleoSnap Finisher(製品コード 740434.10, 740434.50)またはNucleoSpin Finisher(製品コード 740439.10, 740439.50)を用いるとDNAの脱塩、濃縮を迅速に行うこともできる。
サンプルの溶解方法
サンプルの溶解の前処理は、サンプルの種類や目的に応じて液体窒素中での破砕、酵素処理などサンプルに適した方法を選択する。
せん断の少ない長鎖DNAを調製するには酵素処理と界面活性剤による溶解方法が適している。このDNAへの影響の少ない溶解方法を使用した場合、平均150 kbまでのDNAの精製が期待できる。本製品にはタンパク質の溶解とヌクレアーゼの不活化に働くProteinase Kが含まれており、多くの動物組織、細胞、生体液からのDNA抽出に有効である。
バクテリアや酵母からのDNA精製には、それぞれ適した細胞壁溶解酵素を使用することが望ましい。これらの酵素は本製品には含まれないので、別途用意する必要がある。
植物組織に対しては有効な溶解酵素がないのであれば、乳棒と乳鉢によるすりつぶしが有効である。
多検体からの調製で迅速性を優先する場合はMN Bead Tubeが使用できる。ただしこの場合、平均DNA鎖長は50 kb程度に減少する可能性がある。また、超音波破砕やフレンチプレスも同様に平均DNA鎖長を減少させる。
いずれの方法にせよ、十分に試料を溶解させることが重要である。多くの試料が溶解されないままカラムへロードすると、目詰まりを引き起こす可能性がある。
せん断の少ない長鎖DNAを調製するには酵素処理と界面活性剤による溶解方法が適している。このDNAへの影響の少ない溶解方法を使用した場合、平均150 kbまでのDNAの精製が期待できる。本製品にはタンパク質の溶解とヌクレアーゼの不活化に働くProteinase Kが含まれており、多くの動物組織、細胞、生体液からのDNA抽出に有効である。
バクテリアや酵母からのDNA精製には、それぞれ適した細胞壁溶解酵素を使用することが望ましい。これらの酵素は本製品には含まれないので、別途用意する必要がある。
植物組織に対しては有効な溶解酵素がないのであれば、乳棒と乳鉢によるすりつぶしが有効である。
多検体からの調製で迅速性を優先する場合はMN Bead Tubeが使用できる。ただしこの場合、平均DNA鎖長は50 kb程度に減少する可能性がある。また、超音波破砕やフレンチプレスも同様に平均DNA鎖長を減少させる。
いずれの方法にせよ、十分に試料を溶解させることが重要である。多くの試料が溶解されないままカラムへロードすると、目詰まりを引き起こす可能性がある。
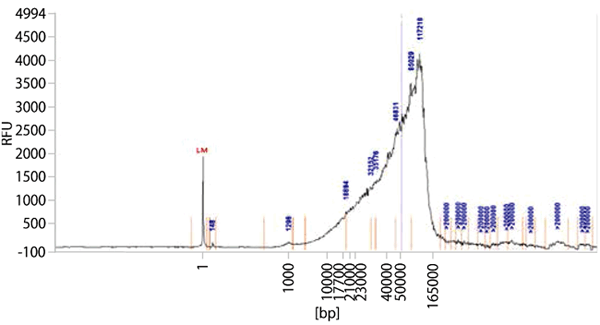
図1. 高分子DNAの精製
NucleoBond HMW DNAを用いてアブラナ属(Brassica sp.)の植物からDNAを精製した。サンプルは機械的な方法で破砕し溶解させた。精製したDNAをFemto Pulse Systemで解析したところ、117,218 bpをピークとして200,000 bpまでのフラグメントが検出され、NucleoBond HMW DNAによって高分子DNAが抽出、精製できることが示された。
表1. 精製DNAのロングリードシーケンスへの適用
NucleoBond HMW DNAを用いてアブラナ属(Brassica sp.)の植物からDNAを精製した。サンプルは機械的な方法で破砕し溶解させた。精製したDNAをFemto Pulse Systemで解析したところ、117,218 bpをピークとして200,000 bpまでのフラグメントが検出され、NucleoBond HMW DNAによって高分子DNAが抽出、精製できることが示された。
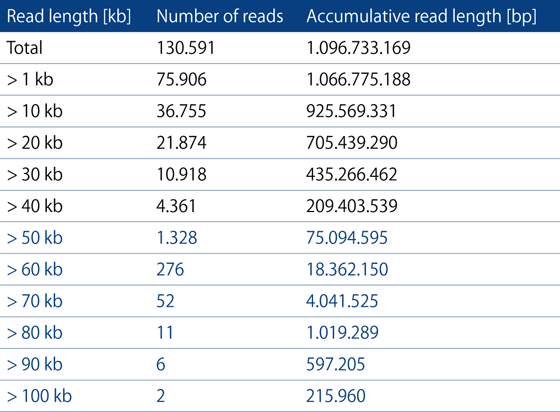
アブラナ属(Brassica sp.)の植物を機械的に破砕し、NucleoBond HMW DNAを用いてDNAを精製し、MinION device(Oxford Nanopore社)によるシーケンスを行った。その結果、1,600以上のリードで50 kb以上の配列が得られ、中には100 kb以上読まれたリードも含まれていた。NucleoBond HMW DNAによってロングリードシーケンスに適した質の高いDNAが精製できることが示された。
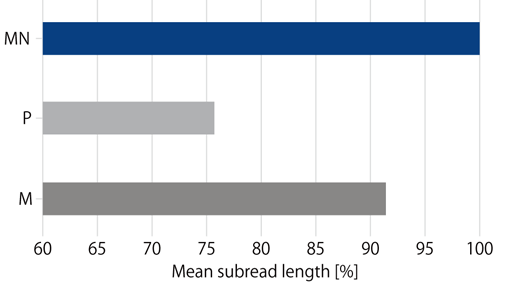
図2. 長鎖配列解析についての他社製品との比較
NucleoBond HMW DNA(MN)、または他社製品(P、M)を用いてHeLa細胞からDNAを精製し、PacBio RS IIシステム(Pacific Biosciences社)によるSingle Molecule Real Time (SMRT) シーケンス解析を行ってサブリード長を比較した。NucleoBond HMW DNAにより精製されたDNAからは他社製品と比較して優れた平均サブリード長が得られており、NucleoBond HMW DNAにより優れたリード長とシーケンス結果が得られるDNAが精製できることが示された。
NucleoBond HMW DNA(MN)、または他社製品(P、M)を用いてHeLa細胞からDNAを精製し、PacBio RS IIシステム(Pacific Biosciences社)によるSingle Molecule Real Time (SMRT) シーケンス解析を行ってサブリード長を比較した。NucleoBond HMW DNAにより精製されたDNAからは他社製品と比較して優れた平均サブリード長が得られており、NucleoBond HMW DNAにより優れたリード長とシーケンス結果が得られるDNAが精製できることが示された。
表2. 様々なサンプルからのDNA精製結果
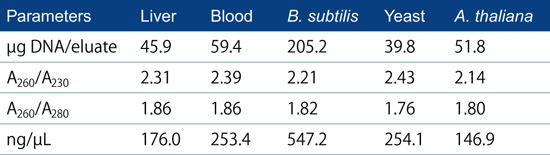
次世代DNAシーケンスで良好な結果を得るには、ゲノムDNAの鎖長、完全性に加えて純度や収率も重要で、一般的な長鎖DNA抽出法でしばしば問題となる点である。
表2.にNucleoBond HMW DNAを用いて多様なサンプルからDNAを抽出した結果を示す。全てのサンプルからシーケンスに十分な量のDNAが得られており、DNA純度を示す吸光度比も、理想的な値に近いA260/A280>1.75、A260/A230>2.00であった。
この製品を見た人は、
こんな製品も見ています
-

NucleoBond® 消耗品(シングルプレップ用)
マッハライ・ナーゲル社
-

NucleoBond® AXG Column
500 μgまでのゲノムDNAが精製できる陰イオン交換カラム
-

NucleoSpin® Tissue
動物組織、細胞、バクテリア、酵母などから効率よくゲノムDNAを精製
-
NucleoMag® HMW DNA
高分子DNAを磁性ビーズで簡便に精製
-

NucleoSnap® Finisher Midi/Maxi
吸引法による陰イオン交換カラム精製後のプラスミドDNAの濃縮・脱塩
-

NucleoSpin® Finisher Midi
遠心法による陰イオン交換カラム精製後のプラスミドDNAの濃縮・脱塩
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。