- シーケンス不要。正しくノックインされたかを迅速に判断
- バルク細胞でもシングルクローン化細胞でも使用可能
- 簡単操作で4時間でアッセイが完了
- 蛍光プレートリーダーまたはリアルタイムPCR装置で測定
- SNPs解析にも最適。対立遺伝子(SNPとWTなど)を同時検出
製品説明
本製品は、ノックインのインサートの長さ(一塩基置換からより長い挿入まで)やゲノム編集を行ったゲノム領域の配列に関係なく、相同組換え(HR)により生じたゲノム編集部位を確実に検出できるシンプルな蛍光ベースの方法を採用している。
シンプルで迅速な本キットのワークフローは、ゲノム標的部位のPCR増幅と、その後の緑色および赤色蛍光の読み取りによる酵素アッセイで構成される。酵素アッセイによる蛍光シグナルの検出は蛍光プレートリーダーまたはリアルタイムPCR装置で行うことができ、他の特別な機器を用意する必要はない。全体のワークフローは約4時間で完了し、蛍光シグナルの検出がゲノム標的部位での目的の配列の存在と相関しているため、確実に変異が検出できる。
より長い配列のノックインの場合は、アッセイにより、組み込まれた配列の5'および3'末端の両方でシームレスな挿入の同時検出が可能である。
一塩基多型(SNP)を導入するアプリケーションでは、このアッセイを使用して、2つの異なる対立遺伝子(例:SNPおよびWTの対立遺伝子)をそれぞれ1コピーずつ含むヘテロ接合クローンを明確に識別できる。
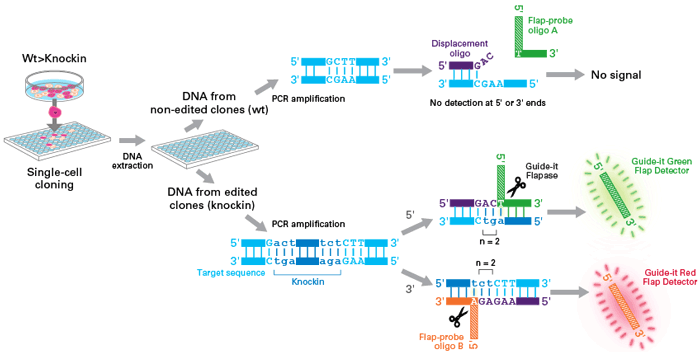
ゲノム編集を行った後、バルク細胞集団や、FACSまたは限界希釈法によって分離されたクローン細胞株は、野生型、インデル、または挿入など、様々なパターンが見られる。
クローン細胞からDNA抽出を行い標的部位のPCR増幅後、PCR産物を2種類の異なるオリゴ(displacement oligo;紫およびflap probe:インサートの5'末端とハイブリダイズするもの(flap-probe oligo A;緑)、または3'末端とハイブリダイズするもの(flap-probe oligo B:オレンジ))とアニーリングする。
相同組換えが成功して目的配列の挿入が成功した場合、両方の末端で、プローブが完全なハイブリダイゼーションするので、Guide-it Flapase(はさみで示す)により、それぞれのflap probeが切断され、緑色と赤色の両方の蛍光シグナルを生成する。1種類の蛍光シグナル(赤または緑)のみ検出される場合は、それぞれ5'末端または3'末端で挿入配列が欠失されたことを示す。蛍光が検出されない場合は、野生型配列または標的部位でindelが生じたことを示す。
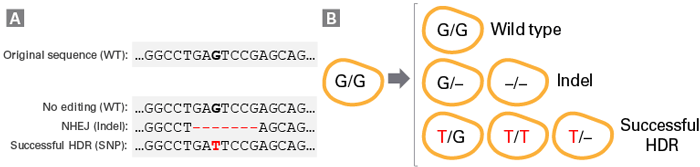
ゲノム編集技術を用いた一塩基置換(G>T)の例を示す。
Panel A.ゲノム標的部位での結果:標的部位において切断が発生しない場合、または切断発生後に非相同末端結合(NHEJ)ベースの正確な修復が行われた場合、標的部位に変化はなく、結果は野生型(WT)シーケンスになる。
標的配列で切断が発生した後に非相同末端結合(NHEJ)ベースの不正確な修復が続く場合、標的部位に挿入または欠失(Indel)が生じ、結果はノックアウト(KO:非常に可能性の高い結果)を引き起こす可能性がある。
切断の後に正確なHDR(相同組換え修復)が続くと、SNPが標的部位に導入される。
Panel B.二倍体細胞における複合対立遺伝子の結果:二倍体細胞でゲノム編集を行うと、各対立遺伝子の結果が異なり、複数の組み合わせが生成される。
ゲノム編集がされなかった場合、細胞はホモ接合性を維持することがでる(Wild type;上)。
NHEJベースの不正確な修復が続いた場合、片方または両方の対立遺伝子が変更される(Indel;中央)。
HDRが成功した場合、片方または両方の対立遺伝子で目的のSNPが挿入された細胞を得ることができる(Successful HDR;下)。
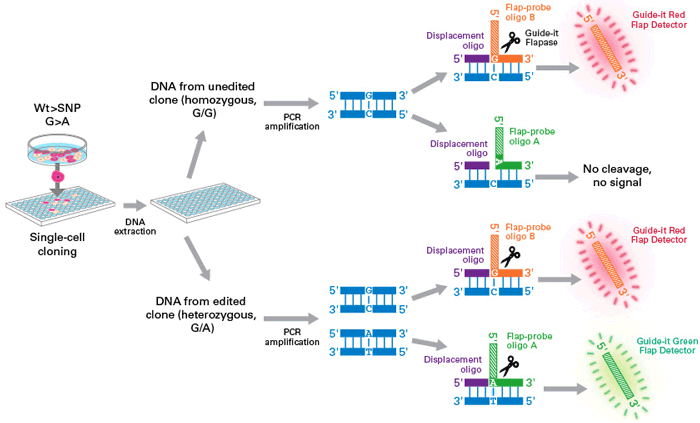
このワークフロー例では、G>A置換の検出例を示す。
Gが野生型でAが置換される塩基である。ゲノム編集後、クローン化したシングルセルからは、目的のゲノム標的部位でいくつかの異なる遺伝子型の結果が得られる。標的部位のPCR増幅後、PCR産物はdisplacement oligo(紫色)とflap-probe oligo A(緑色;SNP対立遺伝子A)またはflap-probe oligo B(オレンジ;WT対立遺伝子G)のいずれかとの組み合わせにより同時にアニーリングされる。PCR産物へのオリゴのアニーリングの後、Guide-it Flapase(はさみで示す)は完全な塩基対を認識し、flap-probe oligoの5'部分(緑色またはオレンジ色)を切断する。切断されたflapは、対応するGuide-it Flap Detectorによって検出され、それぞれ緑色または赤色の蛍光シグナルを発する。上記の例では、対象部位でホモ接合性WT(G/G)であるクローン細胞株の分析では赤色シグナルのみが得られ、編集されたWTとSNPの対立遺伝子(G/A)を含むヘテロ接合性クローンの分析では赤と緑の両方のシグナルが得られる。
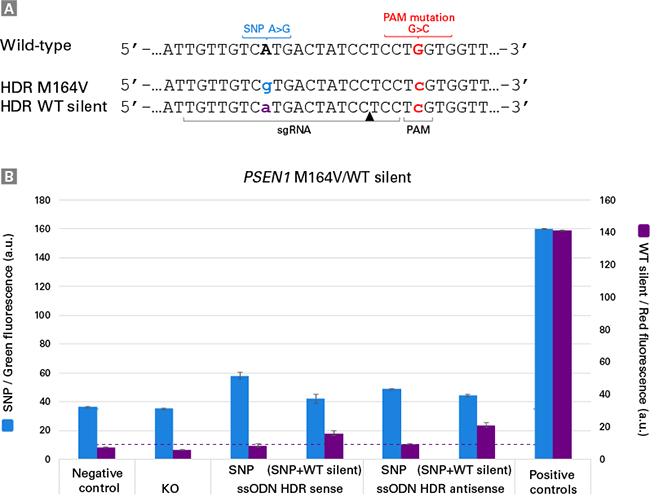
Guide-it Knockin Screening KitのSNP検出機能を実証するために、CRISPR/Cas9システムを使用して、早期発症アルツハイマー病に関連するA>G置換(M164V)をコードするPSEN1遺伝子のバリアントのヘテロ接合iPS細胞株を作製した。
Panel A. PSEN1遺伝子座で正確な編集を行うために使用されるsilent PAM変異を含むHDRテンプレート:HDRが成功した場合、PSEN1遺伝子座は、WTまたはSNP対立遺伝子のいずれかを、隣接するPAM配列のsilent変異と組み合わせて符号化する。
Panel B. ゲノム編集後のバルクiPS細胞集団での塩基置換の検出:センスまたはアンチセンスHDRテンプレートのいずれかを使用する方法で、Guide-it Knockin Screening Kitを使用して、各ケースで編集の成功を検出できるかどうかを確認した。
Displacement oligo または flap-probe oligosは、対応するHDRテンプレートによって符号化されたsilent(G>C)PAM突然変異を持つWTまたはSNP対立遺伝子を検出し、それぞれ赤色および緑色蛍光シグナルを生成するように設計された。
細胞にCas9タンパク質のみ(negative control)、Cas9-sgRNA複合体RNP(KO)、センスまたはアンチセンスWT/SNP HDRテンプレートとRNPを混合したものをそれぞれ別々にエレクトロポレーションを行った。WTまたはSNP配列をコードする合成オリゴをsilent PAM突然変異と組み合わせて、positive controlとして並行してアッセイを行った。HDRテンプレートを用いたエレクトロポレーションの結果では、大量のバルク集団で成功した相同組換えが検出できた。Negative controlの緑および赤の蛍光シグナルは検出されず、未編集のWT配列の存在下では偽陽性の蛍光シグナルを生じなかったため、flap-probe oligoの特異性が確認できた。
内容
Guide-it Knockin Positive Control Mix
Guide-it Knockin Negative Control Mix
Terra PCR Direct Polymerase Mix
2X Terra PCR Direct Buffer (with Mg2+, dNTP)
Dilution Buffer
RNase-free Water
Annealing Buffer
Flapase Buffer
Guide-it Flapase
Guide-it Green Flap Detector (40X)
Guide-it Red Flap Detector (40X)
保存
| MightyPrep Reagent for DNA | : | 4℃ |
| その他 | : | -20℃ |
本製品以外に必要な試薬、機器(主なもの)
- Displacement Oligo および Flap-probe Oligo
Flap-probe Oligoは3'末端に Hexanediol(/3C6/)修飾が必要です。
各オリゴの設計はオンラインツールをご利用ください。 - ゲノムDNAからターゲット配列を増幅するためのPCRプライマー
- Dulbecco's Phosphate Buffered Saline without Ca2+ & Mg2+ (Sigma-Aldrich社, Cat.No. 8537)など
- ホットプレート
- サーマルサイクラー
- サーマルサイクラー用96 wellプレート
- 96ウェルプレート用cap stripまたはフィルム
- 96ウェルプレート用低速遠心機
- 低吸着チューブ (Eppendorf社, Cat. No. 022431005など)
- 96ウェル(黒)プレート (Corning社, Cat. No. 3631など)
- NucleoSpin Tissue(製品コード 740952.250)
- 蛍光プレートリーダーまたはリアルタイムPCR装置
(緑色蛍光(励起485 nm/発光535 nm)および赤色蛍光(励起590 nm/発光620 nm)の検出ができる装置)
用途
- ゲノム編集されたバルク細胞集団における正確なノックインまたはSNPsの迅速な検出
- ヘテロ接合クローン内のゲノム編集済みおよび未編集の対立遺伝子の同時検出
- シングルセルクローニング前のバルク細胞集団での編集効率の分析
オンラインツール
※このオンラインツールは、ノックイン(長い配列の挿入)ではなく、一塩基置換のみを検出するオリゴDNAを設計します。
Oligo design tool for Guide-it SNP screening assaysはこちら
この製品を見た人は、
こんな製品も見ています
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。