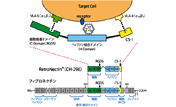
shRNA発現用レトロウイルスベクター
製品説明
pSINsiベクターシリーズは、自己不活型レトロウイルスベクタープラスミドを基本骨格とし、RNA polymerase III (pol III) 系のプロモーターによりヘアピン型RNAを発現させるベクターである。pol III系のプロモーター(human H1、human U6、またはmouse U6)の下流のクローニングサイトにヘアピン型RNAを発現させるための合成オリゴDNA配列を挿入することによって、siRNA発現レトロウイルスベクタープラスミドが得られる。マーカー遺伝子としてはネオマイシン耐性遺伝子を搭載している。siRNA発現レトロウイルスベクタープラスミドから、Retrovirus Packaging Kit Eco/Ampho(製品コード 6160、6161)などを用いて、組換えレトロウイルスを調製することができる。
保存
-20℃
制限酵素地図
pSINsiベクターシリーズではプロモーターの異なる3種のプラスミドベクターを用意している。
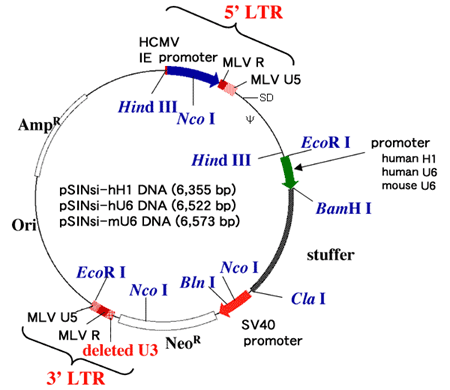
pSINsiベクターの制限酵素地図
pSINsi-hH1 DNA: human H1 promoter (Accession S68670)を搭載。
pSINsi-hU6 DNA: human U6 promoter (Accession X07425)を搭載。
pSINsi-mU6 DNA: mouse U6 promoter (Accession X06980)を搭載。
pSINsi-hU6 DNA: human U6 promoter (Accession X07425)を搭載。
pSINsi-mU6 DNA: mouse U6 promoter (Accession X06980)を搭載。

pSINsiベクターの制限酵素地図
各ベクターのDNAシーケンスデータ
ZIP形式で圧縮しておりますが、StuffIt Expander等でも解凍できます。
pSINsi-hH1 DNAのシーケンスデータ(テキストファイル)
pSINsi-hU6 DNAのシーケンスデータ(テキストファイル)
pSINsi-mU6 DNAのシーケンスデータ(テキストファイル)
pSINsi-hU6 DNAのシーケンスデータ(テキストファイル)
pSINsi-mU6 DNAのシーケンスデータ(テキストファイル)
【注意】シーケンス上では、1259の位置(プロモーター上流、Hind IIIとEcoR Iの間)にCla Iサイトがありますが、通常のdam+大腸菌で調製したプラスミドは、dam methylaseによる影響を受けるため、1259位置のCla Iサイトは切断されません。
原理
pSINsiベクターシリーズのレトロウイルスベクタープラスミドは3'LTR U3領域のプロモーター活性を欠損させている。そのため、逆転写後、染色体に組み込まれたプロウイルスの状態では5'LTRのプロモーター活性が消失し、プロウイルス由来のmRNAは転写されなくなっており、pol III系のプロモーターからのヘアピン型RNAとSV40プロモーターからのネオマイシン耐性遺伝子のみが転写される。
発現したヘアピン型RNAは細胞内のDicerによる分解を受けてsiRNAとなる(下図参照)。
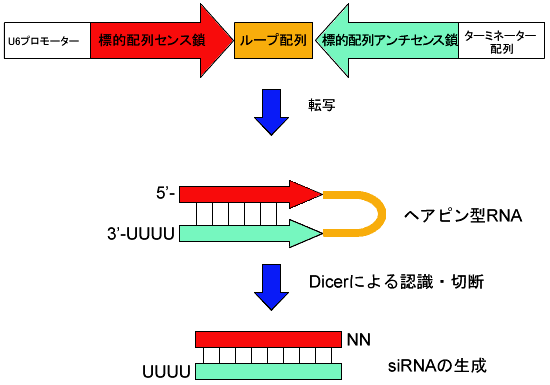
ヘアピン型RNA発現ベクター
発現したヘアピン型RNAは細胞内のDicerによる分解を受けてsiRNAとなる(下図参照)。

ヘアピン型RNA発現ベクター
ヘアピン型RNAを発現させるためのDNA合成
ヘアピン型RNAを発現させるためには、[Ligation用の制限酵素サイト+標的配列(センス)+ループ配列+標的配列(アンチセンス)+ターミネーター配列+Ligation用の制限酵素サイト]の順でデザインした合成DNAをプロモーターの下流に挿入する。pSINsiベクターのクローニングサイトは、上流側はBamH I、下流側はCla Iである。
下図に示すような合成オリゴDNAを作製する(Top strandとBottom strandの2本; N部分が標的配列)。U6プロモーターの転写開始点はプリン塩基(GまたはA)が好ましいため、標的配列がGまたはAで始まらない場合は標的配列の前にGまたはAを挿入する。
ここではループ配列の例としてCTGTGAAGCCACAGATGGG(Boden et al. 参考文献 1)を挙げているが、GTGTGCTGTCC(Miyagishi et al. 参考文献 2)も有用であることが確認されている。また、これ以外のヘアピンループとして、Lee et al.(参考文献 3)、Paddison et al. (参考文献 4)、Paul et al.(参考文献 5)、Sui et al. (参考文献 6)らによってそれぞれ異なった配列が報告されている。
ターミネーター配列には、TTTTTT配列を用いる(Tが4つ続くとpol III系プロモーターによる転写が止まる)。siRNA配列と用いるヘアピンループ配列の組み合わせによってはTが4つ以上続く可能性があるため、合成DNAをデザインした後には必ず、Top strandにTが4つ以上続いていないことを確認することが必要である。

下図に示すような合成オリゴDNAを作製する(Top strandとBottom strandの2本; N部分が標的配列)。U6プロモーターの転写開始点はプリン塩基(GまたはA)が好ましいため、標的配列がGまたはAで始まらない場合は標的配列の前にGまたはAを挿入する。
ここではループ配列の例としてCTGTGAAGCCACAGATGGG(Boden et al. 参考文献 1)を挙げているが、GTGTGCTGTCC(Miyagishi et al. 参考文献 2)も有用であることが確認されている。また、これ以外のヘアピンループとして、Lee et al.(参考文献 3)、Paddison et al. (参考文献 4)、Paul et al.(参考文献 5)、Sui et al. (参考文献 6)らによってそれぞれ異なった配列が報告されている。
ターミネーター配列には、TTTTTT配列を用いる(Tが4つ続くとpol III系プロモーターによる転写が止まる)。siRNA配列と用いるヘアピンループ配列の組み合わせによってはTが4つ以上続く可能性があるため、合成DNAをデザインした後には必ず、Top strandにTが4つ以上続いていないことを確認することが必要である。

siRNA発現用組換えレトロウイルス作製の概要
関連のオンラインガイドはこちら
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。