製品説明
PrimeSTAR HS DNA Polymeraseは、タカラバイオが独自に開発した極めて高い正確性と優れた増幅効率を併せ持つDNA polymeraseである。本酵素は非常に強力な3'→5' exonuclease活性を有し、PCR増幅において抜群の校正力を示す一方、Taq DNA polymeraseに優る高い増幅効率も示す。常温下でのDNA polymerase活性および3'→5' exonuclease活性を抑えるモノクロ-ナル抗体を添加しているため、PCR反応前のミスプライミングやプライマーの消化を防ぐことができ、高い反応特異性が得られる。また、高いプライミング効率を有しているため、アニーリング時間を短時間に設定でき、反応時間を短縮することができる。さらに、高度に至適化したPrimeSTAR Bufferを用いることで、幅広いターゲットに対する高Fidelity、高感度、高特異性、高い成功率での増幅が実現した。
内容
(200反応分、50 μl反応系)
* Mg2+ 濃度は5 mM(5×)です。
| PrimeSTAR HS DNA Polymerase | 2.5 U/μl | 100 μl |
| 5×PrimeSTAR Buffer(Mg2+ plus)* | 5× | 1 ml×2 |
| dNTP Mixture | 2.5 mM each | 800 μl |
* Mg2+ 濃度は5 mM(5×)です。
保存
-20℃
形状
| 50 mM | Tris-HCl (pH8.2 at 4℃) |
| 100 mM | NaCl |
| 0.1 mM | EDTA |
| 1 mM | DTT |
| 0.1% | Tween 20 |
| 0.1% | NP-40 |
| 50% | Glycerol |
添付dNTP Mixture*
| 濃度 | 各2.5 mM |
|---|---|
| 形状 | 水溶液(ナトリウム塩) pH7~9 |
| 純度 | 各98%以上 |
* dATP、dCTP、dGTP、dTTPの等モル混合物で、希釈せずにそのままPCR反応に用いることができる。
特長
- 正確性
- アニ-リング時間
- 増幅効率
- 耐熱性
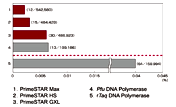
GC richなThermus thermophilus HB8 genomic DNAを鋳型として、任意に選択した8領域(増幅サイズはそれぞれ約500 bp)をPCR増幅後、ベクターにクローニングし、各配列について複数クローンをピックアップしてそのシーケンスを確認し、mutation frequencyを求めた。
その結果、PrimeSTAR HS DNA PolymeraseはTaq DNA Polymeraseと比べて10倍Fidelityが高く、A社High Fidelity酵素に比べても同等以上の正確性を示した。
この方法は、実際のPCRに最も即したFidelityの求め方であり、正確性が重要な反応に安心して本酵素を使用することができる。
各酵素のFidelity 比較 | |
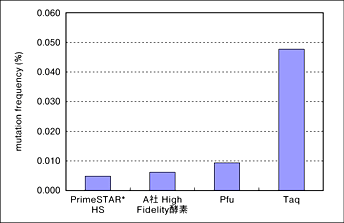 | * PrimeSTAR HSで増幅した場合、実際に解析した249,941塩基のうち、エラーはわずかに12塩基であった。 |
PrimeSTAR HS DNA Polymeraseは、非常に高いプライミング効率を有している。そのため、アニーリング時間を短時間(5秒または15秒)に設定することで特異性の高い増幅が実現する。
条件設定の詳細に関しては、下記の■PCR条件を参照してください。
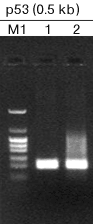 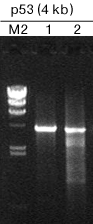 |
M1 : pHY Marker M2 : λ - Hind III digest 1 : アニーリング時間 55℃ 5秒 2 : アニーリング時間 55℃ 30秒 Template : ヒトゲノムDNA 100 ng 反応系 : 50 μl PCR 条件 : 3 step PCR、30 cycles |
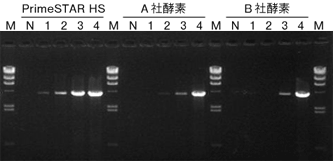
(1) ヒトDCLRE1A遺伝子2 kb、大腸菌ゲノムの4 kb領域をターゲットとして、A社およびB社High Fidelity酵素と反応性を比較した。
| 反応液組成 | :それぞれの至適反応液組成(反応系50 μl) |
| PCR 条件 | :それぞれの推奨条件(3 step PCR、30 cycles) |
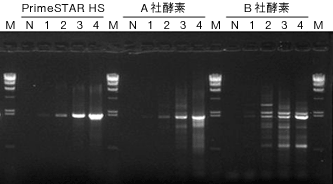 |
(ヒトDCLRE1A 遺伝子 2 kb) M : λ - Hind III digest N : No Template 1 : ヒトゲノムDNA 100 pg 2 : ヒトゲノムDNA 1 ng 3 : ヒトゲノムDNA 10 ng 4 : ヒトゲノムDNA 100 ng |
 |
(E. coli 4 kb) M : λ - Hind III digest N : No Template 1 : JM109 ゲノムDNA 1 pg 2 : JM109 ゲノムDNA 10 pg 3 : JM109 ゲノムDNA 100 pg 4 : JM109 ゲノムDNA 1 ng |
PrimeSTAR HS DNA Polymeraseは良好な反応性を示し、他社High Fidelity酵素よりも特異性が高く、1オーダー高い検出感度が得られた。
(2) ヒトゲノム、大腸菌ゲノムを鋳型として、各サイズの増幅を行った。
| 鋳型 | ヒトゲノムDNA 50 ng /50 μl 反応系 | |||||
| サーマルサイクラ- | PCR Thermal Cycler Dice | |||||
| PCR条件 | 0.5~6 kb;3 step PCR | 7.5~8.5 kb;2 step PCR | ||||
| 98℃ | 10 sec | 30 cycles | 98℃ | 10 sec | 30 cycles | |
| 60℃ | 5 sec | 68℃ | 8 min | |||
| 72℃ | 1 min/kb | |||||
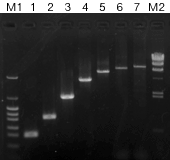 |
M1 : pHY Marker 1 : 0.5 kb 2 : 1 kb 3 : 2 kb 4 : 4 kb 5 : 6 kb 6 : 7.5 kb 7 : 8.5 kb M2 : λ - Hind III digest |
| 鋳型 | 大腸菌ゲノムDNA 100 pg /50 μl反応系 | ||
| サーマルサイクラ- | PCR Thermal Cycler Dice | ||
| PCR 条件 | 3 step PCR | ||
| 98℃ | 10 sec | 30 cycles | |
| 60℃ | 5 sec | ||
| 72℃ | 1 min/kb | ||
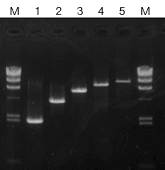 | M : λ - Hind III digest 1 : 2 kb 2 : 4 kb 3 : 6 kb 4 : 8 kb 5 : 10 kb |
(1) 3 step (98℃ 10 sec. /55℃ 15 sec. /72℃ 4 min.)でPCRを行った場合、30サイクル後の残存活性は約80%である。
(2) 2 step (98℃ 10 sec. /68℃ 4 min.)でPCRを行った場合、30サイクル後の残存活性は約85%である。
一般的なPCR反応液組成(Total 50 μl)
| 使用量 | 最終濃度 | |
|---|---|---|
| 5×PrimeSTAR Buffer (Mg2+ plus) | 10 μl | 1× |
| dNTP Mixture (2.5 mM each) | 4 μl | 200 μM each |
| Primer 1 | 10 ~15 pmol | 0.2 ~0.3 μM |
| Primer 2 | 10 ~15 pmol | 0.2 ~0.3 μM |
| Template | <200 ng | |
| PrimeSTAR HS DNA Polymerase(2.5 U/μl) | 0.5 μl | 1.25 U /50 μl |
| 滅菌精製水 | up to 50 μl |
※PCR反応液の調製は室温でも可能である。ただし、酵素などの各試薬は氷上に置いて使用してください。
PCR条件
(A) 3 step PCRの場合
(B) 2 step PCRの場合
本酵素の場合、通常は3 step PCRでの反応を推奨する。
<重要> 本酵素はプライミング効率が非常に高い酵素ですので、アニーリング時間は5 sec. もしくは15 sec. に設定して反応を行ってください。アニーリング時間が長くなると、スメアが生じる場合があります。
| 98℃ | 10 sec. | 30 cycles |
| 55℃ | 5 sec. または15 sec. | |
| 72℃ | 1 min. /kb |
(B) 2 step PCRの場合
| 98℃ | 10 sec. | 30 cycles |
| 68℃ | 1 min. /kb |
本酵素の場合、通常は3 step PCRでの反応を推奨する。
- 変性条件 ; 98℃ 5 ~10 sec. を推奨。94℃で行う場合は10 ~15 sec. に設定する。
- アニーリング温度 ; まず55℃で試す。
- アニーリング時間
Tm値(下記の式で計算)が55℃以上の場合→ 5 sec. に設定
Tm値(下記の式で計算)が55℃未満の場合→ 15 sec. に設定
※Tm 値の計算方法
Tm値(℃)=[(A、Tの数)×2]+[(G、Cの数)×4]-5
プライマーの長さが25 mer以下の場合に適用する。25 merを越える場合は、アニーリング時間を5 sec. に設定する。
<重要> 本酵素はプライミング効率が非常に高い酵素ですので、アニーリング時間は5 sec. もしくは15 sec. に設定して反応を行ってください。アニーリング時間が長くなると、スメアが生じる場合があります。
PCR産物について
PrimeSTAR HS DNA Polymeraseを用いて増幅したPCR産物のほとんどは平滑末端である。したがって、そのPCR産物をそのまま(必要に応じてリン酸化して)平滑末端のベクターにクローニングすることが可能である。平滑末端ベクターへのクローニングにはMighty Cloning Reagent Set (Blunt End)(製品コード 6027) が利用できる。
T-vectorにクローニングする場合は、PCR産物の3'末端にdAを付加する必要がある。T-vectorへのクローニングにはMighty TA-cloning Reagent Set for PrimeSTAR(製品コード 6019)が利用できる。
T-vectorにクローニングする場合は、PCR産物の3'末端にdAを付加する必要がある。T-vectorへのクローニングにはMighty TA-cloning Reagent Set for PrimeSTAR(製品コード 6019)が利用できる。
【ダイレクトシーケンスを行う場合】
本酵素は3’→5’exonuclease活性を有するため、ダイレクトシーケンシングを行う前にフェノール/クロロホルム処理などのタンパク質除去操作を行ってください。PCR検定
●λDNAを鋳型としたPCR(増幅産物 8、10、12、15 kb)において良好な増幅が見られることを確認している。
●ヒトゲノムDNAを鋳型としたPCR(増幅産物 0.5、1、2、4、6、8 kb)において良好な増幅が見られることを確認している。
●ヒトゲノムDNAを鋳型としたPCR(増幅産物 0.5、1、2、4、6、8 kb)において良好な増幅が見られることを確認している。
活性の定義
活性化サケ精子DNAを鋳型/プライマーとして用い、下記の活性測定用反応液中にて74℃において、30分間に10 nmolの全ヌクレオチドを酸不溶性沈殿物に取り込む活性を1 Uとする。
活性測定用反応液組成
| 100 mM | Tris-HCl 緩衝液 (pH8.3 at 37℃) |
| 10 mM | KCl |
| 6 mM | (NH4)2SO4 |
| 2 mM | MgCl2 |
| 0.1% | Triton X-100 |
| 0.001% | BSA |
| 各 200 μM | dATP・dGTP・dCTP |
| 100 μM | [3H]TTP |
| 0.4 mg/ml | 活性化サケ精子 DNA |
純度
●10 Uの本酵素と0.6 μgのλDNAを74℃、1時間反応させてもDNAの電気泳動パターンに変化は起こらない。
●10 Uの本酵素と0.6 μg のsupercoiled pBR322 DNAを74℃、1時間反応させてもメインバンドの電気泳動パターンに変化は起こらない。
●10 Uの本酵素と0.6 μg のsupercoiled pBR322 DNAを74℃、1時間反応させてもメインバンドの電気泳動パターンに変化は起こらない。
「タカラバイオ PCR Enzymes Guide」では、PCR酵素・関連製品とそのアプリケーションをご紹介しています。
下記よりPDFでご覧いただけます。
冊子のお届けは、こちらからご請求ください。
下記よりPDFでご覧いただけます。
冊子のお届けは、こちらからご請求ください。
Application
さまざまな長さの遺伝子をターゲットとした同一PCR条件での増幅
cDNAを鋳型としてのPCR
他社High-Fidelity酵素との品質の比較
PCR反応阻害物質(SDS、腐植酸)の存在下における他酵素との活性の比較
お客様からご提供いただいたPrimeSTAR HS DNA Polymeraseの使用例
お客様からご提供いただいたPrimeSTAR HS DNA Polymeraseの使用例-2
BIO VIEW / Clontechniques
High Fidelity PCR 酵素 PrimeSTAR®シリーズ
~正確性の検証~
制限酵素いらずのDirectional Cloning
「In-Fusion™ PCRクローニングキット」とハイフィデリティーPCR酵素「PrimeSTAR® 」との組み合わせで、極めてエラーの少ない自由自在のクローニングが実現できます。
高い増幅効率を併せ持ったHigh-Fidelity PCR酵素が新登場!!
PrimeSTAR® HS DNA Polymerase
この製品を見た人は、
こんな製品も見ています
-
Mighty Cloning Reagent Set (Blunt End)
PCR産物の平滑化・リン酸化&クローニング
-

NucleoSpin® Gel and PCR Clean-up
PCR産物の精製にもDNAのゲル抽出にも対応
-
PrimeSTAR® GXL DNA Polymerase
長鎖増幅やGCリッチターゲットに強いHigh Fidelity PCR酵素
-

Mighty TA-cloning Reagent Set for PrimeSTAR®
PrimeSTARによるPCR産物をTAクローニング
-

PrimeSTAR® Max DNA Polymerase
最高レベルの正確性&世界最速の伸長性
-
B型肝炎ウイルス表面抗原
- 注意事項
- 本ページの製品はすべて研究用として販売しております。ヒト、動物への医療、臨床診断用には使用しないようご注意ください。また、食品、化粧品、家庭用品等として使用しないでください。
- タカラバイオの承認を得ずに製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。
- タカラバイオ製品に関連するライセンス・パテントについては、ライセンスマークをクリックして内容をご確認ください。
また、他メーカーの製品に関するライセンス・パテントについては、各メーカーのウェブサイトまたはメーカー発行のカタログ等でご確認ください。 - ウェブサイトに掲載している会社名および商品名などは、各社の商号、または登録済みもしくは未登録の商標であり、これらは各所有者に帰属します。

